6. Arrumando os dados de vigilância em saúde
Uma etapa importante para a produção de informações de qualidade é o cuidado com a organização e o armazenamento do dado coletado diariamente. Arrumar um dado significa garantir que houve uma forma padronizada de conectar a estrutura (formato) de um conjunto de dados à sua semântica (significado).
Dados bem estruturados servem para:
- fornecer dados seguros para o processamento e análise de dados por softwares;
- revelar informações e facilitar a percepção de padrões.
Neste tópico vamos conhecer técnicas para arrumar nossos dados e analisá-los de forma eficiente e segura.
6.1 Organizando dados em linhas e colunas (dados retangulares)
Os objetos mais comuns que armazenam dados possuem uma estrutura de duas dimensões: linhas e colunas. Por exemplo, na matriz que calcula taxa de ataque em casos de surtos temos linhas e colunas que armazenam um mesmo tipo de dado, ou seja, apenas números, mas também poderá armazenar outros formatos possíveis, como textos. Já em um data.frame, uma coluna pode conter dados numéricos, outra dados textuais e outra datas.
Você perceberá que há uma organização que nos remete a uma forma geométrica bem definida e estruturada, como a de um retângulo. Por definição, dados organizados em linhas e colunas são chamados dados retangulares.
Já os dados não retangulares seriam aqueles armazenados em uma estrutura diferente como, por exemplo, texto de um discurso, ou até mesmo o campo de observação das fichas de notificação compulsórias.
Assim, dados retangulares são os dados no “formato arrumado” que atendem às seguintes regras:
- cada variável está em uma coluna;
- cada observação* corresponde a uma linha;
- cada valor corresponde a uma célula;
- cada tipo de unidade observacional deve compor uma tabela.
*Como sinônimo de observações você pode encontrar os termos: registros, casos, exemplos, instâncias ou amostras, dependendo da área de aplicação.
Na Vigilância em Saúde, o uso de dados no formato retangular é comum e quase sempre necessário, sendo a forma de armazenamento de todos os Sistemas de Informações em Saúde e, inclusive, de outras fontes de registro comuns na área da saúde, como prontuários, formulários do REDcap, formSUS ou mesmo google forms.
Vamos lá, enquanto profissional de vigilância você necessita buscar informações sobre o acompanhamento de pacientes na atenção primária em saúde. Para isso, irá consultar o registro dos atendimentos realizados no e-SUS AB. Na consulta ao sistema você encontrou o registro de quatro pessoas e seus atendimentos. A partir disso, você abriu uma planilha de Excel e criou uma tabela na qual inseriu o número de identificação da ficha de atendimento, a idade e o sexo biológico de cada um dos pacientes.
Na Figura 4 exibimos a representação visual da planilha criada por você e que aqui será utilizada como nosso banco de dados.
Figura 4: Banco de dados com o número de identificação da ficha de atendimento, a idade e o sexo biológico de cada um dos pacientes.
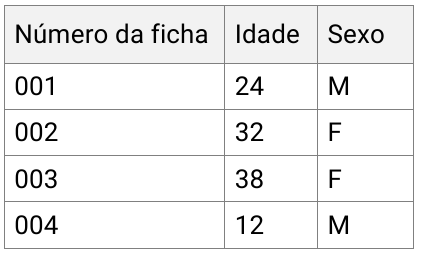
É possível perceber o formato retangular dos dados em que cada coluna se refere a diferentes tipos de dados (a primeira variável, por exemplo, é composta por um número ordenado, a segunda por um número inteiro e a terceira por texto).
Agora, observe a Figura 5: a Figura 5A tem como destaque a coluna com a variável Idade, representando a idade de um paciente em uma única coluna. Na Figura 5B, a linha em destaque se refere a um único paciente, identificado pelo número da ficha de registro, e a Figura 5C evidencia a célula em destaque com um único valor de idade de um único paciente.
Figura 5: Tabelas identificando coluna (A), linha (B) e valor (C).
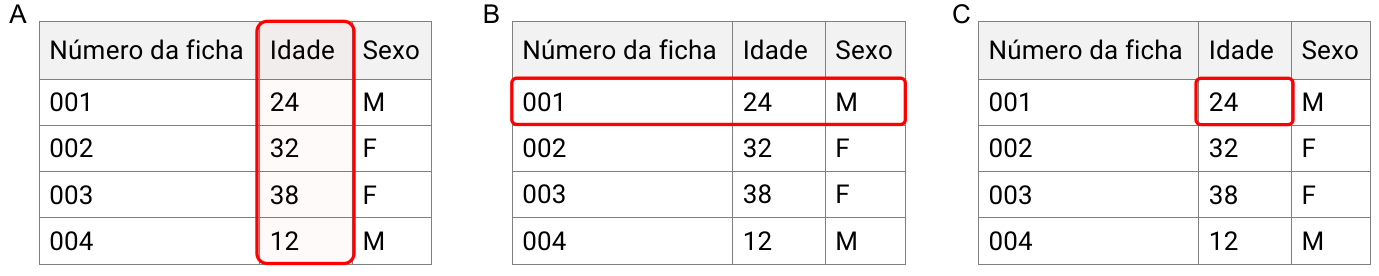
Em todos os casos apresentados perceba que os dados são organizados da forma mais corriqueira, em bancos de dados utilizados no seu dia a dia, quase sempre no formato retangular.
6.2 Modificando o formato de um banco de dados
Os dados retangulares podem apresentar formatos diferentes, sendo os mais comuns largo e longo (em inglês wide e long, respectivamente). O formato longo se refere a um visual no qual a base aparenta ter muitas linhas em relação às colunas, e o formato largo se refere às tabelas que aparentam ter mais colunas, estando assim muito “larga” em relação às suas linhas.
Na linguagem R, vários pacotes e funções são executadas
de forma mais eficiente quando os dados são organizados no formato longo
(muitas linhas em relação às colunas). Alguns deles, como o pacote
utilizado para a visualização de gráficos ggplot2, são
essenciais para uma análise exploratória de dados. Além disso, a própria
prática de explorar os dados torna-se mais fluida quando há uma
padronização uniforme nas bases de dados.
Como profissional de vigilância em saúde, você está acostumado com bases de dados que muitas vezes não estão organizadas no formato longo ou largo ou, ainda, que sigam as regras citadas acima. Vejamos o exemplo abaixo.
Você se lembra do banco de dados disponibilizado pela equipe do Setor
de Imunização do Estado de Rosas com os dados de cobertura vacinal
contra Hepatite B em crianças de até 30 dias de idade? Vamos utilizá-lo
novamente aqui! Estes dados correspondem à vacinação realizada de 2016 a
2020 nos municípios de Prímula e Antúrio, da região norte de Rosas,
exportados pela equipe junto ao módulo de Imunização do Tabnet e
alterados a partir de um arquivo com extensão do tipo .csv
{cobertura_hepatiteb_rosas_2016_2020_A.csv}. Este arquivo
está disponível no menu lateral “Arquivos” deste módulo.
Acompanhe o script abaixo e replique-o em seu
RStudio:
# Importando o banco de dados { `cobertura_hepatiteb_rosas_2016_2020_A.csv }
# para o `R`
dados <- read_csv(file = "Dados/cobertura_hepatiteb_rosas_2016_2020_A.csv")#> # A tibble: 2 × 6
#> Municipio `Ano 2016` `Ano 2017` `Ano 2018` `Ano 2019` `Ano 2020`
#> <chr> <chr> <chr> <chr> <chr> <chr>
#> 1 Primula 99.55% 90.09% 91.54% 86.75% 69.32%
#> 2 Anturio 79.97% 113.83% 131.06% 115.53% 95.16%Perceba que a equipe do Setor de Imunização mantém esta base no formato largo. A tabela produzida a partir do data.frame possui seis colunas e duas linhas, onde cada linha se refere a um município específico. Entretanto, há cinco colunas que se referem à mesma variável (Ano), colunas que representam a mesma característica: cobertura vacinal.
Para analisar as coberturas vacinais por ano, necessitaremos que
todos os dados da tabela {dados} estejam em uma única
coluna. Isso porque precisamos facilitar a organização dos dados em um
formato que se possa analisá-los temporalmente, considerando por tanto
que cada coluna faz referência a um ano. Fique tranquilo, explicaremos
de forma detalhada mais adiante estas etapas.
Dessa forma, modificaremos o formato largo
(wide) para uma tabela em formato longo
(long) com a função de pivotagem no
R.
A pivotagem é uma técnica que se refere à mudança de formato de uma base, sendo uma das principais operações de transformação de dados. Basicamente a pivotagem transforma nosso banco de dados do formato largo para longo (ou vice-versa) e, dessa forma, alterando suas dimensões.
Observe a Figura 6 em que é ilustrada a pivotagem de uma tabela do formato largo (wide) para longo (long).
Figura 6: Transformação de tabelas do formato longo para largo.
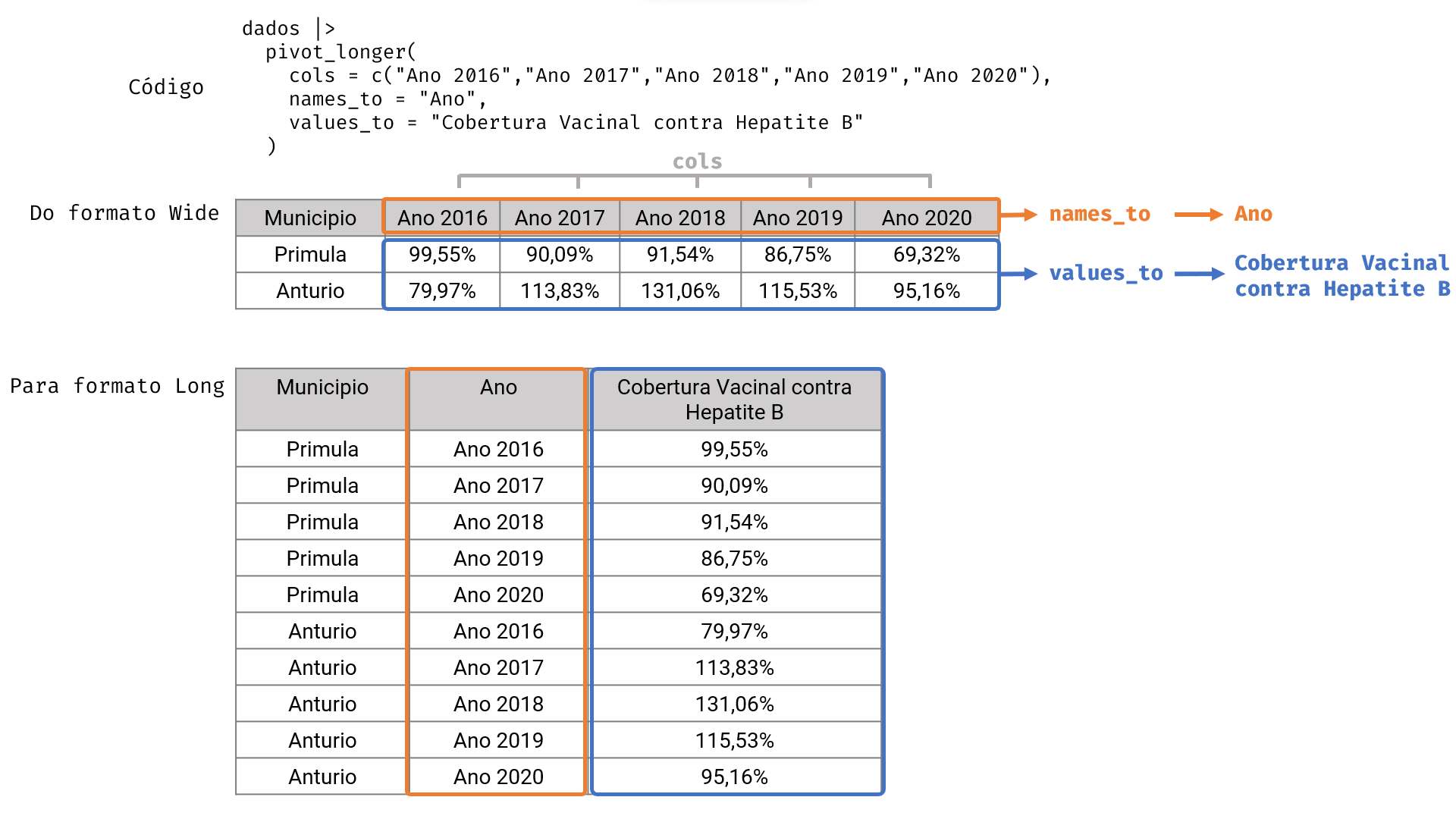
Para seguir com a nossa análise de cobertura vacinal será necessária
a reformatação da tabela {dados} fazendo-se a transposição
de suas seis colunas em três novas variáveis:
Unidade da federação, Ano e
Cobertura vacinal. Acompanhe o passo a passo abaixo:
Criaremos três variáveis na tabela {
dados}: Unidade da federação; Ano e Cobertura vacinal.Na operação de pivotagem vamos, então, aumentar o número de linhas e diminuir o número de colunas, deixando a tabela no formato longo. Para realizar esta operação no
R, vamos utilizar a funçãopivot_longer()do pacotetidyr. Essa função possui três argumentos principais:cols: neste argumento especificamos quais colunas vamos transformar;names_to: neste argumento definimos o nome da variável que estamos criando e que receberá a característica que estamos trazendo do formato largo;values_to: aqui definimos o nome da variável que receberá os valores, os dados propriamente ditos.
Acompanhe os códigos abaixo e replique-os em seu
RStudio:
# realizando a transposição dos dados do formato LARGO (*wide*)
# para uma tabela em formato LONGO (*long*)
dados |>
# Utilizando a função `pivot_longer()` para transformação de colunas
pivot_longer(
# Definindo as colunas que serão transformadas
cols = c("Ano 2016","Ano 2017","Ano 2018","Ano 2019","Ano 2020"),
# Definindo o nome da variável nova que receberá os nomes acima
names_to = "Ano",
# Definindo o nome da variável nova que receberá os valores da tabela
values_to = "Cobertura Vacinal contra Hepatite B",
)#> # A tibble: 10 × 3
#> Municipio Ano `Cobertura Vacinal contra Hepatite B`
#> <chr> <chr> <chr>
#> 1 Primula Ano 2016 99.55%
#> 2 Primula Ano 2017 90.09%
#> 3 Primula Ano 2018 91.54%
#> 4 Primula Ano 2019 86.75%
#> 5 Primula Ano 2020 69.32%
#> 6 Anturio Ano 2016 79.97%
#> 7 Anturio Ano 2017 113.83%
#> 8 Anturio Ano 2018 131.06%
#> 9 Anturio Ano 2019 115.53%
#> 10 Anturio Ano 2020 95.16%A tabela agora contém três colunas e dez linhas. Além disso, cada linha representa um registro específico da cobertura vacinal contra hepatite B, ano e município correspondente. Dessa forma o registro anual está concentrado em uma única coluna, e será possível a construção de um gráfico, por exemplo, de série histórica das coberturas vacinais.
Além dos argumentos citados acima, vamos precisar de mais um para
atender a um detalhe da nossa transformação. Um pequeno problema surgiu
ao transformar a base: toda a coluna Ano contém a palavra
“Ano” antes de cada valor.
Para corrigir isso, vamos utilizar o argumento
name_prefix, definindo a palavra que está repetindo na
conversão da coluna para linha. Basta adicionarmos a palavra entre
aspas. Veja como ficaria o comando incluindo o argumento
name_prefix e a tabela resultante. Vamos salvar todas as
modificações da tabela {dados} em novo objeto que
chamaremos de {dados_longos}, replique o script
abaixo em seu RStudio:
# Criando o objeto {`dados_longos`}
dados_longos <- dados |>
# Utilizando a função `pivot_longer()` para transformação de colunas
pivot_longer(
# Definindo as colunas que serão transformadas
cols = c("Ano 2016", "Ano 2017", "Ano 2018", "Ano 2019", "Ano 2020"),
# Definindo o nome da variável nova que receberá os nomes acima
names_to = "Ano",
# Definindo o nome da variável nova que receberá os valores da tabela
values_to = "Cobertura Vacinal contra Hepatite B",
# Retirando a palavra "Ano " antes de cada valor da variável Ano
# Também estamos retirando o espaço depois da palavra "Ano"
names_prefix = "Ano "
)
# Visualizando a tabela {`dados_longos`} no formato longo
dados_longos#> # A tibble: 10 × 3
#> Municipio Ano `Cobertura Vacinal contra Hepatite B`
#> <chr> <chr> <chr>
#> 1 Primula 2016 99.55%
#> 2 Primula 2017 90.09%
#> 3 Primula 2018 91.54%
#> 4 Primula 2019 86.75%
#> 5 Primula 2020 69.32%
#> 6 Anturio 2016 79.97%
#> 7 Anturio 2017 113.83%
#> 8 Anturio 2018 131.06%
#> 9 Anturio 2019 115.53%
#> 10 Anturio 2020 95.16%Neste exemplo a unidade de tempo utilizada foi o ano. Mas, na rotina, várias outras medidas de tempo são comuns, como semanas epidemiológicas e a data dos primeiros sintomas. Estas duas, em particular, tendem a deixar a tabela muito “larga”, com muitas colunas (52 ou 53 colunas no caso das semanas epidemiológicas e 365 no caso dos dias) e por isso sempre as utilizamos no formado longo.
Já em outras situações pode ser necessário transformar uma tabela em
formato longo para o largo. Ou seja, transpor o conteúdo armazenado em
linhas para colunas. Isso pode ser útil para uma melhor visualização de
uma tabela em um relatório, por exemplo. Para realizar esta operação
faremos o uso da função pivot_wider().
A função pivot_wider() é muito parecida com a
pivot_longer() e tem os seguintes argumentos
principais:
names_from: argumento para especificar qual coluna será transposta;values_from: argumento para especificar qual coluna contém os valores a serem pivotados.
Vamos praticar a utilização da função pivot_wider()!
Considere utilizar novamente a tabela que criamos anteriormente
{dados_longos} mas, dessa vez, vamos retornar a seu formato
largo. Observe o código abaixo e replique-o em seu
RStudio para pivotar os dados de imunização:
# Transformando os dados no formato longo em formato largo
# Criando o objeto {`dados_largos`}
dados_largos <- dados_longos |>
# Utilizando a função `pivot_wider()` para transformação de colunas
pivot_wider(
# Definindo de qual variável estamos resgatando os nomes das colunas
names_from = "Ano",
# Definindo de qual variável estamos resgatando os valores das colunas
values_from = "Cobertura Vacinal contra Hepatite B")
# visualizando a tabela
dados_largos#> # A tibble: 2 × 6
#> Municipio `2016` `2017` `2018` `2019` `2020`
#> <chr> <chr> <chr> <chr> <chr> <chr>
#> 1 Primula 99.55% 90.09% 91.54% 86.75% 69.32%
#> 2 Anturio 79.97% 113.83% 131.06% 115.53% 95.16%Observe como a mudança na apresentação da tabela modifica a visualização dos dados. Devemos sempre realizar estas transformações para enxergar todas as variáveis do banco de dados e manipulá-las de forma eficiente. Tente utilizar esta etapa nas suas análises do dia a dia!
6.3 Separar conteúdo de variáveis em mais colunas
Você já se deparou alguma vez com exportações dos sistemas de informações que contêm variáveis com mais de um dado na mesma célula?
Isso pode ser um pouco frustrante quando tentamos tabular e analisar dados. Quando a formatação de uma base retangular não é seguida demoramos muito tempo arrumando estes dados. Além disso, pode tornar nossas operações de manipulação e análise inseguras, aumentando as chances de errar. Vamos resolver este problema. Acompanhe o exemplo abaixo.
O setor de Imunização do Estado de Rosas lhe pediu apoio para
analisar os eventos adversos pós-vacinais. Para isto, enviou um banco de
dados { notificacao_eapv_2021m.xlsx } com os registros de
eventos adversos pós-vacinais com notificações realizadas no ano de 2021
no Brasil.
Lembre-se que todos os bancos de dados que utilizaremos para nossas análises encontram-se no menu lateral “Arquivos” no Ambiente Virtual deste módulo.
Rode o script abaixo para importar o banco de dados para
manipulá-lo no R, replique o código em seu computador:
# Importando o banco de dados { `notificacao_eapv_2021m.xlsx` } para o `R`
eapv_2021m <- read_xlsx('Dados/notificacao_eapv_2021m.xlsx')Os profissionais de imunização selecionaram algumas variáveis relacionadas à vacina administrada para serem analisadas:
imunobiologico_vacinacontendo o nome da vacina,dosecontendo o número da dose aplicada, edata_da_aplicacao, que armazena a data em que as respectivas doses foram aplicadas.
Mas, ao abrir o arquivo, você percebeu que as informações estão
organizadas de uma forma que dificulta a análise, com várias informações
separadas pelo símbolo (|) (barra horizontal) em uma mesma
variável.
Observe esta tabela utilizando a função View(). Escreva
o script abaixo em seu RStudio:
# Visualizando a tabela {`eapv_2021m`} com a função `View()
View(eapv_2021m)Observe que os dados estão dispostos como na Figura 7. Ao utilizar a
função View() você deve ter percebido que se abriu uma nova
janela no RStudio, que mostrará a tabela contida no objeto
{eapv_2021m}. Veja:
Figura 7: Tela de visualização da tabela {eapv_2021m}.
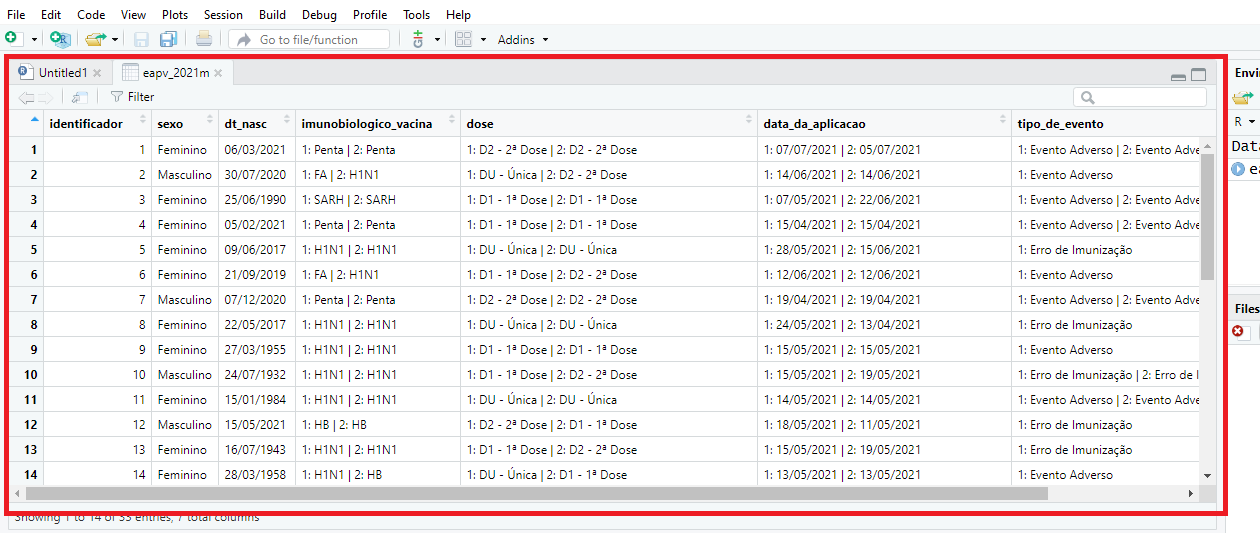
Perceba que algumas colunas possuem um ou mais dados referentes a um
mesmo indivíduo separados por um caractere (|) (barra
horizontal). Rode em seu RStudio o script abaixo e
visualize apenas as colunas imunobiologico_vacina,
dose e data_da_aplicacao da tabela
{eapv_2021m}:
eapv_2021m |>
# Selecionando três colunas do data.frame {`eapv_2021m`}
select(imunobiologico_vacina, dose, data_da_aplicacao) |>
# Utilizando a função `head()` para visualizar as primeiras linhas da tabela
head()#> # A tibble: 6 × 3
#> imunobiologico_vacina dose data_da_aplicacao
#> <chr> <chr> <chr>
#> 1 1: Penta | 2: Penta 1: D2 - 2ª Dose | 2: D2 - 2ª Dose 1: 07/07/2021 | 2: 05…
#> 2 1: FA | 2: H1N1 1: DU - Única | 2: D2 - 2ª Dose 1: 14/06/2021 | 2: 14…
#> 3 1: SARH | 2: SARH 1: D1 - 1ª Dose | 2: D1 - 1ª Dose 1: 07/05/2021 | 2: 22…
#> 4 1: Penta | 2: Penta 1: D1 - 1ª Dose | 2: D1 - 1ª Dose 1: 15/04/2021 | 2: 15…
#> 5 1: H1N1 | 2: H1N1 1: DU - Única | 2: DU - Única 1: 28/05/2021 | 2: 15…
#> 6 1: FA | 2: H1N1 1: D1 - 1ª Dose | 2: D2 - 2ª Dose 1: 12/06/2021 | 2: 12…Veja que para uma mesma pessoa as informações sobre diferentes doses, datas de aplicações e imunobiológicos utilizados em cada aplicação estão mesclados em uma única coluna para cada tipo de informação.
Agora, imagine que você deva utilizar as informações destas colunas para calcular se houve erro de imunização em relação ao intervalo adequado de aplicação das vacinas entre a 1ª, 2ª ou 3ª doses? Com a informação desta forma, fica muito difícil não é mesmo?
Para resolver este problema e conseguir analisar estes dados de forma adequada, devemos realizar a separação dos valores agregados em uma coluna em diversas outras variáveis. Essa operação reorganizará os valores em mais colunas, respeitando a lógica necessária para que cada célula tenha um conteúdo específico e único daquele registro.
Para esta etapa separaremos a coluna de vacina do exemplo acima,
utilizando a função separate() do pacote
tidyr. Essa função possui três argumentos básicos:
col: indicação da coluna que será separada;into: indicação dos nomes das novas colunas a serem criadas;sep: indicação de qual separador está utilizado na célula.
Veja abaixo a aplicação da função separate(). Em seu
RStudio replique o script abaixo:
eapv_2021m |>
# Dividindo a coluna `imunobiologico_vacina` em três novas colunas
separate(
# Definindo a coluna que será separada
col = imunobiologico_vacina,
# Definindo os nomes das novas colunas
into = c("vac_event_1", "vac_event_2", "vac_event_3"),
# Definindo qual o caractere que está sendo utilizado dentro das colunas
sep = "\\|"
) |>
# Selecionando as novas colunas para visualização
select("vac_event_1", "vac_event_2", "vac_event_3")#> Warning: Expected 3 pieces. Missing pieces filled with `NA` in 32 rows [1, 2, 3,
#> 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, ...].#> # A tibble: 33 × 3
#> vac_event_1 vac_event_2 vac_event_3
#> <chr> <chr> <chr>
#> 1 "1: Penta " " 2: Penta" <NA>
#> 2 "1: FA " " 2: H1N1" <NA>
#> 3 "1: SARH " " 2: SARH" <NA>
#> 4 "1: Penta " " 2: Penta" <NA>
#> 5 "1: H1N1 " " 2: H1N1" <NA>
#> 6 "1: FA " " 2: H1N1" <NA>
#> 7 "1: Penta " " 2: Penta" <NA>
#> 8 "1: H1N1 " " 2: H1N1" <NA>
#> 9 "1: H1N1 " " 2: H1N1" <NA>
#> 10 "1: H1N1 " " 2: H1N1" <NA>
#> # … with 23 more rowsComo resultado será armazenada uma nova tabela em que a antiga coluna
imunobiologico_vacina foi dividida em três:
vac_event_1, vac_event_2 e
vac_event_3. Além disso, o R retorna uma
mensagem de warning avisando que algumas linhas foram
preenchidas com NA. Isso se deve a alguns registros não
estarem preenchidos com três vacinas, apenas duas. E nós definimos que
criaríamos três novas colunas. Perceba que, no código acima,
selecionamos as novas colunas para visualização.
Perceba que com poucas linhas de comando foi possível organizar a
tabela e visualizar cada uma das doses aplicadas em uma coluna sem
perder nenhuma informação. Esta etapa tornará sua análise de dados mais
segura e rápida. Para separar outras colunas, repita o processo no seu
RStudio.
Uma observação importante a ser destacada é o uso do argumento
sep. Ele foi utilizado indicando duas barras invertidas e
uma vertical sep = "\\|". Isto ocorreu porque o símbolo
| é um caractere especial, e queremos que ele seja
interpretado como um caractere comum pelo uso da barra invertida
(\). Entretanto, a própria barra invertida é também um
símbolo especial, de modo que precisamos utilizar outra barra invertida
novamente para que ela seja interpretada corretamente pelo
R.
6.4 Recodificando linhas e colunas
Muitas vezes no processo de coleta e digitação de dados ocorrem alguns erros ortográficos que precisarão ser corrigidos antes da análise. Por exemplo, ao preencher o sexo dos pacientes atendidos em uma unidade de saúde, pode-se acabar digitando “masculinA” para um indivíduo e “masculinO” para outro, criando duas categorias diferentes que se referem ao mesmo tipo de sexo biológico. Para evitar esse tipo de erro, em uma análise adequada precisamos que as variáveis sejam recodificadas.
A recodificação em banco de dados é um procedimento frequente, pois, além de padronizar eliminando erros, possibilita a redução do tamanho do banco de dados em si, por armazenar códigos e não todo o texto da variável, além de tornar a programação de algoritmos mais eficiente. É importante frisar que para uma análise exploratória faz-se necessária a recodificação, a transformação dos códigos em dados mais informativos.
Para fazer esta recodificação utilizaremos a função
case_when() do pacote dplyr no R. Esta função
pode ser usada para criar variáveis a partir de variáveis existentes.
Nesta função, cada “caso” é separado por vírgulas. Neste argumento,
utilizamos o formato A ~ B, em que A é um
código com uma proposição lógica (por exemplo, se uma variável é igual a
determinado valor), e B indicará por qual valor iremos
substituir os casos que atendem ao critério descrito na função. O
operador til (~) faz essa comunicação.
Vejamos um exemplo de recodificação:
O profissional de vigilância necessita incluir em sua avaliação
demográfica a distribuição do sexo (CS_SEXO) dos agravos
contidos no Sinan Net. Para isto, utilizaremos o recorte do banco de
dados {NINDINET.dbf} que chamamos de
{base_menor}, criada anteriormente. Recodificaremos da
seguinte forma:
- “M” para “Masculino”;
- “F” para “Feminino”;
- “I” para “Ignorado”, incluiremos também os registros em branco ou nulos como “Ignorado” e;
- se não existir nenhum registro no banco de dados que atenta aos
critérios, um valor
NAserá atribuído.
Já vimos isso anteriormente e será um bom momento para fixarmos o
conteúdo. Observe o script abaixo em que será aplicada a
recodificação com atenção e replique-o em seu RStudio:
base_menor |>
# Renomeando os valores da variável CS_SEXO usando a função `mutate()` e
# `case_when()`
mutate(
sexo_cat = case_when(
CS_SEXO == "M" ~ "Masculino",
CS_SEXO == "F" ~ "Feminino",
CS_SEXO == "I" | is.na(CS_SEXO) ~ "Ignorado",
TRUE ~ NA_character_
)
) |>
# Visualizando a tabela {`base_menor`} recodificada
head()#> DT_NOTIFIC DT_NASC CS_SEXO CS_RACA ID_MN_RESI ID_AGRAVO sexo_cat
#> 1 2012-04-11 2012-04-04 M 4 610213 A509 Masculino
#> 2 2010-09-17 1988-04-23 M 1 610213 W64 Masculino
#> 3 2010-10-19 1971-03-25 M NA 610250 X58 Masculino
#> 4 2008-04-14 1928-05-29 F 4 610213 A90 Feminino
#> 5 2011-06-20 2002-09-18 M 4 610250 B19 Masculino
#> 6 2008-02-12 1953-08-01 F 9 610213 A90 FemininoObserve em seu output como a função case_when()
criou os valores para a nova coluna:
CS_SEXO == "M" ~ "Masculino": Se o valor na coluna CS_SEXO for “M”, então o valor na coluna deve ser “Masculino”;CS_SEXO == "F" ~ "Feminino": Se o valor na coluna CS_SEXO for “F”, então o valor na coluna deve ser “Feminino”;CS_SEXO == "I" | is.na(CS_SEXO) ~ "Ignorado": Se o valor na coluna CS_SEXO for “I” OU nulo, o valor na coluna deve ser “Ignorado”. TRUE ~ NA_character_: Se nenhum dos critérios anteriores forem atendidos, o valor da nova coluna deverá ser NA. Como a nova variável criada será do tipocharacter, oNApode também ser desse tipo.
Agora, repetiremos a mesma operação realizada para a variável
epidemiológica Raça/cor (CS_RACA), veja mais uma vez:
base_menor |>
# Renomeando os valores da variável CS_RACA usando a função `mutate()` e
# `case_when()`
mutate(
raca_cor_cat = case_when(
# Se o valor da coluna for igual a "1" transforme para "Branca"
CS_RACA == "1" ~ "Branca",
# Se o valor da coluna for igual a "2" transforme para "Preta"
CS_RACA == "2" ~ "Preta",
# Se o valor da coluna for igual a "3" transforme para "Amarela"
CS_RACA == "3" ~ "Amarela",
# Se o valor da coluna for igual a "4" transforme para "Parda"
CS_RACA == "4" ~ "Parda",
# Se o valor da coluna for igual a "5" transforme para "Indígena"
CS_RACA == "5" ~ "Indígena",
# Se o valor da coluna for igual a "9" ou nulo transforme para "Ignorado"
CS_RACA == "9" | is.na(CS_RACA) ~ "Ignorado",
# Caso acontecer um valor diferente dos citados acima, transforme para "NA"
TRUE ~ NA_character_
)
) |>
# Utilizando a função `head()` para visualizar as primeiras linhas
head()#> DT_NOTIFIC DT_NASC CS_SEXO CS_RACA ID_MN_RESI ID_AGRAVO raca_cor_cat
#> 1 2012-04-11 2012-04-04 M 4 610213 A509 Parda
#> 2 2010-09-17 1988-04-23 M 1 610213 W64 Branca
#> 3 2010-10-19 1971-03-25 M NA 610250 X58 Ignorado
#> 4 2008-04-14 1928-05-29 F 4 610213 A90 Parda
#> 5 2011-06-20 2002-09-18 M 4 610250 B19 Parda
#> 6 2008-02-12 1953-08-01 F 9 610213 A90 IgnoradoObserve que as variáveis foram todas recodificadas, facilitando a identificação do conteúdo da coluna sem uso do dicionário de dados.
Outra organização nos dados que o profissional de vigilância realiza com frequência para suas análises é a criação de categorias, como a transformação das idades em faixas etárias para construir análises comparativas entre grupos etários diferentes ou representação gráficas como pirâmides etárias. Para isso, a categorização é um procedimento que recodifica uma variável em tipo de dado diferente, pois está transformando um valor do tipo número em um valor do tipo texto.
Para fazer esta categorização utilizaremos aqui a função
if_else() do pacote dplyr no R.
Esta função cria uma coluna baseada a um critério específico,
equivalente às sentenças lógicas do tipo SE do Microsoft
Excel. Veja o exemplo abaixo.
Ainda utilizando a tabela {base}, que está armazenando
os dados importados do banco de dados {NINDINET.dbf}, o
profissional de vigilância necessitará analisar a distribuição por faixa
etária de todos os agravos notificados no Sinan Net.
Para facilitar a visualização vamos selecionar algumas colunas que alteramos. Calma que logo abaixo explicamos os códigos para você!
Insira os comandos do script abaixo no seu
RStudio e observe como ficaram as colunas modificadas:
base |>
# Utilizando a função `mutate()` para criar colunas
mutate(
# Criando uma coluna de idade conforme a codificação da variável NU_IDADE_N
idade_anos = if_else(str_sub(NU_IDADE_N, 1, 1) == "4",
as.numeric(str_sub(NU_IDADE_N, 2, 4)), 0),
# Criando uma coluna de faixa etária a partir da variável idade dos casos
# notificados
# utilizando a função `cut()`
fx_etaria = cut(
# Definindo qual variável será classificada em faixas
x = idade_anos,
# Definindo os pontos de corte das classes
breaks = c(0, 10, 20, 60, Inf),
# Definindo os rótulos das classes
labels = c("0-9 anos", "10-19 anos", "20-59 anos", "60 anos e+"),
# Definindo o tipo do ponto de corte
right = FALSE
)
) |>
# Selecionando as variáveis que queremos utilizar com a função `select()`
select(NU_NOTIFIC, ID_AGRAVO, NU_IDADE_N, idade_anos, fx_etaria) |>
# Visualizando a tabela {`base`} modificada
head()#> NU_NOTIFIC ID_AGRAVO NU_IDADE_N idade_anos fx_etaria
#> 1 7671320 A509 2001 0 0-9 anos
#> 2 0855803 W64 4022 22 20-59 anos
#> 3 8454645 X58 4039 39 20-59 anos
#> 4 3282723 A90 4079 79 60 anos e+
#> 5 9799526 B19 4008 8 0-9 anos
#> 6 7275624 A90 4054 54 20-59 anosO código acima possui vários detalhes que são necessários explicar.
Perceba que estamos utilizando a função mutate() para criar
duas colunas: idades_anos e fx_etaria. Na nova
variável idade_anos usamos a função if_else(),
que verifica se o código da idade no banco de dados é “4” (que significa
que o registro da idade é em anos).
Isso é necessário pois o primeiro dígito da variável idade no banco
do Sinan Net informa se a idade está em anos, meses, dias ou horas. Se o
critério for verdadeiro, a função str_sub() do pacote
stringr extrai os dois últimos dígitos, pegando somente o
valor da idade. Se o critério não for verdadeiro, será registrado o
valor zero. A função as.numeric() transforma o resultado
para o tipo numérico.
Para criar a variável fx_etaria, estamos utilizando a
função cut(). Essa função classifica a variável
recém-criada idade_anos em quatro categorias. Os argumentos
utilizados pela função são:
x: variável numérica que será categorizada;breaks: cortes de idades. Aqui definimos os cortes começando em 0, depois em 10, 20, 60 e, o último, incluirá qualquer valor depois de 60 (Inf, que denota infinito);labels: rótulos para as classes;right: define se corte da categoria acontece antes ou depois dos números definidos embreaks. Por exemplo, comright = TRUE, uma idade de 20 anos será incluída na categoria 10-19 anos. Seright = FALSE, a idade de 20 anos será incluída na categoria 20-59 anos.
Pronto. Perceba que agora temos os casos notificados no Sinan Net
organizados por faixa etária na variável fx_etaria, nas
seguintes categorias: “0-9 anos”, “10-19”, “20-59 anos”, “60 anos e+”.
Após esta categorização você será capaz de perceber se existem diferença
entre os agravos em relação à idade.
Agora que você já sabe reorganizar suas tabelas, corrigir erros de codificação, recategorizá-las e armazenar corretamente seus dados, vamos cruzar tabelas para construir sua análise de situação de saúde.


