4. Preparando as variáveis para análise de dados de vigilância
Com o R poderemos construir as tabelas personalizadas
para nossas análises. Nesta seção você irá conhecer algumas maneiras de
selecionar e transformas variáveis de interesse para vigilância em
saúde.
4.1 Excluindo colunas na tabela
Quase sempre o profissional de vigilância necessita excluir colunas e
reorganizar a sua ordem na tabela, e você já aprendeu neste curso que
podemos selecionar colunas no R utilizando a função
select(). Agora, utilizaremos esta função para excluir
colunas.
Esta etapa é importante, pois permitirá excluir dados que não serão
utilizados em nossa análise, tornando nossa tabela menor e mais “leve”,
reduzindo o tempo gasto para seu processamento com R.
Para isso, a função select() deverá receber a inclusão
do operador lógico subtração (“-”). A subtração deverá ser
acrescentada antes do nome da coluna a ser excluída.
Para praticarmos excluiremos a variável de data de nascimento
(DT_NASC) da tabela {base_menor} criada
anteriormente. Acompanhe o passo a passo para excluirmos a variável
DT_NASC do data.frame {base_menor} e
replique-o em seu RStudio:
base_menor |>
# Excluindo a variável "DT_NASC" da tabela {`base_menor`}
select(-DT_NASC) |>
# Utilizando a função `head()` para visualizar as primeiras linhas da tabela
# após a exclusão
head()#> DT_NOTIFIC CS_SEXO CS_RACA ID_MN_RESI ID_AGRAVO
#> 1 2012-04-11 M 4 610213 A509
#> 2 2010-09-17 M 1 610213 W64
#> 3 2010-10-19 M NA 610250 X58
#> 4 2008-04-14 F 4 610213 A90
#> 5 2011-06-20 M 4 610250 B19
#> 6 2008-02-12 F 9 610213 A90Observe em seu output que o resultado da execução possui uma
tabela com todas as colunas presentes em {base_menor},
exceto a variável DT_NASC. Fácil, não é mesmo?!
Atenção
Lembre-se de conferir todos os símbolos, palavras e pontos do seu
código! O R retorna erros quando seu código está escrito de
forma incorreta.
4.2 Inserindo novas colunas à tabela
Outra ação corriqueira quando analisamos dados de vigilância é
adicionarmos colunas ao nosso banco de dados, seja com o resultado de
algum cálculo, com informações de confirmação de um caso suspeito, ou
ainda uma coluna com o status de uma investigação de surto. Para criar
uma variável no R utilizamos a função
mutate(). Seguiremos, portanto, o seguinte passo a
passo:
- definir o nome da coluna;
- utilizar o sinal de igual (
=) e; - escrever os comandos para incluir os valores que preencherão a nova coluna.
Lembre-se que estamos analisando a situação das notificações de casos no Estado de Rosas. Esta etapa apoiará nossa avaliação de sensibilidade do sistema de vigilância em saúde, pois criaremos uma nova variável que armazena valores com o tempo (em dias) que está levando para que uma notificação seja digitada no Sinan Net, ou seja, o tempo em dias de atraso da digitação das notificações do Estado de Rosas.
Para isso, criaremos a variável que daremos o nome de
TEMPO_DIGITA, que será a diferença em dias entre a coluna
com valores da data de digitação (DT_DIGITA) e a coluna
data de notificação (DT_NOTIFIC). Como padrão, o
R adicionará a nova variável criada ao final da tabela
(última coluna).
Primeiro criamos um objeto que será chamado
{base_menor_2}. Ele receberá as colunas selecionadas e seus
respectivos registros da tabela {base} (criada
anteriormente a partir do arquivo {NINDINET.dbf}). Observe
o script abaixo e execute os comandos em seu
RStudio:
# Criando a {`base_menor_2`}
base_menor_2 <- base |>
# Selecionando as variáveis que queremos utilizar com a função `select()`
select(NU_NOTIFIC, ID_AGRAVO, DT_NOTIFIC, DT_DIGITA) |>
# Utilizando a função `mutate()` para criar a nova coluna
mutate(TEMPO_DIGITA = DT_DIGITA - DT_NOTIFIC)Agora, digite os códigos abaixo em seu script e pressione Run para visualizar como ficou sua nova variável criada:
# Utilizando a função `head()` para visualizar as primeiras linhas do novo objeto
base_menor_2 |> head()#> NU_NOTIFIC ID_AGRAVO DT_NOTIFIC DT_DIGITA TEMPO_DIGITA
#> 1 7671320 A509 2012-04-11 2012-11-09 212 days
#> 2 0855803 W64 2010-09-17 2010-11-17 61 days
#> 3 8454645 X58 2010-10-19 2011-03-14 146 days
#> 4 3282723 A90 2008-04-14 2008-04-24 10 days
#> 5 9799526 B19 2011-06-20 2011-09-14 86 days
#> 6 7275624 A90 2008-02-12 2008-02-26 14 daysVeja que esta nova tabela possui a coluna TEMPO_DIGITA
no final, e suas linhas são a diferença da data - em dias - de digitação
e de notificação. Após o número de dias, você verá a informação
days, indicando que esta variável se trata de uma variável
de tempo (classe = difftime).
Para fazer cálculos matemáticos e novas análises, precisaremos
padronizar o tipo de dado contido na variável TEMPO_DIGITA
e, aqui, optaremos pelo tipo numérico (numeric, em inglês).
Neste caso, é possível alterar o tipo de dado da variável atribuindo-lhe
o mesmo nome. Observe os script abaixo e replique-o em seu
computador:
base_menor_2 |>
# Transformando a coluna "TEMPO_DIGITA" da tabela {`base_menor_2`}
# no tipo numérico com a função `as.numeric()` dentro da função `mutate()`
mutate(TEMPO_DIGITA = as.numeric(TEMPO_DIGITA)) |>
# Utilizando a função `head()` para visualizar as primeiras linhas
head()#> NU_NOTIFIC ID_AGRAVO DT_NOTIFIC DT_DIGITA TEMPO_DIGITA
#> 1 7671320 A509 2012-04-11 2012-11-09 212
#> 2 0855803 W64 2010-09-17 2010-11-17 61
#> 3 8454645 X58 2010-10-19 2011-03-14 146
#> 4 3282723 A90 2008-04-14 2008-04-24 10
#> 5 9799526 B19 2011-06-20 2011-09-14 86
#> 6 7275624 A90 2008-02-12 2008-02-26 14Visualize seu output e perceba que a coluna
TEMPO_DIGITA é composta agora apenas por números
(numeric), e não mais uma medida de tempo
(difftime), nos apoiando a compreender o tempo que está
levando para que as notificações de agravos do município de Rosas sejam
digitadas no Sinan Net, em dias.
4.3 Limpando caracteres e padronizando colunas
Em análise de dados, algumas vezes não é possível reconhecer de imediato o formato de colunas ou mesmo se torna complicado utilizar caracteres como acentos, espaço entre as palavras, símbolos e outros, até mesmo nas funções de código.
Para resolver estes problemas, quase sempre necessitamos padronizar o nome das colunas do nosso banco de dado, limpando seus nomes e formatando-os para que haja uma fácil manipulação dos dados.
Para executarmos estas ações com o R, utilizamos o
pacote janitor e sua função clean_names().
Esta função tornará todas as variáveis minúsculas e retirará os
caracteres como acentos e os espaços entre as palavras, quando
houver.
Para esta etapa, utilizaremos o banco de dados do tipo
.csv que contêm os códigos do CID-10 exportados diretamente
do site do Datasus: o {CID-10-CATEGORIAS.csv}. Este arquivo
é muito utilizado para agrupar categorias de doenças, possibilitando a
recategorização de CIDs em todos os bancos de dados do Datasus. Você o
encontrará disponível no menu lateral “Arquivos”, no Ambiente Virtual do
curso. Acompanhe o passo a passo abaixo e replique-o em seu
RStudio:
- Primeiro, importe o arquivo {
CID-10-CATEGORIAS.csv}, armazenando-o no objeto do tipo data.frame {cid10_categorias}. Replique o script abaixo para importá-lo para o seuR:
# Importando o banco de dados { `CID-10-CATEGORIAS.CSV } para o `R`
cid10_categorias <- read_csv2('Dados/CID-10-CATEGORIAS.CSV')- Acrescente ao script a função
colnames()para inspecionar o nome das colunas com o seguinte código:
# Inserindo a função `colnames()` para checar as variáveis
colnames(cid10_categorias)#> [1] "CAT" "CLASSIF" "DESCRICAO" "DESCRABREV" "REFER"
#> [6] "EXCLUIDOS"Observe o output em seu console. Você visualizará o vetor
contendo os nomes de todas as colunas de sua tabela
cid10_categorias.
- Agora utilize a função
clean_names()do pacotejanitorao rodar o seguinte código:
# Utilizando a função `clean_names()` para editar o nome das variáveis
cid10_categorias_nova <- clean_names(cid10_categorias)
# Visualizando as variáveis após a transformação
colnames(cid10_categorias_nova)#> [1] "cat" "classif" "descricao" "descrabrev" "refer"
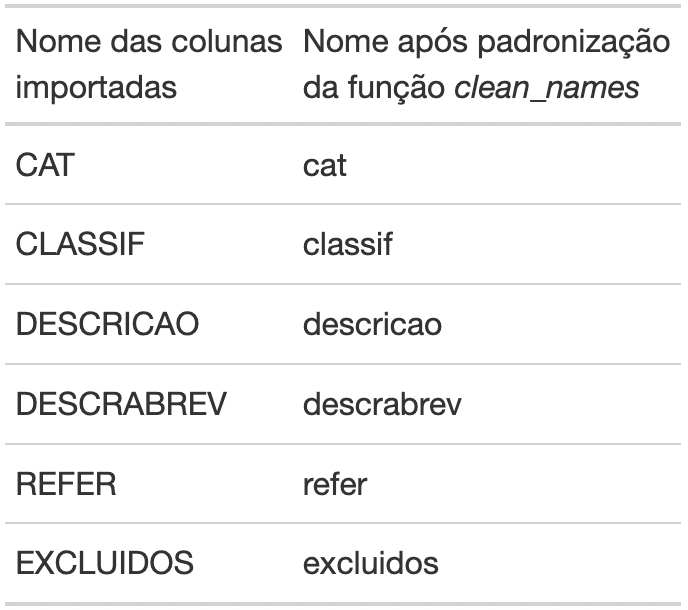
#> [6] "excluidos"Perceba que os nomes das variáveis estão totalmente em minúsculo, sem acentos e sem espaço entre as palavras.
Observe de forma detalhada a comparação dos nomes das colunas antes e
após a transformação que realizamos com a função
clean_names().
Figura 2: Tabela comparativa da transformação das variáveis
com a função clean_names().
Atenção
Você deverá repetir este processo muitas vezes durante sua análise de dados. Esta etapa é fundamental para todos os próximos passos que daremos neste curso.
Ela nos permitirá ter segurança e não errar o nome das variáveis ao ter que digitá-los novamente para realizar filtros, seleções ou edições.
4.4 Filtrando colunas
Muito bem, já vimos como excluir colunas, transformá-las e renomeá-las para facilitar a análise dos dados. Agora, você irá aprender a filtrar os registros de uma tabela para obter as informações precisas para sua análise.
Para isso, utilizaremos a função filter() que permitirá
filtrar os valores (linhas) da variável selecionada a partir de um ou
mais critérios necessários. Para realizar esses filtros precisamos
acrescentar os operadores lógicos indicando as ações de filtragem. Veja
na lista abaixo quais são os principais operadores e seus usos:

Vamos praticar! Utilizaremos o data.frame
{base_menor_2}, criada anteriormente a partir da tabela
{base}, para filtrar todos os registros que contenham o
código da CID-10 referente à hepatite viral não especificada (B19) e que
o tempo de digitação foi maior que sete dias. Execute o script
abaixo em seu RStudio:
base_menor_2 |>
# Filtrando os CIDs igual a B19, da coluna "ID_AGRAVO"
# com tempo de digitação maior que sete dias
filter(ID_AGRAVO == "B19", TEMPO_DIGITA > 7) |>
# Utilizando a função `head()` para visualizar as primeiras linhas da tabela
head()#> NU_NOTIFIC ID_AGRAVO DT_NOTIFIC DT_DIGITA TEMPO_DIGITA
#> 5 9799526 B19 2011-06-20 2011-09-14 86 days
#> 34 4218628 B19 2009-07-10 2010-05-11 305 days
#> 51 9943142 B19 2010-08-03 2010-09-24 52 days
#> 76 1856028 B19 2009-06-22 2009-07-09 17 days
#> 85 0025650 B19 2011-01-31 2011-07-25 175 days
#> 91 0733923 B19 2007-01-25 2007-03-28 62 daysVeja que, por padrão, quando se utiliza a vírgula para separar os
diversos filtros que vamos utilizar
filter(ID_AGRAVO == "B19", TEMPO_DIGITA > 7), a função
filter() realiza a ação considerando o primeiro critério
E o segundo e assim sucessivamente.
Caso exista necessidade de que o filtro seja um critério do tipo
OU, você necessitará utilizar o operador barra
horizontal (|). Observe no exemplo abaixo que o filtro está
selecionando registros de notificação de hepatite (CID B19)
OU de leptospirose (CID A279) OU
malária (CID B54):
base_menor_2 |>
# Filtrando os registros iguais a B19 OU A279 OU B54 na coluna "ID_AGRAVO"
filter(ID_AGRAVO == "B19" | ID_AGRAVO == "A279" | ID_AGRAVO == "B54") |>
# Utilizando a função `head()` para visualizar as primeiras 20 linhas da tabela
head(20)#> NU_NOTIFIC ID_AGRAVO DT_NOTIFIC DT_DIGITA TEMPO_DIGITA
#> 5 9799526 B19 2011-06-20 2011-09-14 86 days
#> 34 4218628 B19 2009-07-10 2010-05-11 305 days
#> 51 9943142 B19 2010-08-03 2010-09-24 52 days
#> 76 1856028 B19 2009-06-22 2009-07-09 17 days
#> 85 0025650 B19 2011-01-31 2011-07-25 175 days
#> 91 0733923 B19 2007-01-25 2007-03-28 62 days
#> 106 8181226 B19 2009-07-06 2009-07-23 17 days
#> 112 5619327 A279 2009-12-30 2010-01-21 22 days
#> 128 4231128 B19 2009-08-04 2010-05-04 273 days
#> 157 6644624 B19 2007-01-17 2007-04-12 85 days
#> 160 4232928 B19 2009-09-29 2009-11-23 55 days
#> 169 4360123 B19 2012-12-27 2013-01-28 32 days
#> 172 1821124 B54 2008-08-11 2008-08-29 18 days
#> 175 1449327 B19 2010-05-13 2010-10-25 165 days
#> 190 1448227 B19 2009-08-05 2010-09-21 412 days
#> 198 1250223 B19 2007-02-15 2007-06-05 110 days
#> 212 8469526 B19 2008-01-18 2008-07-03 167 days
#> 220 9777227 B19 2009-04-22 2009-05-21 29 days
#> 231 9941142 B19 2010-08-03 2010-09-21 49 days
#> 242 2338425 B19 2008-01-14 2008-08-11 210 daysTambém é possível utilizar o operador (%in%) nesta
situação, quando a mesma variável (ID_AGRAVO) pode conter
vários valores passíveis de seleção (filtros). Nesta situação, portanto,
selecionamos os agravos dentro do conjunto dado pela função
c().
Acompanhe o script abaixo e replique-o:
base_menor_2 |>
# Filtrando os agravos utilizando o operador `%in%`
filter(ID_AGRAVO %in% c("B19", "A279", "B54")) |>
# Utilizando a função `head()` para visualizar as primeiras 20 linhas da tabela
head(20)#> NU_NOTIFIC ID_AGRAVO DT_NOTIFIC DT_DIGITA TEMPO_DIGITA
#> 5 9799526 B19 2011-06-20 2011-09-14 86 days
#> 34 4218628 B19 2009-07-10 2010-05-11 305 days
#> 51 9943142 B19 2010-08-03 2010-09-24 52 days
#> 76 1856028 B19 2009-06-22 2009-07-09 17 days
#> 85 0025650 B19 2011-01-31 2011-07-25 175 days
#> 91 0733923 B19 2007-01-25 2007-03-28 62 days
#> 106 8181226 B19 2009-07-06 2009-07-23 17 days
#> 112 5619327 A279 2009-12-30 2010-01-21 22 days
#> 128 4231128 B19 2009-08-04 2010-05-04 273 days
#> 157 6644624 B19 2007-01-17 2007-04-12 85 days
#> 160 4232928 B19 2009-09-29 2009-11-23 55 days
#> 169 4360123 B19 2012-12-27 2013-01-28 32 days
#> 172 1821124 B54 2008-08-11 2008-08-29 18 days
#> 175 1449327 B19 2010-05-13 2010-10-25 165 days
#> 190 1448227 B19 2009-08-05 2010-09-21 412 days
#> 198 1250223 B19 2007-02-15 2007-06-05 110 days
#> 212 8469526 B19 2008-01-18 2008-07-03 167 days
#> 220 9777227 B19 2009-04-22 2009-05-21 29 days
#> 231 9941142 B19 2010-08-03 2010-09-21 49 days
#> 242 2338425 B19 2008-01-14 2008-08-11 210 daysPronto, você armazenou na tabela ou data.frame
{base_menor_2} apenas os registros que contenham
notificações de hepatites virais (CID B19), leptospirose (CID A279) e
malária (CID B54).
A qualquer momento em que desejar obter uma tabela com dados que
contenham todas as notificações do Estado de Rosas você deverá utilizar
o objeto {base} criado no início deste módulo.
Lembre-se que o objeto {base} é do tipo
data.frame e armazenou os dados importados do banco de dados
{NINDINET.dbf}, disponibilizado no Ambiente Virtual do
curso.
4.5 Unindo colunas
Ainda na etapa de limpeza e organização de dados, frequentemente o profissional de vigilância precisa agrupar registros para suas análises. A variável idade é uma das variáveis epidemiológicas mais utilizadas, por exemplo, para divisão de grupos por faixa etária em quase todas as análises demográficas.
Na linguagem R, a função group_by() é o
verbo que utilizaremos para agrupamento dos registros de acordo com a
coluna especificada, sendo a variável o principal argumento da função.
Aqui, destacamos que é muito frequente seu uso com outras funções junto
ao banco de dados.
Perceba que na expressão abaixo realizamos comandos de agrupamento
(group_by) de todos os casos confirmados ou suspeitos
notificados pela mesma doença ou agravo (ID_AGRAVO). Em
seguida realizamos um comando para contar (usando a função
count()) quantos casos foram registrados com a mesma doença
ou agravo. Permaneceremos utilizando a tabela
{base_menor_2} criada anteriormente. Rode o código a seguir
em seu computador e verifique o resultado:
base_menor_2 |>
# Agrupando as notificações pelos agravos
group_by(ID_AGRAVO) |>
# Contando a frequência de notificações por agravos
count(ID_AGRAVO)#> # A tibble: 63 × 2
#> # Groups: ID_AGRAVO [63]
#> ID_AGRAVO n
#> <chr> <int>
#> 1 A010 2
#> 2 A051 2
#> 3 A059 2
#> 4 A09 4
#> 5 A169 2347
#> 6 A279 49
#> 7 A309 266
#> 8 A35 4
#> 9 A379 26
#> 10 A509 329
#> # … with 53 more rowsO código digitado foi executado e retornou um objeto do tipo
tibble e mostra três colunas: a primeira é uma coluna índice
que marca o número da linha, não sendo uma variável do banco em si; a
segunda é a ID_AGRAVO com todos os CID10 notificados
(variável do tipo character, <chr>); e a
n tem o número total de vezes que este agravo foi
notificado (variável do tipo numérica, <int>).
Perceba também, que ao final do código, há as mensagens:
#> # … with 53 more rows #> # ℹ Use print(n = …) to see more rows
Esses avisos aparecem porque o R retorna por padrão
apenas 10 linhas da tabela para visualização. Caso queira que ele
retorne mais linhas você deverá inserir o valor de linhas valor que
deseja visualizar na função print() dentro de seu argumento
n =. Observe o script abaixo como fazemos e
reproduza em seu RStudio:
base_menor_2 |>
# Agrupando as notificações pelos agravos
group_by(ID_AGRAVO) |>
# Contando a frequência de notificações por agravos
count(ID_AGRAVO) |>
# Visualizando as 20 primeiras linhas da tabela
# {`base_menor_2`}
# usando a função `print()`
print (n = 20)#> # A tibble: 63 × 2
#> # Groups: ID_AGRAVO [63]
#> ID_AGRAVO n
#> <chr> <int>
#> 1 A010 2
#> 2 A051 2
#> 3 A059 2
#> 4 A09 4
#> 5 A169 2347
#> 6 A279 49
#> 7 A309 266
#> 8 A35 4
#> 9 A379 26
#> 10 A509 329
#> 11 A510 1
#> 12 A513 3
#> 13 A53 121
#> 14 A530 1
#> 15 A539 234
#> 16 A60 32
#> 17 A63 7
#> 18 A630 76
#> 19 A64 5
#> 20 A779 12
#> # … with 43 more rowsAgora já conseguimos saber que a leptospirose (CID10 = A279) aparece notificada por 49 vezes nesta tabela. Esse pequeno comando é mais simples, seguro e rápido que contar no Excel ou gerar uma tabela dinâmica.
Assim, todas as vezes em que for necessário realizar a contagem das
frequências de uma variável epidemiológica, o profissional de vigilância
poderá utilizar o verbo group_by() para fazê-lo em
segundos.
4.6 Resumindo os valores de uma coluna
Outro aspecto importante do trabalho da vigilância é a capacidade de
extrair dados e resumi-los. Assim, nesta etapa você irá aprender a
função summarise(), similar à função mutate()
utilizada anteriormente. A principal diferença entre as duas funções é a
possibilidade de criação de novas colunas realizando operações de
síntese e resumo, conjuntamente. Assim, a função
summarise() possibilita o resumo de muitos valores em uma
única linha.
Para praticar o uso do summarise() calcularemos
a média do tempo de atraso (em dias) em que as equipes de
vigilância do Estado de Rosas têm digitado suas notificações
seguindo os seguintes passos:
utilizaremos como banco de dados a tabela {
base_menor_2} criada a partir da tabela {`base},em seguida, utilizaremos a função
group_by()para contabilizar o número de casos por agravos ou doenças notificados, utilizando a função de contagemn()da variávelTEMPO_DIGITA, criada anteriormente, epor fim, utilizaremos a função
mean(), a qual já vimos que calcula a média de uma coluna.
Agora observe o script abaixo e replique-o em seu
RStudio:
base_menor_2 |>
# Agrupando as notificações pelos agravos
group_by(ID_AGRAVO) |>
# Utilizando a função `summarise()` para criar novas colunas de síntese
summarise(
# Criando uma coluna de total, utilizando a função `n()`
total_agravos = n(),
# Criando uma coluna de média, utilizando a função `mean()`
media_digita = mean(TEMPO_DIGITA)
)#> # A tibble: 63 × 3
#> ID_AGRAVO total_agravos media_digita
#> <chr> <int> <drtn>
#> 1 A010 2 122.00000 days
#> 2 A051 2 10.50000 days
#> 3 A059 2 27.00000 days
#> 4 A09 4 193.50000 days
#> 5 A169 2347 151.67490 days
#> 6 A279 49 78.55102 days
#> 7 A309 266 165.64286 days
#> 8 A35 4 70.75000 days
#> 9 A379 26 36.03846 days
#> 10 A509 329 130.58055 days
#> # … with 53 more rowsO comando executado retornou quatro colunas:
- a primeira a coluna é a índice, que mostra o número da linha;
- a segunda é a
ID_AGRAVOcom todos os CID10 notificados (variável do tipo texto,<chr>); - a coluna total_agravos corresponde ao número total
de vezes que este agravo foi notificado (variável do tipo inteiro,
<int>) e; - por fim a variável que criamos media_digita
(variável do tipo data,
<drtn>).
Assim, com esta nova tabela conseguimos concluir que leptospirose (CID10 = A279) aparece notificada por 49 vezes e o tempo médio de digitação no sistema foi 78,55102 dias.
4.7 Modificando a ordem das colunas
No dia a dia é comum precisarmos modificar a ordem de apresentação
dos valores de uma coluna como por exemplo ordenar idades do mais velhos
para os mais novos, ou mesmo ordenar uma coluna por ordem alfabética,
como de A-Z ou de Z-A. Para esta reordenação de valores em uma variável
no R utilizamos a função arrange().
Para executar esta função, precisamos apenas das colunas que queremos
ordenar como argumento. Por padrão, essa função ordena do menor valor
para o maior (ordem crescente). Caso exista a necessidade de ordenar uma
coluna de forma decrescente, devemos também utilizar a função
desc(), como no exemplo abaixo:
arrange(desc(media_digita)).
Vejamos o mesmo exemplo anterior, mas agora adicionando o comando de ordenação decrescente para variável media_digita, apontando, assim, o agravo ou doença que leva o maior tempo para ser digitado no Estado de Rosas. Utilize os seguintes comandos:
base_menor_2 |>
# Agrupando as notificações pelos agravos
group_by(ID_AGRAVO) |>
# Utilizando a função `summarise()` para criar novas colunas de síntese
summarise(total_agravos = n(),
media_digita = mean(TEMPO_DIGITA)) |>
# Ordenando a tabela pela ordem decrescente da média de tempo de digitação
arrange(desc(media_digita))#> # A tibble: 63 × 3
#> ID_AGRAVO total_agravos media_digita
#> <chr> <int> <drtn>
#> 1 F99 3 666.6667 days
#> 2 C80 1 560.0000 days
#> 3 Z206 23 374.9130 days
#> 4 H833 11 331.6364 days
#> 5 N485 5 304.6000 days
#> 6 Z209 500 293.6880 days
#> 7 B09 109 283.8807 days
#> 8 B42 150 269.0333 days
#> 9 A630 76 263.8289 days
#> 10 R36 77 261.9740 days
#> # … with 53 more rowsComo resultado você verá quatro colunas. A primeira com o índice com o número da linha, a segunda com a identificação do código do agravo, a terceira com o total de casos e a última com a média do tempo de digitação para cada agravo.
Veja que como as colunas estão ordenadas em ordem decrescente, com o retorno do comando é possível visualizar o maior valor primeiro (666,6667 dias), que se refere ao agravo relacionado ao trabalho, e os valores com a menor média de digitação aparecem ao final da coluna.


