5 Realizando o linkage
De posse do SIVEP-Gripe devidamente deduplicado, armazenado no objeto
{sivep_dedup}, vamos prosseguir fazendo um linkage
com o SIM.
No SIM de Rosas temos uma base de dados sintética somente com as
variáveis: nomes, nome da mãe e
data de nascimento, não existindo uma variável com
identificador único como o número do CPF. Isto fará com que o
linkage entre as bases tenha de ser realizado com menos
informações.
Você deve ter percebido que nas bases de dados exportadas nos sistemas reais, tanto para o SIVEP-Gripe quando para o SIM, existiriam outras variáveis que poderiam nos apoiar neste pareamento de dados, como a evolução do caso e a data da evolução no SIVEP-Gripe, e a data de óbito no SIM junto a causa associada da morte, por exemplo.
Neste exemplo de linkage optamos por introduzir apenas a
causa básica principal (CAUSABAS) para ilustrar algumas
possibilidades de análise e estimulá-lo a experimentar em seu dia a dia.
Vamos lá!
Atenção
Todos os bancos de dados utilizados para análises neste curso se
encontram no menu lateral “Arquivos” do curso. Lembre-se de fazer o
download do material do curso diretamente do Ambiente Virtual
de Aprendizagem do curso e arquivá-los em um diretório que deverá
indicar para que o R o localize.
Em um primeiro passo vamos importar os registros fictícios do Sistema
de Informação de Mortalidade (SIM) do Estado de Rosas, uma base de nome
DO_ROSAS.csv. Não iremos realizar nenhum tipo de filtragem
nesta base, ou seja, utilizaremos o arquivo com todas as suas variáveis.
Você irá observar que o arquivo do SIM de Rosas possuí algumas das
principais causas de morte no estado.
Vamos lá! Copie e cole o código a seguir em seu RStudio
para importar a base de dados de óbitos de Rosas:
# Carregando a base de dados
do_rosas <- read_csv2("Dados/DO_ROSAS.csv")Conforme vimos neste curso, será necessário fazer a deduplicação da
base para executar o linkage, por isso faremos agora todos os
passos que você aprendeu de uma única vez. Fique atento que agora
teremos somente três campos em compare_pairs() e, portanto,
o valor select_threshold() é de no máximo 3 e usaremos aqui
o valor de 2.7, ou seja, 90% de similaridade.
Acompanhe o script a seguir e replique-o em seu
RStudio:
# Criando um novo objeto de linkage `p5` a partir do objeto `sivep_dedup`
p5 <- sivep_dedup |>
# Realizando a blocagem dos dados pela variável "sexo" com uso da
# função pair_blocking(), e realizando o linkage com objeto do_rosas
pair_blocking(do_rosas, blocking_var = "sexo") |>
# Filtrando os pares para deduplicação com a função
# filter_pairs_for_deduplication()
filter_pairs_for_deduplication() |>
# Comparando os pares das colunas selecionadas com a
# função compare_pairs()
compare_pairs(
by = c("nome", "data_nasc", "nome_mae"),
default_comparator = jaro_winkler(0.9)
) |>
# Calculando o escore de similaridade com a função score_simsum()
score_simsum() |>
# Definindo um ponto de corte de 2.7 com a função select_threshold()
select_threshold(2.7) |>
# Adicionando a variável de identificação a partir da coluna x
add_from_x(nu_not = "nu_notific") |>
# Adicionando a variável de identificação a partir da coluna y
add_from_y(nu_do = "nu_do")Atenção
Caso seu computador possua memória RAM inferior a 16 GB você poderá encontrar lentidão no processamento dos scripts deste curso. Sim, pode demorar alguns minutos até que se possa obter o output desejado.
Se possuir outros programas, aplicativos ou abas da internet abertos, feche-os! Assim você consumirá menos memória.
Pronto! Com o objeto {p5} que criamos com a base do SIM
deduplicada, identificaremos agora quantos linkages entres os
dois bancos foram encontrados. Para isso filtramos as variáveis
utilizando a função select() igual a TRUE
.
Acompanhe o script a seguir e replique-o em seu
RStudio:
# Criando objeto com identificação do linkage entre os dois bancos
ident <- p5 |>
# Transformando em um objeto de tabela no formato tibble
as_tibble() |>
# Filtrando as linhas de acordo com a variável select
filter(select)
# Visualizando o número de linhas no objeto ident
nrow(ident)#> [1] 2417Observe que ao utilizar o comando nrow(ident) obtivemos
um resulto indicando que temos 2.417 registros de óbitos no SIVEP-Gripe
de Rosas. Agora vamos fazer uma etapa semelhante à utilizada
anteriormente para saber quantos óbitos estão registradas no SIM de
Rosas.
Acompanhe o script a seguir e replique-o em seu
RStudio:
loc_do <- do_rosas |>
# Filtrando os números da Declaração de Óbito (do) presentes no objeto ident
# com uso da função filter()
filter(nu_do %in% ident$nu_do)
# Visualizando o número de linhas no objeto loc_do
nrow(loc_do)#> [1] 2410Você deve ter percebido que com o dado que manipulamos foi possível descobrir à priori que o SIM contém 2.410 óbitos. Ou seja, encontramos 7 óbitos a mais comparando o SIVEP-Gripe ao SIM. Dessa forma, podemos inferir que estes registros adicionais poderão ser, provavelmente, óbitos que não foram relatados no SIM mas estão no SIVEP-Gripe.
Vamos agora, dar mais um passo e inspecionar a Classificação
Internacional de Doenças (CID-10) contida na variável
CAUSABAS desses registros identificados! Acompanhe o
script a seguir e replique-o em seu RStudio:
# Calculando a tabela de frequência de CIDs com a função table()
table(loc_do$CAUSABAS)#>
#> B342 B972 C159 C679 G20 I10 I251 I619 J129 J189 N390 R99 U071
#> 2390 2 1 1 1 1 1 1 1 4 2 2 3Podemos ver no output acima que mais de 99% dos registros linkados entre o SIVEP-Gripe e o SIM têm como causa básica o CID-10 = B342 (covid-19) e apenas 20 registros possuíam outros CID-10s.
Aqui estamos levando em conta as datas de internação no SIVEP-Gripe e a data do óbito registrada no SIM, o que seria extremamente importante visto que alguns óbitos de pessoas que estiveram internadas podem ter ocorrido por outras causas não relacionadas à internação registrada no SIVEP-Gripe.
Então vamos praticar! Listaremos a seguir os registros que não foram
linkados mas que possuem o CID10 = B342 registrados no SIM como
causa básica. Acompanhe o script a seguir e replique-o em seu
RStudio:
# Criando uma nova tabela com o nome nao_loc
nao_loc <- do_rosas |>
# Filtrando por registros que não foram *linkados e com CID B342 com o uso da
# função filter()
filter(!(nu_do %in% ident$nu_do) & CAUSABAS == 'B342')# Visualizando a tabela criada com o uso da função kable()
kable(nao_loc)Tabela 11: Registros
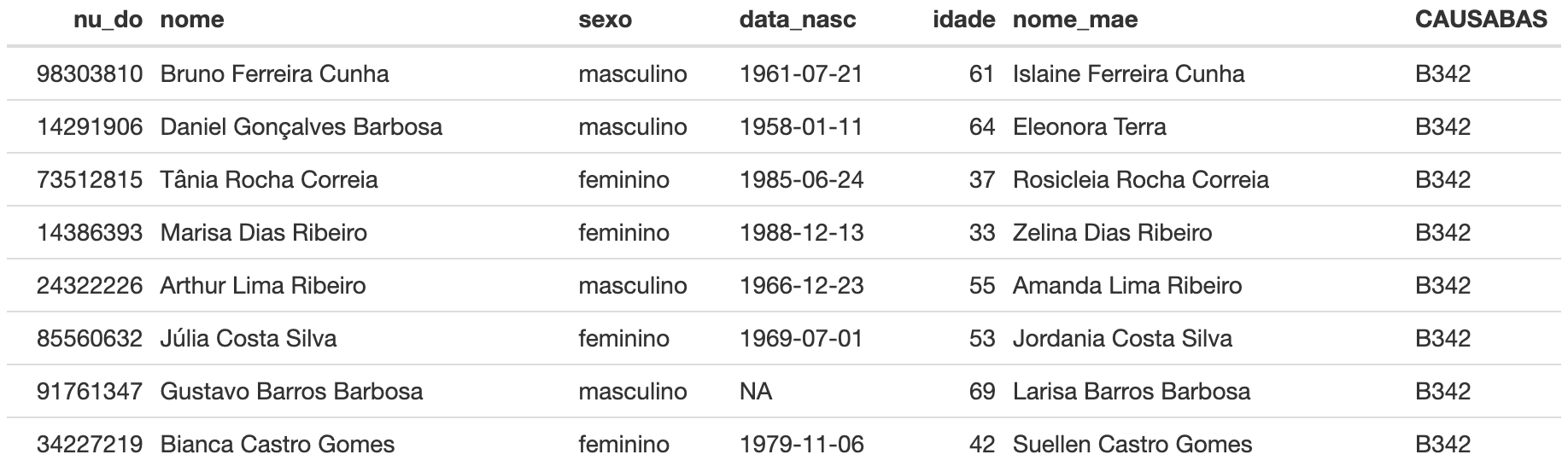
Pronto. Agora precisamos utilizar o nome completo dos registros que ainda não foram localizados para buscá-los no SIVEP-Gripe.
Acompanhe o script a seguir e replique-o em seu
RStudio:
sivep_dedup |>
# Filtrando os nomes presentes na tabela nao_loc com o uso da função filter()
filter(nome %in% nao_loc$nome) |>
# Visualizando a tabela criada com o uso da função kable()
kable()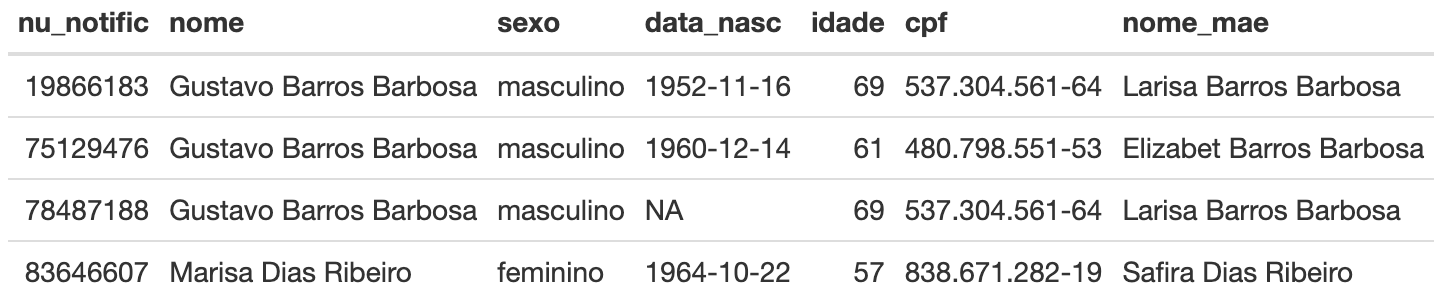
Atenção
Caso seu computador possua memória RAM inferior a 16 GB você poderá encontrar lentidão no processamento dos scripts deste curso. Sim, pode demorar alguns minutos até que se possa obter o output desejado.
Se possuir outros programas, aplicativos ou abas da internet abertos, feche-os! Assim você consumirá menos memória.
Note acima que a pessoa de nome igual a “Gustavo Barros Barbosa”
aparece três vezes em uma lista sendo que em duas vezes trata-se de
duplicidade. Podemos inferir, neste caso, avaliando as variáveis
nome da mãe e CPF, que a terceira menção
trata-se de outra pessoa, ou seja, é apenas um homônimo.
É interessante observar que essa duplicidade não foi detectada na
deduplicação do SIVEP-Gripe, possivelmente porque havia um
NA em um dos registros. Os NA poderiam ser
detectados e tratados antes da deduplicação, por exemplo, e caso sejam
muitos você pode optar por não usar essa informação como chave de
cruzamento entre bancos melhorando a sua acurácia de linkage.
Outra opção é ser mais permissivo com os pontos de coorte do banco de
dados e aplicar uma melhor similaridade na deduplicação
(simsum).
Um linkage nunca é perfeito pode-se inclusive calcular a sensibilidade e especificidade de um pipeline de linkage que vai ser empregado desde que se tenha um padrão para comparação que pode ser uma revisão manual nos dados proveniente do linkage ou ainda um linkage manual de uma amostra. Para maiores detalhes consulte:


