4 Preparando sua base de dados para fazer o linkage
Para o relacionamento de bases de dados são executados um conjunto de processos, tidos como boas práticas, para o estabelecimento de linkage de forma otimizada e com maior acurácia do resultado. Estes passos que foram daptados de podem variar conforme a estratégia de linkage acompanhe a seguir:
Realizar a de padronização dos campos (variáveis) comuns a serem empregados no relacionamento (ex.: quebra do campo nome em seus componentes e a transformação de caracteres para caixa alta). A princípio não se deve remover os missings (NA) pois a não informação vai fazer parte da métrica de similaridade a ser usada.
Deve-se identificar qual a variável a ser usada como blocagem para minimizar o custo com o processamento de dados e a perda de pares verdadeiros.
A escolha de método de linkage, com o uso de cálculo de escores, que sumarizam o grau de concordância global entre registros de um mesmo par.
A definição de limiares para a classificação dos pares de registros relacionados em pares verdadeiros, não pares e pares duvidosos.
A revisão manual dos pares duvidosos visando a classificação dos mesmos como pares verdadeiros ou não pares. Nem sempre é possível se fazer essa etapa entretanto ela pode ajudar a entender e validar especialmente nas primeiras vezes que você vai fazer um linkage.
Adaptado de Coeli & Camargo Jr (https://doi.org/10.1590/S1415-790X2002000200006)
4.1 Deduplicando da base de dados
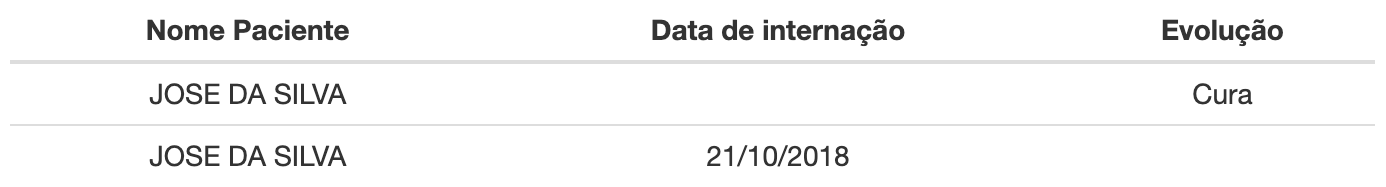
O primeiro passo para iniciar e fazer o linkage é a “deduplicação” das bases de dados. A deduplicação consiste em identificar e remover as duplicidades presentes nos dados. Nem sempre este é um procedimento simples que possa ser automatizado.
Nesta etapa é importante que você reconheça onde estão os melhores registros (de maior qualidade) das bases para o cruzamento. Em alguns registros, parte das informações relevantes pode estar em cada uma das duplicatas. Por exemplo, em relação a um mesmo paciente, a data de internação pode estar presente em um dos registros, enquanto a informação sobre a evolução do caso está gravada em um segundo registro. Em casos como os apresentados será necessária uma verificação manual para consolidar e reconstituir um registro único.
Tabela 7: Exemplos de tratamento de deduplicação.
O R possui diversos pacotes para fazer linkage
de registros. Destacamos alguns que podem ser explorados.
Neste curso nossa escolha recaiu sobre o pacote reclin
por ser mais simples que a versão mais nova (reclin2) e
permitir demostrar os principais conceitos de linkage de bases
de dados.
Outros pacotes que podem ser considerados são:
RecordLinkage: Record Linkage Functions for Linking and Deduplicating Data Setsreclin2: Record Linkage Toolkit (versão mais nova mas não idêntica)diyar: Record Linkage and Epidemiological Case Definitions inRBRL: Beta Record Linkage methodologyfastLink: Fast Probabilistic Record Linkage with Missing DataPPRL: Privacy Preserving Record Linkage
Saiba que o termo linkage se refere a várias técnicas diferentes seja na área de estatística, genética ou teoria de grafos.
Agora, vamos praticar! Abra o seu RStudio para
realizarmos algumas análises.
Acompanhe o script a seguir para instalar e carregar os pacotes tidyverse, reclin e outros. Eles serão utilizados para as práticas neste curso.
if(!require(tidyverse)) install.packages("tidyverse");library(tidyverse)
if(!require(reclin)) install.packages("reclin");library(reclin)
if(!require(digest)) install.packages("digest");library(digest)
if(!require(knitr)) install.packages("knitr");library(knitr)
if(!require(DT)) install.packages("DT");library(DT)Pronto. Agora precisamos importar a base de dados que será utilizada
durante o curso. Acesse o menu lateral “Arquivos” do curso e faça
download do banco de dados de nome
sivep_identificado.csv e a de nome
DO_ROSAS.csv. Lembre-se de manter todas as bases de dados
deste curso em uma mesma pasta ou diretório!
Para nosso exemplo, iremos realizar um linkage com dados de dados exportadas do Sistema de Informação de Mortalidade (SIM) e do Sistema de Informação da Vigilância Epidemiológica da Gripe (SIVEP-Gripe) do Estado de Rosas (ambos fictícios). Vamos lá!
Imagine que a imprensa divulgou alguns óbitos por covid-19 diferentes dos que você tem acompanhado no SIM. A imprensa de Rosas refere que o estado está superestimando os óbitos, sua chefia imediata solicita então que seja feita um cruzamento entre as base de dados de internados e de óbitos para avaliar esse rumor.
Assim, você enquanto profissional de vigilância em saúde deverá realizar um linkage de bancos de dados e encontrar se existe diferenças de registros entre os sistemas e comunicar o resultado de forma imediata para a correção dos dados e sua publicação, caso couber.
Para esta avaliação, iniciaremos importando a base de dados do
SIVEP-Gripe. Acompanhe o script a seguir e replique-o em seu
RStudio:
# Importando dados do SIVEP-Gripe
sivep <- read_csv2("Dados/sivep_identificado.csv")Observe que objeto {sivep} possui 10196 registros
contendo as seguintes variáveis (colunas) que analisaremos para realizar
a nossa avaliação:
nu_notific,nome,sexo,data_nasc,idade,cpf, enome_mae.
Para conhecer melhor esse banco, vamos visualizar os 100 primeiros
registros da tabela {sivep}, conforme o script a
seguir. Copie-o em sue RStudio.
DT::datatable(head(sivep, 100))Tabela 8: Base de dados fictícia do SIVEP.
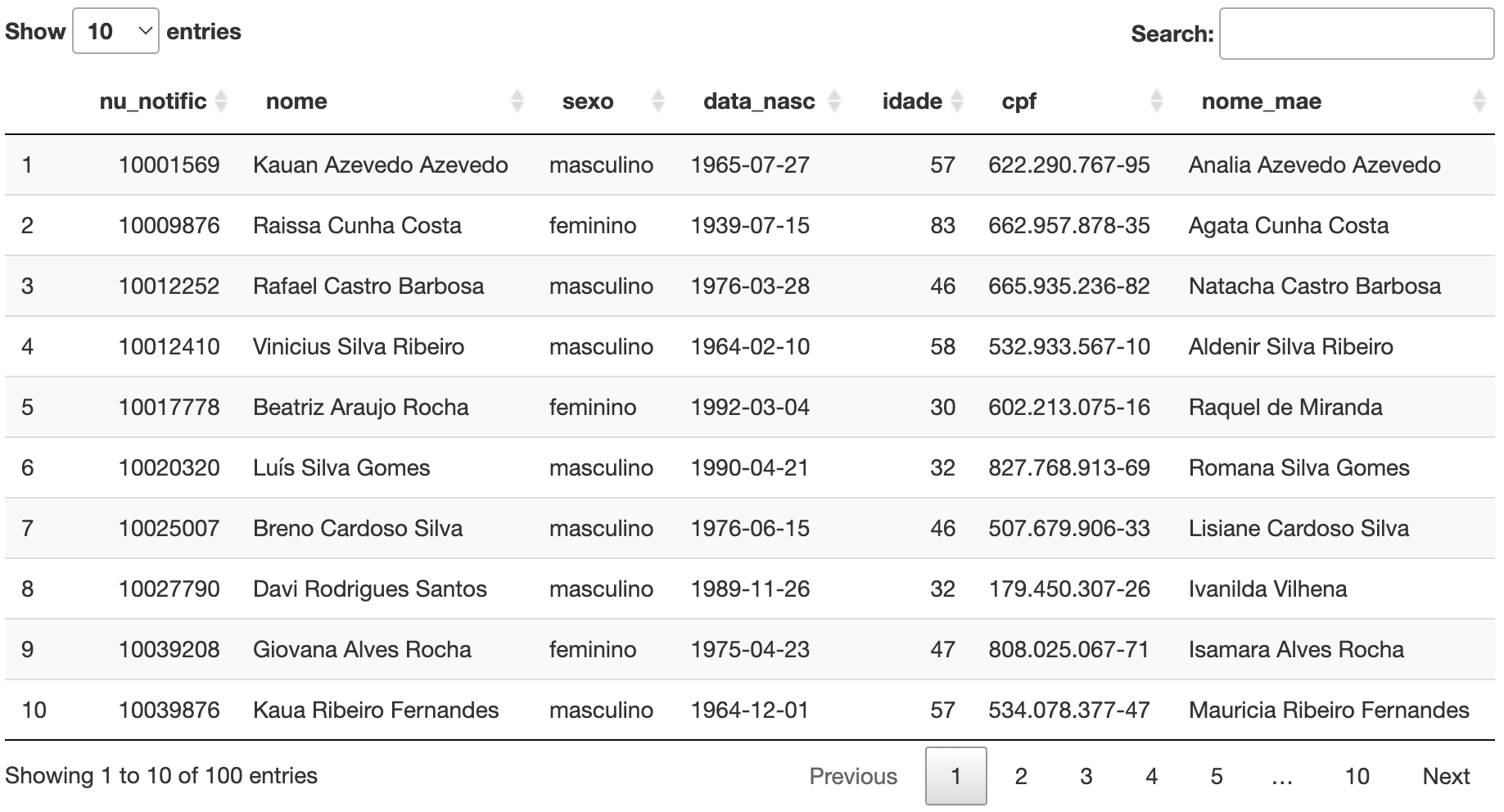
Observe que há duplicações de registros, por vezes idênticos, mas por
vezes com campos diferentes tais como nome,
nome da mãe, CPF e
data de nascimento. Nessa primeira etapa vamos usar o
algoritmo de deduplicação para achar essas duplicidades.
Atenção
Todos os bancos de dados utilizados para análises neste curso se
encontram no menu lateral “Arquivos” do curso. Lembre-se de fazer o
download do material do curso diretamente do Ambiente Virtual
de Aprendizagem do curso e arquivá-los em um diretório que você deverá
indicar para que o R os localize.
4.2 Gerando e filtrando os pares
Vamos um pouco mais adiante em nosso linkage. Em primeiro lugar, precisamos especificar que estamos deduplicando, ou seja, comparando o banco de dados do SIVEP-Gripe com ele mesmo. Dessa forma vamos identificar se há registros duplicados nessa base e resolver a duplicidade, caso seja necessário.
Para isso, utilizaremos o método de blocagem. Precisaremos, então,
especificar a variável que usaremos como blocagem. Nesse caso será a
variável sexo.
Apesar da pouca redução de dimensão que esta variável oferece, ela é bem confiável pois possui poucos erros e proporciona uma redução do número de pares considerável: indo de pouco menos de 52 milhões de pares para aproximadamente 26 milhões.
No código a seguir utilizamos a função pair_blocking()
do pacote reclin e especificamos a blocagem por
sexo. Já a função
filter_pairs_for_deduplication() reduzirá o número de pares
a serem comparados levando em conta a blocagem e o fato de que você está
comparando o banco contra ele mesmo. Vamos lá!
Primeiro, armazenaremos no objeto {pares_blocagem} todos
os pares a serem comparados.
Acompanhe o script a seguir e replique-o em seu
RStudio:
# Realizando a blocagem dos dados pela variável "sexo" com uso da função
# pair_blocking()
pares_blocagem <- pair_blocking(x = sivep, y = sivep, blocking_var = c("sexo")) |>
# Filtrando os pares duplicados com a função filter_pairs_for_deduplication()
filter_pairs_for_deduplication()
# Visualizando a tabela resultante
pares_blocagem#> Simple blocking
#> Blocking variable(s): sexo
#> First data set: 10 196 records
#> Second data set: 10 196 records
#> Total number of pairs: 25 985 290 pairs
#>
#> ldat with 25 985 290 rows and 2 columns
#> x y
#> 1 1 3
#> 2 1 4
#> 3 1 6
#> 4 1 7
#> 5 1 8
#> 6 1 10
#> 7 1 11
#> 8 1 12
#> 9 1 13
#> 10 1 16
#> : : :
#> 25985281 10192 10193
#> 25985282 10192 10194
#> 25985283 10192 10195
#> 25985284 10192 10196
#> 25985285 10193 10194
#> 25985286 10193 10195
#> 25985287 10193 10196
#> 25985288 10194 10195
#> 25985289 10194 10196
#> 25985290 10195 10196Observe que ao visualizarmos o objeto {pares_blocagem} o
R apresentará no Painel Console algumas informações sobre o
processo de blocagem:
- Simple blocking: indica que se trata de um método simples de blocagem, isto é, quando os nomes das variáveis utilizadas são iguais entre si.
- First data set e Second data set: número de registros em cada banco de dados. Como neste exemplo comparamos a mesma base consigo própria, temos 10.196 registros.
- Total number of pairs: indica o total de pares encontrados. Neste exemplo, foram encontrados quase 26 milhões de pares compatíveis. Isto representa perto de 25% do total de pares que seriam comparados caso todos os mais de 10 mil registros fossem comparados um a um.
Após estas informações, o R retorna uma amostra da
tabela listando as linhas que foram pareadas. Este objeto será utilizado
nos próximos passos do linkage. Vamos adiante.
4.3 Aplicando o método de linkage determinístico
Agora aplicaremos o linkage do tipo determinístico. Isto porque com ele o pareamento dos indivíduos no banco de dados é feito pela correspondência exata entre registros. Vamos lá!
Como primeira etapa, iremos realizar o linkage informando quais variáveis queremos comparar. Em nosso exemplo, iremos utilizar as variáveis
nome,data de nascimento,cpfenome da mãe.Então precisaremos comparar se os valores das variáveis são iguais. Ao aplicar esta etapa poderemos obter os valores TRUE (verdadeiro) ou FALSE (falso). Para que você possa compreender melhor, se um par de registros no qual as quatro variáveis escolhidas fossem TRUE (verdadeiras) representaria um linkage com alta acurácia entre estes registros.
Observe o script a seguir com as duas etapas descritas
anteriormente, e realize esta comparação em seu computador. Replique o
código em seu RStudio:
# Criando objeto com o nome p_deter com resultados do linkage determinístico
p_deter <- pares_blocagem|>
# Comparando os pares pelas variáveis de nome, data de nascimento, cpf e
# nome da mãe para realização do linkage determinístico
compare_pairs(by = c("nome", "data_nasc", "cpf", "nome_mae"))Perceba que o objeto {p_deter} armazenou as informações
de quais registros foram comparados de acordo com a blocagem anterior e
quais colunas de cada um desses pares apresentam correspondência
completa de acordo com o critério de linkage determinístico.
A partir dos objetos criados até aqui, poderemos realizar algumas
análises extraindo informações sobre o linkage. Agora,
verificaremos quantos e quais são as correspondências (matchs)
perfeitas que conseguimos realizar na etapa anterior e armazenados no
objeto {p_deter}. Em seu RStudio, rode as
seguintes linhas de código:
# Criando objeto com correspondências perfeitas
pares_iguais <- p_deter |>
# Transformando em um objeto de tabela no formato tibble
as_tibble() |>
# Filtrando apenas os registros em que todas as correspondências são iguais
filter(nome & data_nasc & cpf & nome_mae)
# Visualizando os registros em que todas as correspondências são iguais
pares_iguais#> # A tibble: 16 × 6
#> x y nome data_nasc cpf nome_mae
#> <dbl> <int> <lgl> <lgl> <lgl> <lgl>
#> 1 528 3372 TRUE TRUE TRUE TRUE
#> 2 933 6984 TRUE TRUE TRUE TRUE
#> 3 978 3026 TRUE TRUE TRUE TRUE
#> 4 1023 9323 TRUE TRUE TRUE TRUE
#> 5 1829 1830 TRUE TRUE TRUE TRUE
#> 6 2129 3740 TRUE TRUE TRUE TRUE
#> 7 2223 5120 TRUE TRUE TRUE TRUE
#> 8 2550 6312 TRUE TRUE TRUE TRUE
#> 9 3546 8218 TRUE TRUE TRUE TRUE
#> 10 4382 5132 TRUE TRUE TRUE TRUE
#> 11 5021 6366 TRUE TRUE TRUE TRUE
#> 12 5344 7502 TRUE TRUE TRUE TRUE
#> 13 5953 6263 TRUE TRUE TRUE TRUE
#> 14 6057 6231 TRUE TRUE TRUE TRUE
#> 15 6787 9039 TRUE TRUE TRUE TRUE
#> 16 7769 9719 TRUE TRUE TRUE TRUEObserve que, neste caso, temos 16 registros idênticos. Note que o
valor para o primeiro registo de x[1] (528) e y[1]
(3372) indicam a linha que esses registros duplicados aparecem na base
de dados {sivep}. Assim, a tabela resultante
{pares_iguais} apresenta todos os pares idênticos para as
variáveis que escolhemos.
Agora vamos visualizar quais são esses registros. Para isso, podemos
utilizar a função slice(). Acompanhe o código a seguir e
replique-o em seu RStudio:
sivep |>
# Selecionando no objeto SIVEP-Gripe a primeira linha dos registros duplicados
# no objeto `iguais` com a função slice()
slice(pares_iguais$x[1], pares_iguais$y[1]) |>
# Visualizando a tabela com a função kable()
kable()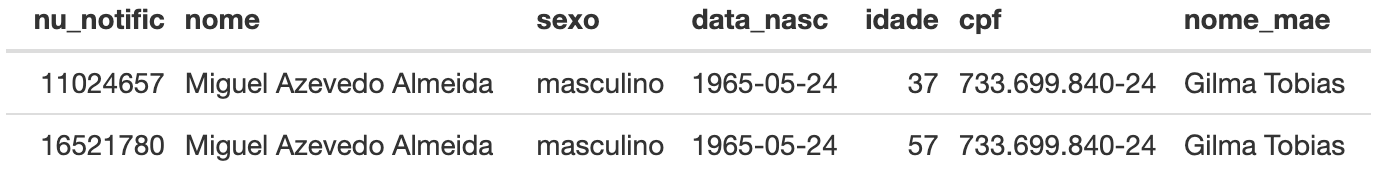
sivep |>
# Selecionando no objeto SIVEP-Gripe a nona linha dos registros duplicados
# no objeto `iguais` com a função slice()
slice(pares_iguais$x[9],pares_iguais$y[9]) |>
# Visualizando a tabela com a função kable()
kable()
Conseguiu visualizar?
Com o resultado obtido seria possível nesse ponto simplesmente apagar do SIVEP-Gripe do Estado de Rosas todas as linhas correspondentes aos valores em y visto que são idênticos ao menos para essas quatro variáveis (atributos).
Considere que este é o banco de dados do SIVEP-Gripe e que ele pode nos informar sobre as reinfecções, reinternação, transferências de pacientes para outros hospitais, ou seja, ele pode conter duplicação de pacientes não por erro, mas porque houve necessidade. Em uma rotina de vigilância é preciso sempre analisar com cuidado esses casos de duplicata e decidir como tratá-los a partir das regras do sistema de informação.
Nesse exercício, tomaremos a decisão de manter todos os registros para a fase seguinte onde faremos um linkage do tipo probabilístico.
Atenção
Lembre-se que o R trabalha com os dados em memória, e
como vimos, esses objetos oriundos do linkage são em geral
grandes. Por exemplo, o objeto {p_deter} tem em
aproximadamente 6.6 MB. Assim, quando não se vai usar mais um objeto
convém ir apagando-o.
Outra recomendação nesse sentido é que se use a função
gc() (Garbage Collection), que como o nome indica
otimiza a memória após o uso de objetos maiores.
Acompanhe o script a seguir e replique-o em seu
RStudio:
# Removendo o objeto `p_deter`
rm(p_deter)
# Otimizando o uso de memória na sessão de R
gc()#> used (Mb) gc trigger (Mb) max used (Mb)
#> Ncells 1359632 72.7 2197014 117.4 2197014 117.4
#> Vcells 2514418 19.2 141220573 1077.5 171904409 1311.6Você pode ter encontrado um resultado diferente do que apresentamos aqui no curso em seu computador. Não se preocupe! O resultado apresentado em seu output pode ser divergente do visualizado aqui porque a quantidade de memória RAM existente em cada computador, que processa o mesmo código aqui escrito, determinará os valores exibidos. Assim, mesmo se os valores exibidos sejam diferentes o processo é o mesmo e os resultados semelhantes.
4.4 Aplicando o método de linkage probabilístico
Agora, iremos realizar um linkage probabilístico. Para isso,
utilizaremos todas as etapas armazenadas no objeto
{pares_blocagem}, criado anteriormente, e aplicaremos a ele
novamente a função compare_pairs().
Dessa vez vamos especificar o nome das variáveis a serem comparadas
adicionando o argumento by. Também vamos especificar o
método para calcular a similaridade de cada uma das variáveis!
Neste exemplo, vamos usar a distância de jaro winkler (semelhante à distância de Levenshtein, só que mais sensível à detecção de similaridades). Para saber mais clique aqui e acesse a Wikipedia.
Para usarmos jaro winkler, vamos especificar no argumento
default_comparator da função compare_pairs()
uma outra função chamada jaro_winkler(), do pacote
reclin. Essa função especifica o ponto de corte de 0,9 para
o nível de similaridade que aceitaremos (o padrão é 0,95).
Acompanhe o script a seguir e replique-o em seu
RStudio:
# Criando o objeto 'pares_link_prob' com o resultado do linkage probabilístico
pares_link_prob <- pares_blocagem |>
# Realizando o linkage probabilístico com a função compare_pairs()
compare_pairs(by = c("nome", "data_nasc", "cpf", "nome_mae"),
default_comparator = jaro_winkler(threshold = 0.9))
# Visualizando o objeto com resultado do linkage
pares_link_prob#> Compare
#> By: nome, data_nasc, cpf, nome_mae
#>
#> Simple blocking
#> Blocking variable(s): sexo
#> First data set: 10 196 records
#> Second data set: 10 196 records
#> Total number of pairs: 25 985 290 pairs
#>
#> ldat with 25 985 290 rows and 6 columns
#> x y nome data_nasc cpf nome_mae
#> 1 1 3 0.5079365 0.7833333 0.5714286 0.5151515
#> 2 1 4 0.4148629 0.7523810 0.6517857 0.5667388
#> 3 1 6 0.4474206 0.6777778 0.6761905 0.5200337
#> 4 1 7 0.5414926 0.7047619 0.6428571 0.5685426
#> 5 1 8 0.5264550 0.6666667 0.5714286 0.5275673
#> 6 1 10 0.6139971 0.7523810 0.5634921 0.5463869
#> 7 1 11 0.4076479 0.6666667 0.6517857 0.5642602
#> 8 1 12 0.5125985 0.7333333 0.5476190 0.5015152
#> 9 1 13 0.5507937 0.6500000 0.5476190 0.4632035
#> 10 1 16 0.5414926 0.7285714 0.5476190 0.5012626
#> : : : : : : :
#> 25985281 10192 10193 0.5420635 0.6777778 0.6137566 0.5236842
#> 25985282 10192 10194 0.5105820 0.6777778 0.5476190 0.5087719
#> 25985283 10192 10195 0.4682540 0.7333333 0.6137566 0.5397233
#> 25985284 10192 10196 0.5006901 0.6222222 0.5000000 0.4737037
#> 25985285 10193 10194 0.6749158 0.6777778 0.5238095 0.5980861
#> 25985286 10193 10195 0.5150794 0.8250000 0.6137566 0.5036995
#> 25985287 10193 10196 0.5544686 0.6777778 0.4801587 0.5649708
#> 25985288 10194 10195 0.5105820 0.6666667 0.5000000 0.5959632
#> 25985289 10194 10196 0.6883483 0.8041667 0.6507937 0.5788596
#> 25985290 10195 10196 0.6255242 0.6500000 0.5000000 0.6038509Atenção
Caso seu computador possua memória RAM inferior a 16 GB você poderá encontrar lentidão no processamento dos scripts deste curso. Sim, pode demorar alguns minutos até que se possa obter o output desejado.
Se possuir outros programas, aplicativos ou abas da internet abertos, feche-os! Assim você consumirá menos memória.
O objeto que criamos {pares_link_prob} irá guardar as
informações dos registros que apresentaram as correspondências de acordo
com os critérios de probabilidade utilizados na função
compare_pairs(). Perceba que ele é muito semelhante ao que
obtivemos com {p_deter}, porém, ao invés de retornar
TRUE ou FALSE, ele retornou um escore
de similaridade que varia de 0 a 1
para cada uma das variáveis em consideração.
Usando esses escores de similaridade para cada variável podemos
construir uma nova variável que, por padrão (default), é
chamada simsum e contém a soma dos escores de similaridade.
Essa soma é criada pela função score_simsum() do pacote
reclin.
Tendo calculado a variável simsum vamos usar agora a
função select_threshold(), também do pacote
reclin, para selecionar os pares a partir de um ponto de
corte. O valor do ponto de corte é a fração de similaridade onde você
deseja selecionar. No nosso exemplo, utilizamos apenas quatro variáveis
e, por isso, o valor máximo do nosso escore de similaridade é
4. Dessa forma, você deve especificar uma fração que represente a
similaridade desejada.
Para resolver a nossa avaliação comparando os registros de óbitos do
SIM e SIVEP-Gripe vamos utilizar o valor de 3,55, que corresponde a um
corte de similaridade 0,875 (3,5 / 4). Ou seja, de uma maneira geral,
bons resultados são atingidos com valores próximos a 0,90 (o que no
nosso exercício seria 3,6/4). Como próximo passo, em seguida, vamos
executar o objeto criado p3.
Acompanhe o script a seguir e replique-o em seu
RStudio:
p3 <- pares_link_prob |>
# Calculando o escore de similaridade com a função score_simsum()
score_simsum() |>
# Definindo um ponto de corte de 3.5 com a função select_threshold()
select_threshold(threshold = 3.5)
# Visualizando o objeto com resultado do linkage
p3#> Compare
#> By: nome, data_nasc, cpf, nome_mae
#>
#> Simple blocking
#> Blocking variable(s): sexo
#> First data set: 10 196 records
#> Second data set: 10 196 records
#> Total number of pairs: 25 985 290 pairs
#>
#> ldat with 25 985 290 rows and 8 columns
#> x y nome data_nasc cpf nome_mae simsum select
#> 1 1 3 0.5079365 0.7833333 0.5714286 0.5151515 2.377850 FALSE
#> 2 1 4 0.4148629 0.7523810 0.6517857 0.5667388 2.385768 FALSE
#> 3 1 6 0.4474206 0.6777778 0.6761905 0.5200337 2.321423 FALSE
#> 4 1 7 0.5414926 0.7047619 0.6428571 0.5685426 2.457654 FALSE
#> 5 1 8 0.5264550 0.6666667 0.5714286 0.5275673 2.292118 FALSE
#> 6 1 10 0.6139971 0.7523810 0.5634921 0.5463869 2.476257 FALSE
#> 7 1 11 0.4076479 0.6666667 0.6517857 0.5642602 2.290361 FALSE
#> 8 1 12 0.5125985 0.7333333 0.5476190 0.5015152 2.295066 FALSE
#> 9 1 13 0.5507937 0.6500000 0.5476190 0.4632035 2.211616 FALSE
#> 10 1 16 0.5414926 0.7285714 0.5476190 0.5012626 2.318946 FALSE
#> : : : : : : : : :
#> 25985281 10192 10193 0.5420635 0.6777778 0.6137566 0.5236842 2.357282 FALSE
#> 25985282 10192 10194 0.5105820 0.6777778 0.5476190 0.5087719 2.244751 FALSE
#> 25985283 10192 10195 0.4682540 0.7333333 0.6137566 0.5397233 2.355067 FALSE
#> 25985284 10192 10196 0.5006901 0.6222222 0.5000000 0.4737037 2.096616 FALSE
#> 25985285 10193 10194 0.6749158 0.6777778 0.5238095 0.5980861 2.474589 FALSE
#> 25985286 10193 10195 0.5150794 0.8250000 0.6137566 0.5036995 2.457535 FALSE
#> 25985287 10193 10196 0.5544686 0.6777778 0.4801587 0.5649708 2.277376 FALSE
#> 25985288 10194 10195 0.5105820 0.6666667 0.5000000 0.5959632 2.273212 FALSE
#> 25985289 10194 10196 0.6883483 0.8041667 0.6507937 0.5788596 2.722168 FALSE
#> 25985290 10195 10196 0.6255242 0.6500000 0.5000000 0.6038509 2.379375 FALSECom o objeto criado {p3} temos os pares devidamente
classificados e podemos agora calcular quantas possíveis duplicidades
foram identificadas. Faremos isso calculando a frequência do valores
identificados pela variável select como TRUE.
Para isso, basta utilizarmos a função count() do pacote
dplyr. Acompanhe o script a seguir e replique-o em
seu RStudio:
p3 |>
# Convertendo em um objeto de tabela no formato tibble
as_tibble() |>
# Contando os valores da coluna select com a função count()
count(select)#> # A tibble: 2 × 2
#> select n
#> <lgl> <int>
#> 1 FALSE 25985102
#> 2 TRUE 188Observe que de acordo com os resultados apresentados no objeto
{p3} foram identificados 188 registros como possíveis
duplicidades (valores iguais a TRUE).
Para ilustrar esse exercício, segue a seguir a Tabela 9 mostrando o
impacto dos valores que poderiam ser usados como ponto de corte na
função select_threshold(). Note que se usarmos 3.99, que
equivale a uma similaridade de 99,75%, somente 16 pares são detectados,
os mesmos apontados por nosso linkage determinístico.
Tabela 9: Valores de similaridade (%) e respectivos pares, a partir da escolha de valores de corte.
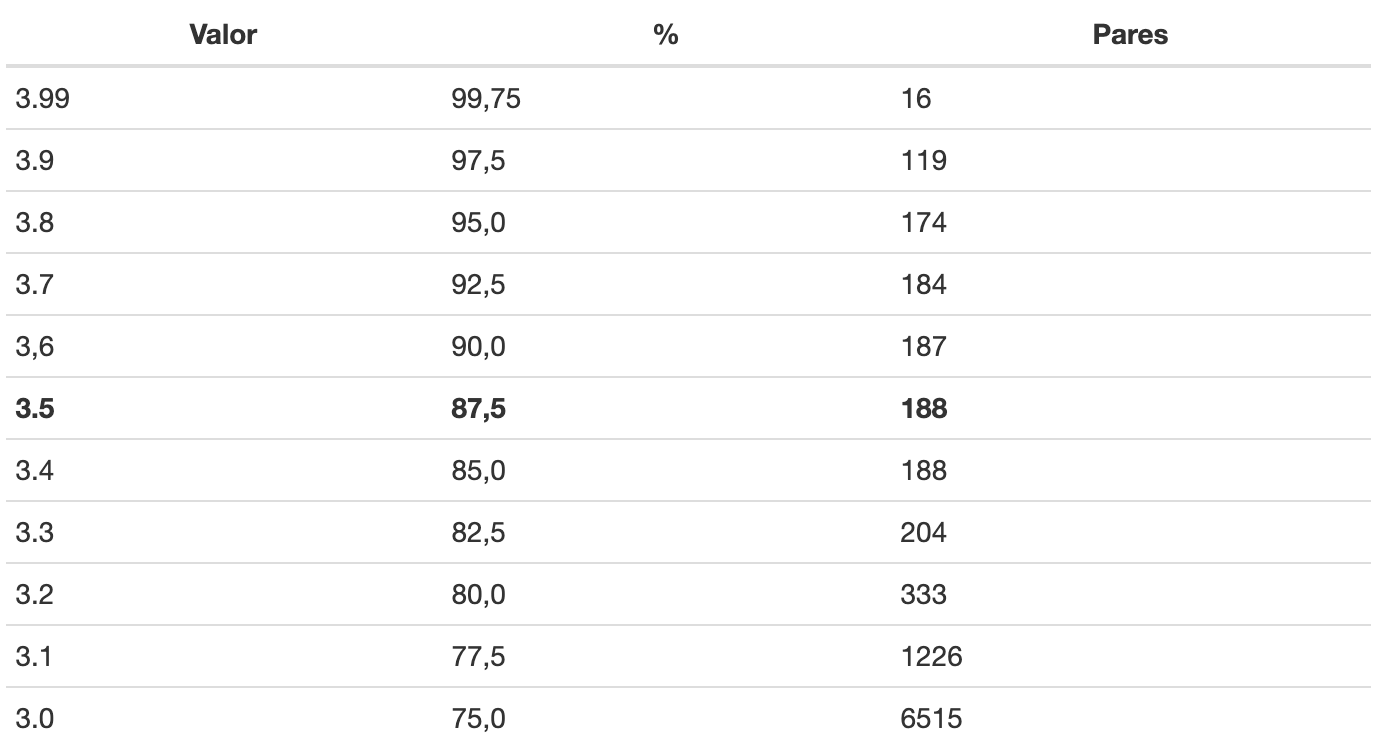
No exemplo que estamos utilizando, temos o controle prévio de que 200 duplicidades foram criadas para nosso exercício. Assim, a posteriori, o valor de 3,3 (82,5%) pode ser mais adequado.
Por sua vez, na prática, esta é uma decisão para minimizar os falsos negativos não deixando escapar possíveis duplicidades e minimizar os falsos positivos que vão consumir energia caso se prossiga a comparação com uma revisão manual. Com esse parâmetro em 3,5, identificamos 94% das duplicidades.
Vamos adicionar agora o número de notificações ao nosso objeto
p3 para identificarmos os pares e adicionar os números.
Acompanhe o script a seguir e replique-o em seu
RStudio:
p3 <- p3 |>
# Adicionando a variável de identificação
add_from_x(nu_not_x = "nu_notific") |>
add_from_y(nu_not_y = "nu_notific")Pronto. Agora vamos listar as variáveis selecionadas ordenadas por
similaridade (simsum) da menor para a maior.
Acompanhe o script a seguir e replique-o em seu
RStudio:
# Criando objeto com o registro de duplicidades denominado reg_dup
reg_dup <- p3 |>
# Transformando em um objeto de tabela no formato tibble
as_tibble() |>
# Filtrando apenas os valores verdadeiros (TRUE) na coluna select com a função
# filter()
filter(select) |>
# Selecionando apenas as colunas de interesse com a função select()
select(x, y, simsum, select, nu_not_x, nu_not_y) |>
# Reordenando as linhas de acordo com a coluna simsum, com uso da função arrange()
arrange(simsum)
# Visualizando o objeto de registro de duplicidades
reg_dup#> # A tibble: 188 × 6
#> x y simsum select nu_not_x nu_not_y
#> <dbl> <int> <dbl> <lgl> <dbl> <dbl>
#> 1 19 2301 3.53 TRUE 10059521 14378854
#> 2 2716 5250 3.63 TRUE 15174637 20001041
#> 3 195 2312 3.67 TRUE 10389866 14399150
#> 4 5074 9982 3.70 TRUE 19625124 95911775
#> 5 1134 5912 3.71 TRUE 12267310 21239686
#> 6 774 3166 3.72 TRUE 11545407 16096758
#> 7 3566 8351 3.73 TRUE 16883275 65485742
#> 8 7384 8581 3.73 TRUE 47417237 69735956
#> 9 2735 6931 3.74 TRUE 15205964 38497474
#> 10 2658 4698 3.76 TRUE 15058156 18878152
#> # … with 178 more rowsPerceba que o objeto {reg_dup} contém os 188 registros
que foram identificados como duplicidades pelo algoritimo de
linkage.
Caso você queira fazer uma revisão manual desses registros, pode-se
obter uma lista de duplicidades. Para isso, utilize a função
deduplicate_equivalence() do pacote reclin,
que inclui uma chave de duplicação para cada registro, ou seja, os
registros duplicados recebem a mesma chave.
Acompanhe o script a seguir e replique-o em seu
RStudio:
# Criando novo objeto com a chave de duplicação de cada registro
res <- deduplicate_equivalence(pairs = p3)Observe em seu RStudio que o objeto {`res``} possui uma
nova coluna contendo o registro das duplicidades encontradas. Nesse
caso, cada conjunto de registros duplicados possui um rótulo (numeração)
diferente.
Acompanhe o script a seguir e replique-o em seu
RStudio:
# Criando um novo objeto com as duplicidades encontradas
dup_grupos <- res |>
# Agrupando os registros de acordo com os códigos da coluna duplicate_groups,
# com uso da função group_by()
group_by(duplicate_groups) |>
# Subagrupando a tabela de valores duplicados e salvando-os como uma lista em
# cada linha
nest() |>
# Criando uma nova coluna denominada pares com o número de duplicatas com o uso
# da função mutate()
mutate(pares = map_dbl(data, nrow))Com o objeto res podemos criar um objeto que agrega os
registros duplicados em uma lista e também criamos uma variável chamada
pares que indica quantos indivíduos foram identificados
naquele grupo.
Nosso interesse são os grupos que têm mais de um indivíduo. Assim,
selecionando esses registros, obtemos uma lista de possíveis
duplicidades. Acompanhe o script a seguir e replique-o em seu
RStudio:
# Criando um novo objeto com a lista de duplicidades
lista_dup <- dup_grupos |>
# Filtrando os pares com mais de um registro
filter(pares > 1) |>
# Desagrupando os valores da lista presentes na "coluna" data
unnest(data) # Visualizando o início da tabela de duplicidades
kable(head(lista_dup))Tabela 10: Tabela de duplicidades.
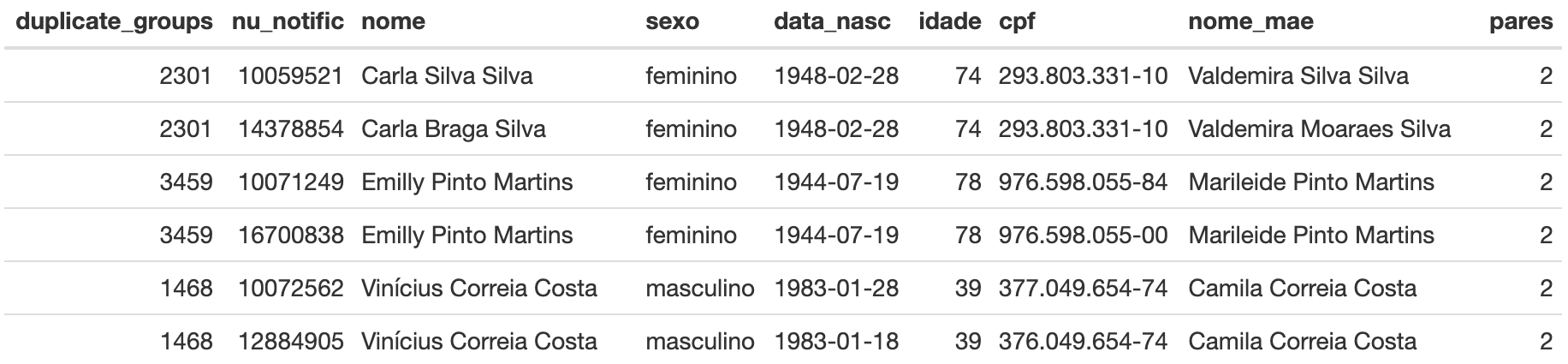
Caso queira, a lista pode ser salva em formato texto ou csv para ser
investigada pela equipe responsável pelo sistema. Para isso, utilize o
script a seguir e replique-o em seu RStudio:
# Salvando a lista de duplicidades em um arquivo .csv
write_csv2(lista_dup, file = 'lista_duplicidades.csv')Para limpar automaticamente as duplicidades identificadas, basta
eliminar os registros duplicados que constam do objeto
reg_dup. Nesse exemplo, vamos remover os registros com
chave em nu_not_y.
Em sua rotina de análises é sempre recomendável que seja feita uma análise mais detalhada que considere a completitude dos dados avaliados, que liste quais as informações mais recentes, e outras eventuais discrepâncias entre registros identificados.
Vamos lá. Acompanhe o script a seguir e replique-o em seu
RStudio:
# Criando um vetor com os registros de duplicidades a partir do objeto reg_dup
registros_remover <- reg_dup$nu_not_y
# Criando um novo objeto sem as duplicidades
sivep_dedup <- sivep |>
# Filtrando os registros com duplicidades indicados no objeto remover com a
# função filter()
filter(!(nu_notific %in% registros_remover ))E, por fim, vamos comparar o número de linhas em cada um e confirmar
que sivep_dedup tem 188 linhas a menos. Acompanhe o script a
seguir e replique-o em seu RStudio:
# Subtraindo o número de linhas no objeto sivep com o número de linhas no objeto
# sivep_dedup
nrow(sivep) - nrow(sivep_dedup)#> [1] 188Fomos apresentando o passo a passo para a deduplicação, mas alguns passos não são necessários (como por exemplo o linkage determinístico). Assim tudo pode ser feito de uma forma mais direta como, por exemplo, descrito no script a seguir:
p3 <- pair_blocking(sivep, sivep,blocking_var ="sexo") |>
filter_pairs_for_deduplication() |>
compare_pairs(c("nome","data_nasc","cpf","nome_mae"),
default_comparator = jaro_winkler(0.9)) |>
score_simsum() |>
select_threshold(3.5) |>
add_from_x( nu_not_x= "nu_notific") |>
add_from_y( nu_not_y = "nu_notific")E para criar uma versão do banco deduplicada (sem nenhuma revisão), replique o código a seguir:
reg_dup <- p3 |>
as_tibble() |>
filter(select ) |>
select(x,y, simsum,select, nu_not_x ,nu_not_y) |>
arrange(simsum)
sivep_dedup <- sivep |>
filter(!(nu_notific %in% reg_dup$nu_not_y ))

