3. Indicadores de Mortalidade
Os indicadores de mortalidade são amplamente utilizados na vigilância em saúde e em diversas outras áreas para além da saúde. O Sistema de Informação de Mortalidade (SIM) é um dos sistemas de informações no SUS focado na melhoria das ações em saúde. É um sistema com boa cobertura, preenchimento e completitude. Algumas de suas principais vantagens são:
- ter seus dados regularmente coletados;
- possuir pontos de coleta, ou seja, capilaridade em todo país;
- ter um padrão de preenchimento nacional, e
- possuir uma longa série histórica de coleta de dados.
Desta forma, existem vários indicadores de mortalidade que integram as análises de saúde. Nesta subseção vamos revisar alguns deles: taxa bruta de mortalidade, taxas específicas de mortalidade por idade e sexo, mortalidade proporcional por grupo de causas, taxa de mortalidade infantil e taxa de mortalidade materna. Todos serão calculados utilizando os dados de óbitos entre 2015 e 2020 do Estado do Acre.
Vamos lá! Comece importando o arquivo {do_ac.dbf} para o
R utilizando a função read.dbf(). Também
importaremos vários arquivos necessários para os cálculos dos
indicadores, são eles:
- Banco de dados {
CID-10-GRUPOS.CSV} que contém variáveis sobre classificação internacional de doenças por grupos de causas (capítulos); - Banco de dados {
pop_ac_09_21.csv} que contém a estimativas da população total do Estado do Acre de 2009 a 2021; - Banco de dados {
pop_ac_sexo_idade_20.csv} que contém a população do Estado do Acre em 2020 estratificada por sexo e faixa etária e; - Banco de dados {
nv_ac_15_20.csv}, com dados de nascidos vivos no Estado do Acre entre 2015 e 2020.
Todos os dados foram importados do repositório do Datasus e estão disponíveis localizado no menu lateral “Arquivos”, do módulo.
# Importando diferentes banco de dados para o R
base_obito_ac <- read.dbf(file = 'Dados/do_ac.dbf')
grupos_causas <- read_csv2(file = 'Dados/CID-10-GRUPOS.csv')
pop_ac <- read_csv2(file = 'Dados/pop_ac_09_21.csv', col_types = list("character"))
pop_ac_sexo_idade <- read_csv2(file = 'Dados/pop_ac_sexo_idade_20.csv')
nv_ac <- read_csv2(file = 'Dados/nv_ac_15_20.csv')Atenção
Fique atento ao caminho escolhido para armazenar os bancos de dados
em seu computador. Caso tenha colocado-as em outro diretório ou pasta,
escreva o caminho deste arquivo para que o R possa
importá-lo para sua análise.
Para os cálculos de indicadores de mortalidade, devido a várias características dos métodos de cálculos, algumas transformações do banco de dados são oportunas e devem serem feitas de imediato. Isso é para facilitar a manipulação sempre que forem realizar alguma análise com bancos de dados. Uma das tarefas mais importante é criar um conjunto de novas variáveis.
Acompanhe a tabela abaixo que apresenta as variáveis que serão criadas e a descrição de cada uma delas, com as alterações que realizaremos para facilitar nossas análises:
Figura 2: Tabela com as alterações realizadas no banco de dados de óbitos.
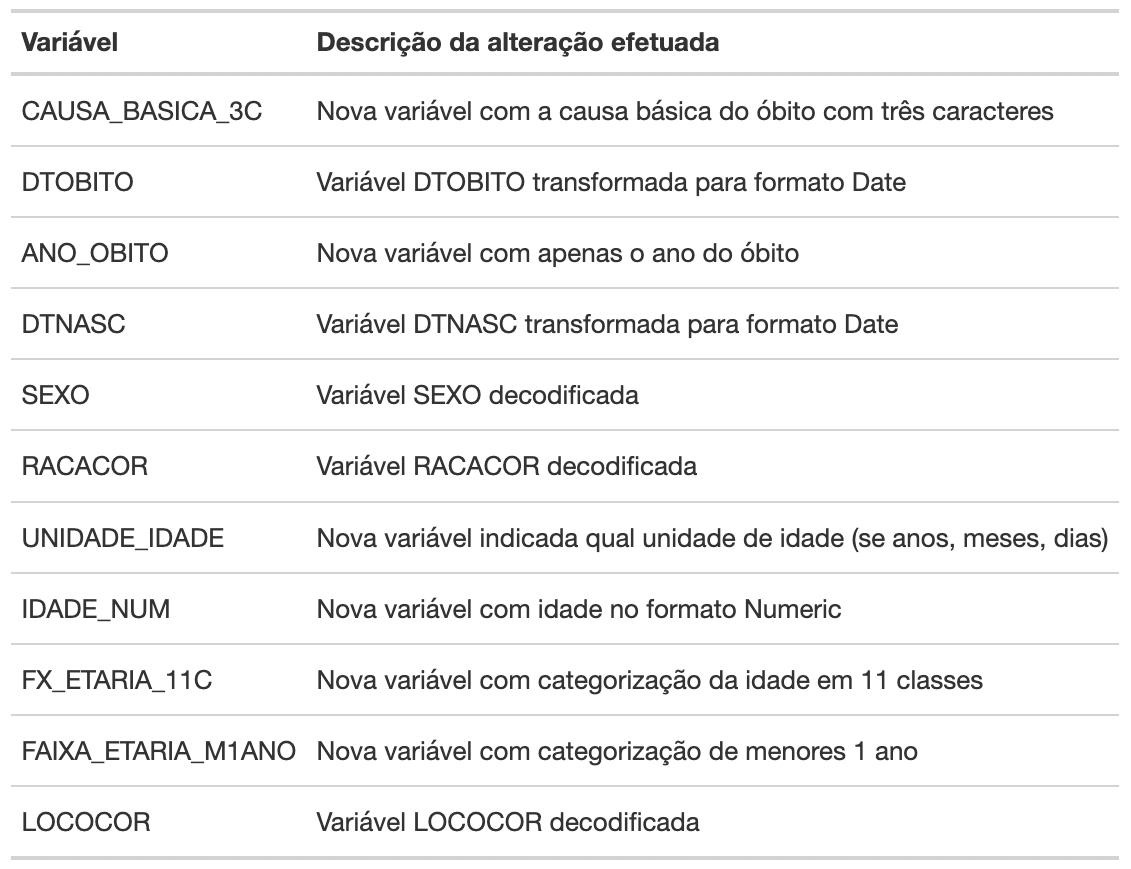
Observe que realizaremos de uma só vez todas essas alterações apontadas, escrevendo diversas linhas de código. Caso sinta-se mais seguro, você poderá realizar as mesmas alterações variável por variável, de forma mais confortável, construindo seu estilo de trabalho. Entretanto, isso implicará em um maior tempo de escrita do seu script.
Agora observe no passo a passo abaixo como colocaremos em prática estas transformações:
- São utilizados no código as funções
mutate(),str_sub(),dmy(),year(),case_when()efactor(). Lembre-se que na funçãocase_when(), quando nenhum dos critérios é atendido, usamosTRUE ~ NA_character_para indicar que estes valores devem ser convertidos emNA. Caso tenha mais alguma dúvida, consulte os módulos anteriores do curso.
Veja o script abaixo com muita atenção, leia os comentários
explicando cada parte e replique-o no seu RStudio:
# Criando uma tabela (dataframe) com o nome base_obito_ac
base_obito_ac <- base_obito_ac |>
# Criando novas colunas com a função mutate()
mutate(
# Criando coluna com extração de apenas os 3 primeiros caracteres
# da coluna CAUSABAS pelo uso da função str_sub()
CAUSA_BASICA_3C = str_sub(CAUSABAS, 1, 3),
# Convertendo as informações na coluna DTOBITO para o formato de
# data (DD-MM-YYYY) com a função dmy()
DTOBITO = dmy(DTOBITO),
# Obtendo apenas o ano de óbito a partir da coluna DTOBITO com a função year()
ANO_OBITO = year(DTOBITO),
# Convertendo as informações na coluna DTNASC para o formato de
# data (DD-MM-YYYY) com a função dmy()
DTNASC = dmy(DTNASC),
# Transformando os códigos de sexo nos nomes correspondentes com a
# função case_when()
SEXO = case_when(
SEXO == "1" ~ "masculino",
SEXO == "2" ~ "feminino",
TRUE ~ NA_character_
),
# Convertendo a coluna SEXO em uma variável do tipo factor
SEXO = factor(SEXO, levels = c("masculino", "feminino")),
# Transformando os códigos de raça/cor nos nomes correspondentes com a
# função case_when()
RACACOR = case_when(
RACACOR == "1" ~ "Branca",
RACACOR == "2" ~ "Preta",
RACACOR == "3" ~ "Amarela",
RACACOR == "4" ~ "Parda",
RACACOR == "5" ~ "Indígena",
TRUE ~ NA_character_
),
# Transformando os códigos de unidade da idade nos nomes correspondentes com a
# função case_when(). A classificação utiliza apenas o primeiro caractere da
# coluna IDADE, com uso da função str_sub()
UNIDADE_IDADE = case_when(
(str_sub(IDADE, 1, 1) == "5") ~ "anos+",
(str_sub(IDADE, 1, 1) == "4") ~ "anos",
(str_sub(IDADE, 1, 1) == "3") ~ "meses",
(str_sub(IDADE, 1, 1) == "2") ~ "dias",
(str_sub(IDADE, 1, 1) == "1") ~ "horas",
(str_sub(IDADE, 1, 1) == "0") ~ "minutos"
),
# Extraindo os dois últimos dígitos da idade com a função str_sub() e
# convertendo para número utilizando a função as.numeric()
IDADE_NUM = as.numeric(str_sub(IDADE, 2, 3)),
# Criando uma classificação de faixa etária por intervalos utilizando
# as variáveis criadas acima
FX_ETARIA_11C = case_when(
(UNIDADE_IDADE == "anos" & between(IDADE_NUM, 0, 4)) ~ "0 a 4",
(UNIDADE_IDADE == "anos" & between(IDADE_NUM, 5, 9)) ~ "5 a 9",
(UNIDADE_IDADE == "anos" & between(IDADE_NUM, 10, 14)) ~ "10 a 14",
(UNIDADE_IDADE == "anos" & between(IDADE_NUM, 15, 19)) ~ "15 a 19",
(UNIDADE_IDADE == "anos" & between(IDADE_NUM, 20, 29)) ~ "20 a 29",
(UNIDADE_IDADE == "anos" & between(IDADE_NUM, 30, 39)) ~ "30 a 39",
(UNIDADE_IDADE == "anos" & between(IDADE_NUM, 40, 49)) ~ "40 a 49",
(UNIDADE_IDADE == "anos" & between(IDADE_NUM, 50, 59)) ~ "50 a 59",
(UNIDADE_IDADE == "anos" & between(IDADE_NUM, 60, 69)) ~ "60 a 69",
(UNIDADE_IDADE == "anos" & between(IDADE_NUM, 70, 79)) ~ "70 a 79",
((UNIDADE_IDADE == "anos" & IDADE_NUM >= 80) | UNIDADE_IDADE == "anos+") ~
"80 e mais",
TRUE ~ NA_character_
),
# Convertendo a coluna FX_ETARIA_11C em uma variável do tipo factor
FX_ETARIA_11C = factor(
FX_ETARIA_11C,
levels = c(
"0 a 4" , "5 a 9" , "10 a 14", "15 a 19", "20 a 29", "30 a 39",
"40 a 49", "50 a 59", "60 a 69", "70 a 79", "80 e mais"
)
),
# Reclassificando variáveis da coluna FX_ETARIA_M1ANO para os casos em que
# a idade seja menor do que 1 ano
FX_ETARIA_M1ANO = case_when(
(UNIDADE_IDADE == "anos" & IDADE_NUM < 1) ~ "< 1 ano",
(UNIDADE_IDADE == "dias") ~ "< 1 ano",
(UNIDADE_IDADE == "meses") ~ "< 1 ano",
(UNIDADE_IDADE == "horas") ~ "< 1 ano",
(UNIDADE_IDADE == "minutos") ~ "< 1 ano",
TRUE ~ NA_character_
),
# Transformando os códigos de local de ocorrência nos nomes correspondentes
# com a função case_when()
LOCOCOR = case_when(
LOCOCOR == "1" ~ "Hospital",
LOCOCOR == "2" ~ "Outros estab saude",
LOCOCOR == "3" ~ "Domicilio",
LOCOCOR == "4" ~ "Via publica",
LOCOCOR == "5" ~ "Outros",
TRUE ~ NA_character_
)
) |>
# Unindo a tabela resultante com a tabela grupos_causas pelas colunas com os
# códigos de causa básica em cada uma
left_join(grupos_causas, by = c("CAUSA_BASICA_3C" = "CAT"))Agora que já preparamos nosso banco de dados de análise, estamos prontos para realizar o cálculo dos indicadores de mortalidade. Vamos em frente!
3.1 Taxa bruta de mortalidade ou taxa geral de mortalidade
Esse indicador representa o número total de óbitos, por mil (1.000) habitantes, na população residente em determinado local e ano considerado. Ele expressa a intensidade que a mortalidade atinge uma população. É um indicador muito usado na demografia, e exige cuidado ao se utilizar para comparações entre populações distintas.

Para a contagem do número total de óbitos, considere agrupar os dados
por ano do óbito. Para isto, utilizaremos a função count()
para contar o número de óbitos em cada ano.
Vamos guardar os dados resultantes desse comando em um objeto novo:
ob_geral_ac_15_20, para que possamos utilizá-lo em
seguida.
Acompanhe o script abaixo e replique em seu
RStudio:
# Criando uma tabela (dataframe) com o nome ob_geral_ac_15_20
ob_geral_ac_15_20 <- base_obito_ac |>
# Contando o número de casos por ano de óbito (ANO_OBITO) com o uso
# da função count()
count(ANO_OBITO)
# Visualizando a tabela resultante
ob_geral_ac_15_20#> ANO_OBITO n
#> 1 2015 3517
#> 2 2016 3763
#> 3 2017 3832
#> 4 2018 4094
#> 5 2019 4098
#> 6 2020 4860A tabela gerada apresentará duas colunas: a ANO_OBITO,
contendo o ano de ocorrência de cada óbito, e a coluna n,
que por sua vez contém o total de óbitos registrados em cada um dos
anos.
Para o cálculo da taxa bruta de mortalidade, siga o passo a passo abaixo:
Utilizando a função
summarise()vamos resumir os dados de população do Acre, somando as estimativas para cada município e ano.Além disso, vamos estruturar a tabela para ficar na mesma estrutura dos dados de mortalidade. Para isso, vamos utilizar a função
pivot_longer()do pacotedplyrpara transformar os dados do formato largo para longo, conforme vimos no Módulo 3. Esta etapa é importante para que criemos as variáveis necessárias para o cálculo da taxa bruta de mortalidade.
Veja o script abaixo e escreva em seu
RStudio:
# Criando uma tabela (dataframe) com o nome pop_ac_15_20
pop_ac_15_20 <- pop_ac |>
# Calculando o número de casos por ano com a função summarise()
summarise(
pop_2015 = sum(`2015`),
pop_2016 = sum(`2016`),
pop_2017 = sum(`2017`),
pop_2018 = sum(`2018`),
pop_2019 = sum(`2019`),
pop_2020 = sum(`2020`)
) |>
# Transformando os dados do formato largo para o formato longo
pivot_longer(cols = pop_2015:pop_2020,
values_to = "pop_ac")
# Visualizando a tabela resultante
pop_ac_15_20#> # A tibble: 6 × 2
#> name pop_ac
#> <chr> <dbl>
#> 1 pop_2015 831665
#> 2 pop_2016 844137
#> 3 pop_2017 856457
#> 4 pop_2018 869265
#> 5 pop_2019 881935
#> 6 pop_2020 894470A tabela resultante irá apresentar em sua primeira coluna
(name) informações sobre o ano, e na segunda coluna
(pop_ac) a população estimada para o Acre para cada ano
correspondente. Observe que os bancos de dados de óbito e de população
possuem a mesma dimensão (2 colunas, 6 linhas) e se referem à mesma
unidade de tempo nas linhas: o ano.
- No terceiro passo utilizaremos a função
bind_cols()do pacotedplyrpara unir as tabelas de óbitos e população para calcular a taxa bruta de mortalidade. Vamos executar juntos os códigos do script abaixo, e replique-o em seuRStudio:
# Unindo as duas tabelas com a função bind_cols()
bind_cols(ob_geral_ac_15_20, pop_ac_15_20)#> ANO_OBITO n name pop_ac
#> 1 2015 3517 pop_2015 831665
#> 2 2016 3763 pop_2016 844137
#> 3 2017 3832 pop_2017 856457
#> 4 2018 4094 pop_2018 869265
#> 5 2019 4098 pop_2019 881935
#> 6 2020 4860 pop_2020 894470Veja como foi simples unir as duas tabelas utilizando a função
bind_cols() !
- Agora podemos criar uma nova coluna contendo a taxa geral de
mortalidade usando a função
mutate(). Vamos lá, acompanhe o script abaixo e replique-o no seu computador:
# Unindo as duas tabelas com a função bind_cols()
bind_cols(ob_geral_ac_15_20, pop_ac_15_20) |>
# Criando uma nova coluna com a taxa geral de mortalidade com a função mutate()
mutate(tx_bruta_ac = (n / pop_ac) * 1000)#> ANO_OBITO n name pop_ac tx_bruta_ac
#> 1 2015 3517 pop_2015 831665 4.228866
#> 2 2016 3763 pop_2016 844137 4.457807
#> 3 2017 3832 pop_2017 856457 4.474247
#> 4 2018 4094 pop_2018 869265 4.709726
#> 5 2019 4098 pop_2019 881935 4.646601
#> 6 2020 4860 pop_2020 894470 5.433385Você deverá encontrar esta tabela acima resultante da união de
tabelas que foram geradas nos passos anteriores. Esta nova tabela possui
a adição de uma coluna nova: a tx_bruta_ac que indica a
taxa bruta de mortalidade para todo o Estado do Acre, nos anos entre
2015 e 2020. Observe no output que a taxa bruta de mortalidade
ou taxa geral de mortalidade do Estado do Acre variou entre 4,23 e 5,43
por mil habitantes, nestes anos.
Atenção
Unir tabelas com a função bind_cols() do pacote
dplyr será possível apenas quando ambas as tabelas
possuírem a mesma dimensão (número de linhas) e lógica de armazenamento
de dados. Nestes casos, os argumentos da função são as tabelas a serem
conectadas!!!
3.2 Taxas específicas de mortalidade por idade e sexo
Também podemos calcular se há diferenças entre sexo e idade por uma mesma doença nos óbitos. Para isto, utilizamos a taxa específica de mortalidade por idade e sexo, calculamos o número de óbitos por uma causa de morte específica e a comparamos por 100 mil habitantes residentes em mesmo local e ano determinado.
Observe abaixo, no script que selecionamos para análise, os óbitos por causas externas no ano de 2020. Lembre-se que permanecemos utilizando o banco de dados de óbitos do Estado do Acre.
Para a construção do script que nos retornará taxas específicas de mortalidade por idade e sexo seguiremos os seguintes passos:
filtraremos os seguintes critérios:
- registros em que o ano do óbito (ANO_OBITO) é igual a 2020;
- registros em que as variáveis faixa etária
(
FX_ETARIA_11C) e sexo (SEXO) não estão em branco (lembre-se de que o operador!tem significado de “NÃO”, e irá inverter os Verdadeiro e Falso da operação lógica); - registros cuja causa de morte pertence ao grupo de causas externas de óbito.
relizaremos a contagem dos óbitos por faixa etária e sexo;
transformaremos a variável
SEXOde linha para coluna, usando a funçãopivot_wider();uniremos a tabela de população por sexo e idade {
pop_ac_sexo_idade}, importada anteriormente;criaremos as variáveis com as taxas usando a função
mutate();por fim, selecionaremos as variáveis necessárias para o cálculo da taxa.
Agora, veja com atenção como ficam todos estes passos no código
abaixo. Replique a escrita deste script em seu
RStudio:
base_obito_ac |>
# Filtrando registros utilizando a função filter()
filter(
#Filtrando os dados por óbitos ocorridos em 2020 com a
ANO_OBITO == 2020,
# Filtrando os dados e removendo aqueles com valores ausentes para a faixa
# etária
!(is.na(FX_ETARIA_11C)),
# Filtrando os dados e removendo aqueles com valores ausentes para sexo
!(is.na(SEXO)),
# Filtrando os dados por óbitos ocorridos por "Causas externas"
GRUPOS == "Causas externas") |>
# Contando o número de casos por faixa etária e sexo com a função count()
count(FX_ETARIA_11C, SEXO) |>
# Transformando a tabela do formato longo para o formato largo com a função
# pivot_wider()
pivot_wider(names_from = SEXO,
values_from = n) |>
# Unindo a tabela resultante com a tabela pop_ac_sexo_idade com a função
# bind_cols()
bind_cols(pop_ac_sexo_idade) |>
# Criando colunas de taxa específica para homens e mulheres com a função mutate()
mutate(
taxa_especifica_homem = masculino / Pop_Masculino * 100000,
taxa_especifica_mulher = feminino / Pop_Feminino * 100000
) |>
# Selecionando apenas as colunas Faixa Etaria 1, taxa_especifica_homem e
# taxa_especifica_mulher com a função select()
select("Faixa Etaria 1",
taxa_especifica_homem,
taxa_especifica_mulher)#> # A tibble: 11 × 3
#> `Faixa Etaria 1` taxa_especifica_homem taxa_especifica_mulher
#> <chr> <dbl> <dbl>
#> 1 0 a 4 anos 11.8 2.47
#> 2 5 a 9 anos 11.5 7.23
#> 3 10 a 14 anos 22.0 6.83
#> 4 15 a 19 anos 179. 35.1
#> 5 20 a 29 anos 177. 22.0
#> 6 30 a 39 anos 172. 18.9
#> 7 40 a 49 anos 121. 16.9
#> 8 50 a 59 anos 110. 14.4
#> 9 60 a 69 anos 94.7 14.3
#> 10 70 a 79 anos 111. 18.1
#> 11 80 anos e mais 169. 39.4Observe no output que, para o ano de 2020 no Estado do Acre, a taxa de mortalidade por causas externas em homens é muito superior à taxa de mortalidade por causas externas em mulheres. A faixa etária mais acometida é a de 15 a 19 anos em homens e 80 anos e mais em mulheres.
3.3 Mortalidade proporcional por grupo de causas
Um importante indicador é o de mortalidade proporcional. Ele
representa a distribuição percentual (%) de óbitos e pode
ser analisada com foco em grupos de causas de óbito. Lembre-se que
sempre deverá analisar uma mesma população residente em um mesmo espaço
geográfico (local) e no tempo (dias, meses ou anos).
Este indicador é importante para medir a carga de cada grupo de causa no total de óbitos da região geográfica analisada. Pode ser calculado matematicamente da seguinte forma:

Para todo cálculo que representar uma proporção pode-se utilizar cálculos simples para a porcentagem, ou seja, multiplicado por 100.
Para o cálculo deste indicador no R, você deverá criar
uma tabela com frequência absoluta e frequência relativa, cuja coluna de
frequência relativa representará o resultado do cálculo da mortalidade
proporcional. Para esta etapa, siga o passo a passo abaixo:
Primeiro, selecionaremos apenas os dados por óbitos ocorridos em 2020 com a função
filter().Depois iremos realizar o cálculo da frequência absoluta de óbitos por grupos com a função
count().E por fim, utilizamos a função
mutate()para calcular a frequência relativa de óbitos para cada grupo.
Agora, observe o script abaixo e replique em seu
RStudio.
base_obito_ac |>
# Filtrando os dados por óbitos ocorridos em 2020 com a função filter()
filter(ANO_OBITO == 2020) |>
# Contando o número de casos por grupos com a função count()
count(GRUPOS) |>
# Calculando o número de mortes proporcionais em cada grupo pelo número
# total com a função mutate()
mutate(mort_proporcional = (n / sum(n)) * 100)#> GRUPOS n mort_proporcional
#> 1 Algumas afecções originadas no período perinatal 125 2.5720165
#> 2 Causas externas 596 12.2633745
#> 3 Causas Mal Definidas 662 13.6213992
#> 4 Causas Maternas 5 0.1028807
#> 5 Doenças do aparelho circulatório 771 15.8641975
#> 6 Doenças do aparelho respiratório 488 10.0411523
#> 7 Doenças infecciosas e parasitárias 1006 20.6995885
#> 8 Neoplasias 539 11.0905350
#> 9 Outros grupos 668 13.7448560Assim, ao observar o output do código executado podemos concluir que o maior grupo de causa de morte no Acre é o de Doenças infecciosas e parasitárias, com 20,69% de todas as mortes ocorridas no Estado. Essa informação é muito importante para a vigilância, pois o serviço precisa conhecer as principais causas de mortalidade e desencadear ações de saúde e intersetoriais.
3.4 Taxa de mortalidade infantil
A taxa de mortalidade infantil representa o número de óbitos ocorridos em crianças menores de um ano de idade por mil nascidos vivos no mesmo período. Este indicador é muito importante por permitir avaliar as condições de vida e de saúde de uma dada população. Com o cálculo da sua taxa, é possível estimar o risco de uma criança morrer antes de chegar a um ano de vida, sendo consideradas como altas (50 ou mais), médias (20-49) e baixas (menos de 20).
As taxas de mortalidade infantil, em especial a mortalidade pós-neonatal, quando elevadas refletem as precárias condições de vida e saúde e valores abaixo do nível de desenvolvimento social e econômico. Já taxas reduzidas podem sinalizar bons indicadores sanitários e sociais, mas também podem encobrir más condições de vida em segmentos sociais específicos.
No caso do cálculo da mortalidade infantil e de seus componentes, o denominador é o número de nascidos vivos de mães residentes do local. Observe como é possível calcular este indicador matematicamente pela seguinte expressão:

Agora, vamos escrever o script que corresponderá à fórmula
matemática e que permite que seu cálculo possa ser realizado de maneira
automatizada. Observe abaixo e replique no seu RStudio:
base_obito_ac |>
# Filtrando os dados por registros de casos com menos de 1 ano com a função
# filter()
filter(FX_ETARIA_M1ANO == "< 1 ano") |>
# Contando o número de casos por ano de óbito com a função count()
count(ANO_OBITO) |>
# Unindo a tabela resultante com a tabela nv_ac com a função bind_cols()
bind_cols(nv_ac) |>
# Calculando a taxa de mortalidade infantil com a função mutate()
mutate(tx_mortalidade_infantil = (n / n_nascidos_vivos) * 1000)#> ANO_OBITO n Ano do nascimento n_nascidos_vivos tx_mortalidade_infantil
#> 1 2015 291 2015 16980 17.13781
#> 2 2016 239 2016 15773 15.15248
#> 3 2017 223 2017 16358 13.63247
#> 4 2018 273 2018 16543 16.50245
#> 5 2019 259 2019 16280 15.90909
#> 6 2020 243 2020 15142 16.04808A taxa de mortalidade infantil por mil nascidos vivos no Acre chegou a diminuir de 17,13 em 2015 para 13,63 em 2017, mas voltou a aumentar e chegou a 16,04 em 2020.
3.5 Taxa de mortalidade materna
A taxa de mortalidade materna é calculada a partir do número de óbitos maternos ocorridos a cada 100 mil nascidos vivos de mães residentes. Esta taxa é definida como a morte durante a gravidez ou no prazo de 42 dias após o final da gestação. A taxa de mortalidade materna é um indicador muito utilizado para entender e comparar um importante problema de saúde pública global: a condições de saúde das mulheres, o desenvolvimento econômico e as desigualdades sociais dos países.
Observe abaixo como é possível calcular a taxa de mortalidade materna matematicamente:

Agora, vamos escrever o script que corresponderá à fórmula
matemática e permitirá que seu cálculo possa ser realizado de maneira
automatizada, replique o código abaixo em seu RStudio:
base_obito_ac |>
# Utilizando a função filter() para diversos critérios de filtragem de dados
filter(
# Filtrando os dados por registros de casos do sexo feminino
SEXO == "feminino",
# Filtrando os registros com casos nas faixas etárias entre 10 e 49 anos
FX_ETARIA_11C %in% c("10 a 14", "15 a 19",
"20 a 29", "30 a 39",
"40 a 49"),
# Filtrando os registros de óbitos por causas maternas
GRUPOS == "Causas Maternas"
) |>
# Contando o número de casos por ano de óbito com a função count()
count(ANO_OBITO) |>
# Unindo a tabela resultante com a tabela nv_ac com a função bind_cols()
bind_cols(nv_ac) |>
# Calculando a taxa de mortalidade materna com a função mutate()
mutate(razao_mort_materna = (n / n_nascidos_vivos) * 100000)#> ANO_OBITO n Ano do nascimento n_nascidos_vivos razao_mort_materna
#> 1 2015 11 2015 16980 64.78210
#> 2 2016 9 2016 15773 57.05953
#> 3 2017 8 2017 16358 48.90573
#> 4 2018 9 2018 16543 54.40368
#> 5 2019 8 2019 16280 49.14005
#> 6 2020 5 2020 15142 33.02074Observe que o nosso output informa para cada ano o total de
óbitos maternos (coluna n), o número de nascidos vivos
(coluna n_nascidos_vivos), e o valor da razão de
mortalidade materna (coluna razao_mort_materna).
Ao analisarmos a tabela construída, é possível observar melhora na taxa de mortalidade materna ao longo desses 6 anos, o que pode indicar esforços do Estado do Acre para interferir sobre o indicador. Lembre-se dos vieses que influenciam este indicador como a subnotificação do denominador (número de nascidos vivos) e problemas de investigação do óbito materno (número de óbitos por causas de morte materna).
Para tornar sua análise ainda mais aprofundada sugere-se que você análise além da causa específica da morte materna, também a idade da mulher ao morrer, o estado marital, o número de filhos tidos e os ainda vivos, a paridade, o intervalo entre gestações, consultas no pré-natal, hábito de fumar, de beber e outras. Algumas destas variáveis, não são obtidas a partir dos atestados de óbito, necessitando análises do SINASC, e-SUS AB PEC ou outros bancos de dados disponíveis em seu município.
Lembre-se que todo e qualquer indicador de saúde deve prezar pela simplicidade para que as informações sejam transmitidas de forma clara e sem inconsistências.
Caso não tenha compreendido os dados ou valores, retorne ao seu cálculo e refaça. Torne-o simples!
Para consultar mais indicadores de saúde, visite o site das fichas de qualificação de indicadores da Ripsa.
Com base nos códigos até agora descritos você já pode expandir suas análises para diferentes doenças e agravos, locais, anos, faixas etárias, etc. Mas vamos adiante. Há mais possibilidades à frente.


