2. Indicadores de morbidade
Os indicadores de morbidade apoiam o estudo da relação entre o aparecimento de doenças em uma determinada região. A morbidade frequentemente é avaliada a partir de quatro indicadores básicos: a incidência, a prevalência, a taxa de ataque e a distribuição proporcional.
Atenção
Lembre-se que um bom indicador é aquele que:
- possui uma fonte de dados confiável,
- possui um parâmetro para análise comparativa, e
- suas limitações ou vieses são conhecidos.
2.1 Incidência
O uso da incidência é muito frequente na vigilância em saúde, pois expressa o número de casos novos de uma determinada doença durante um período que se deseja estudar em uma população sob o risco de desenvolver a doença, no mesmo período.
Seu cálculo favorece a medição da velocidade da ocorrência de uma doença na população. Quando obtemos esta medida, sua interpretação se dá como risco de adoecimento por um determinado agravo na população. Assim, altos valores de incidências podem ser compreendidos como um alto risco coletivo de adoecimento.
Para esse indicador, a expressão matemática do cálculo da incidência será:
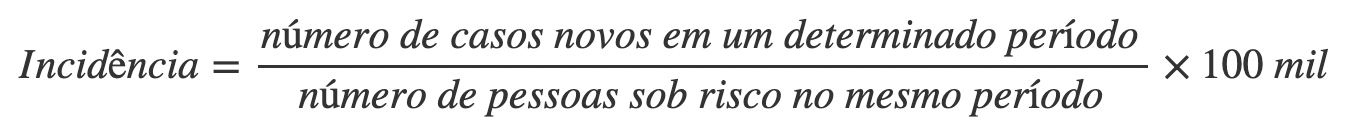
Ao realizar este cálculo, é importante especificar o período de tempo (ano ou mês, por exemplo) analisado. Dessa forma, tanto os casos novos quanto as pessoas que já estão sob risco devem se referir ao mesmo período de tempo estudado. Além disso, o numerador do cálculo da incidência (casos novos) deve fazer parte da população que estaria exposta ao risco (denominador).
Atenção
A expressão matemática para o cálculo da incidência usualmente envolve a multiplicação por 100 mil, ou seja, expressamos a incidência comparando-a a 100.000 (100 mil) habitantes. A escolha dessa unidade de referência é arbitrária, podendo ser modificada por valores como 1.000 (1 mil) ou 10.000 (10 mil). Entretanto, essa transformação é utilizada para comparação entre municípios pequenos e grandes regiões que foram padronizados para a mesma base decimal!
Nesta subseção destacaremos os cálculos da
incidência utilizando o banco de dados
{NINDINET.dbf}, que é resultado do preenchimento da Ficha
Individual de Notificação (FIN) do Estado de Rosas, exportada por meio
do Sinan Net - Sistema de Informação de Agravos de Notificação.
Acompanhe os exemplos, pratique no seu RStudio e vamos
juntos entender como calcular.
Lembre-se que todos os bancos de dados que serão utilizados neste módulo se encontram no menu lateral “Arquivos”, no módulo. Você deve fazer o download do material do curso diretamente da plataforma moodle.
Observe o script abaixo. Nele iremos calcular a incidência das hepatites virais por etiologia desconhecida (CID10 = B19) por município, apenas durante o ano de 2012, no Estado de Rosas. Para isto, acompanhe o passo a passo:
Primeiro vamos importar o banco de dados {
NINDINET.dbf}, disponível no menu lateral “Arquivos”, no módulo.Segundo, criaremos o numerador de casos novos: utilizaremos as funções
filter()para selecionar somente o CID B19 referente às hepatites virais notificadas no Sinan Net e também selecionar o ano de 2012 (o uso da funçãoyear()do pacotelubridatefoi detalhado no Módulo 3). Também utilizamos a funçãogroup_by()para realizarmos a contagem dos casos por município de residência com a funçãon()dentro dasummarise().
Acompanhe e pratique os comandos executados no script abaixo:
# criando objeto do tipo dataframe (tabela) {`Dados`} com o banco de dados
# {`NINDINET.dbf`}
dados <- read.dbf(file = 'Dados/NINDINET.dbf')
# Criando um novo objeto a partir da tabela {`Dados`}
tabela_inc_hepatite <- dados |>
# Utilizando a função filter() para diversos critérios de filtragem de dados
filter(
# Filtrando os agravos de hepatites virais (código "B19")
ID_AGRAVO == "B19",
# Filtrando apenas casos com ano de primeiros sintomas igual a 2012
year(DT_SIN_PRI) == 2012) |>
# Removendo as categorias (levels) ausentes na coluna ID_AGRAVO filtrada pelo
# uso da função droplevels()
mutate(ID_AGRAVO = droplevels(ID_AGRAVO)) |>
# Agrupando por município de residência
group_by(ID_MN_RESI) |>
# Contanto o número de casos novos por município de residência
summarise(N_CASOS_NOVOS = n())
# Visualizando a tabela resultante
tabela_inc_hepatite#> # A tibble: 14 × 2
#> ID_MN_RESI N_CASOS_NOVOS
#> <int> <int>
#> 1 610045 2
#> 2 610170 7
#> 3 610213 72
#> 4 610260 1
#> 5 610270 1
#> 6 610285 1
#> 7 610320 1
#> 8 610330 3
#> 9 610350 5
#> 10 610414 1
#> 11 610490 3
#> 12 610510 4
#> 13 610520 1
#> 14 610580 1Observe o output: como resultado, você verá uma tabela com
duas colunas. A primeira coluna (ID_MN_RESI) indica o
código de cada município e a segunda coluna (N_CASOS_NOVOS)
indica o número de casos em cada município.
- No terceiro passo vamos utilizar a população dos municípios do Estado de Rosas como nosso denominador para que possamos calcular a incidência de hepatites virais de etiologia desconhecida. Para isso, importaremos a tabela de população por município, disponível no menu lateral “Arquivos”, no módulo.
Acompanhe o script abaixo e replique em seu
RStudio:
# Criando a tabela "tabela_populacao" (dataframe) a partir do arquivo
# "tabela.csv", que contém o tamanho populacional por município de Rosas
tabela_populacao <- read_csv2("Dados/tabela.csv")
# Visualizando a tabela carregada
tabela_populacao#> # A tibble: 14 × 2
#> cod_mun POPULACAO
#> <dbl> <dbl>
#> 1 610045 15009
#> 2 610170 69189
#> 3 610213 201452
#> 4 610260 30205
#> 5 610270 19688
#> 6 610285 20448
#> 7 610320 6713
#> 8 610330 17559
#> 9 610350 10018
#> 10 610414 4164
#> 11 610490 79524
#> 12 610510 32042
#> 13 610520 43143
#> 14 610580 3434Observe que na primeira coluna desta tabela (cod_mun),
você verá novamente o código de cada município. Já na segunda coluna
(POPULACAO) temos os valores referentes ao número de
habitantes em cada município de Rosas.
Lembre-se de que é uma boa prática ao usar uma linguagem de programação ir criando novos objetos e salvando suas tabelas de análises!
Cada objeto criado armazenará seus dados e permitirá que estes sejam acessados no momento que desejar e inclusive podendo ser reutilizáveis, tornando sua análise mais rápida e segura. Caso tenha dúvida em como criar objetos acesse o Módulo 2 deste curso.
- No quarto passo, vamos agregar a população para o cálculo utilizando
a função
left_join(). Isto porque, para o cálculo da incidência, precisaremos unir a tabela de casos novos e a tabela de população. Para isso utilizaremos como chave única para cruzamento o código do município, variável comum às duas tabelas. Para isto, no argumentobydentro da funçãoleft_join()incluímos as variáveis"ID_MN_RESI"e"cod_mun".
Veja abaixo como deve ser feito o script e replique no seu
RStudio:
# Unindo a tabela "tabela_inc_hepatite" com "tabela_populacao", pela coluna com
# o código do município em cada uma
tabela_inc_hepatite |>
# unindo a tabala de casos de hepatites virais com a tabela de população
# utilizando os códigos do municípios de cada tabela
left_join(tabela_populacao, by = c("ID_MN_RESI" = "cod_mun")) |>
# Criando uma nova coluna com a incidência de casos por 100000 habitantes com
# a função mutate()
mutate(INCIDENCIA = N_CASOS_NOVOS / POPULACAO * 100000)#> # A tibble: 14 × 4
#> ID_MN_RESI N_CASOS_NOVOS POPULACAO INCIDENCIA
#> <dbl> <int> <dbl> <dbl>
#> 1 610045 2 15009 13.3
#> 2 610170 7 69189 10.1
#> 3 610213 72 201452 35.7
#> 4 610260 1 30205 3.31
#> 5 610270 1 19688 5.08
#> 6 610285 1 20448 4.89
#> 7 610320 1 6713 14.9
#> 8 610330 3 17559 17.1
#> 9 610350 5 10018 49.9
#> 10 610414 1 4164 24.0
#> 11 610490 3 79524 3.77
#> 12 610510 4 32042 12.5
#> 13 610520 1 43143 2.32
#> 14 610580 1 3434 29.1Observe no output que a nova tabela criada possui quatro colunas:
ID_MN_RESIindica o código do município,N_CASOS_NOVOSdemonstra o total de casos novos,POPULACAOcontém o tamanho populacional eINCIDENCIAapresenta o índice que queremos: a incidência de casos a cada 100 mil habitantes.
Na avaliação dos municípios do Estado de Rosas é possível verificar também que o município 610350 (Flor de Lis) possui a maior incidência, 35,7 casos por 100 mil habitantes, enquanto o município 610520 (Lótus) possui a menor incidência, com 2,3 por 100 mil habitantes no Estado.
Observe também os municípios 610330 (Glicínia) e o 610490 (Zínia) possuem o mesmo número de casos: três casos, porém suas valores de incidência bastante distintas (17,1 e 3,77 por 100 mil habitantes, respectivamente), isto ocorre pois o tamanho de suas populações é distinto!
Perceba que utilizamos sempre o operador pipe
(|>) para indicar uma sequência de ações. Caso encontre
erros para executar seu código, faça revisão de toda a escrita desde a
ortografia, até a escrita de pontos e inclusão de símbolos. Lembre-se
sempre que o comando somente será executado corretamente se o
script for escrito da maneira correta.
Para buscar ajuda, cole o erro ou o warning no seu navegador da internet (consulte o google ou o Stack Overflow).
2.2 Prevalência
A prevalência é o indicador mais utilizado para mensurar a frequência de doenças crônicas de longa duração. Isto porque seu numerador se refere ao total de pessoas que se apresentam doentes no período analisado. Na prevalência são somados os casos novos e os casos já conhecidos. A sua interpretação não se refere a risco de adoecer, e sim o quanto uma doença persiste numa população.
Para calcular esse indicador, a expressão matemática do cálculo da prevalência é:

Atenção
A expressão matemática para o cálculo da prevalência usualmente envolve a multiplicação por 100 mil, ou seja, expressamos a prevalência comparando por 100.000 (100 mil) habitantes. A escolha dessa unidade de referência é arbitrária, podendo ser modificada por valores como 1.000 (1 mil) ou 10.000 (10 mil). Entretanto, essa transformação é utilizada para comparação entre municípios pequenos e grandes regiões que foram padronizados para a mesma base decimal!!
Agora vamos praticar o cálculo deste indicador utilizando os dados referentes à prevalência de hanseníase no Estado do Acre durante o ano de 2021. Este banco de dados se encontra no menu lateral “Arquivos” no módulo!
A prevalência de hanseníase estima a magnitude da endemia no Acre. Ela deve ser estudada com base na totalidade de casos em tratamento no momento em que realizamos a sua avaliação. Uma alta prevalência (valores acima de 5 casos por 10 mil habitantes) pode indicar um cenário de baixo desenvolvimento socioeconômico e falta de ações efetivas do município para o controle da doença. Por outro lado, a baixa prevalência (valores menores que 1 caso por 10 mil habitantes) pode indicar que a hanseníase não deve considerada um problema de saúde pública.
Nesta análise, o profissional de vigilância necessitará importar o
arquivo {base_hans_ac.dbf} no formato “.dbf” dos casos de
hanseníase no Acre, disponível no menu lateral “Arquivos”, no módulo.
Lembre-se que a importação de arquivos nesse formato é feita pela função
read.dbf() do pacote foreign.
Acompanhe abaixo o script e replique o código em seu
RStudio:
# criando objeto do tipo data.frame {`base_hans`} que armazenará o banco
# de dados de hanseníase do Estado do Acre
base_hans <- read.dbf(file = 'Dados/base_hans_ac.dbf')Como vamos avaliar o ano de 2021, vamos selecionar um recorte de dois anos anteriores (2019 e 2020) e, assim, incluir os casos em tratamento que podem ter iniciado o esquema terapêutico nesses anos, mas se mantêm em andamento no ano de avaliação. Dessa forma, vamos filtrar os casos abertos diagnosticados entre 2019 e 2021. Acompanhe os passos:
Primeiro, vamos criar uma nova variável, extraindo o ano da data de diagnóstico utilizando a função
year()do pacotelubridatedentro da funçãomutate().Em seguida, vamos filtrar os casos diagnosticados entre 2019 e 2021 e, simultaneamente, vamos selecionar os casos que ainda estão em tratamento, ou seja, aqueles cuja variável tipo de saída (
TPALTA_N) ainda não foi preenchida (está nula ou em branco). Para filtrar os casos abertos, vamos utilizar a funçãois.na()para selecionar os registros com a variávelTPALTA_Nem branco, dentro da funçãofilter().Por fim, vamos contar os casos confirmados de hanseníase com a função
count(). Mas não vamos indicar nenhuma variável dentro da função, pois queremos a contagem total do filtro realizado antes.
Acompanhe o código abaixo e reproduza no seu
RStudio:
base_hans |>
# Criando uma nova coluna com o ano de diagnóstico com a função mutate() e year()
mutate(ano_diag = year(DT_DIAG)) |>
# Filtrando os casos diagnosticados entre 2019 e 2021 e "tipo de saída"
# (TPALTA_N) com valores em branco
filter(ano_diag >= 2019, ano_diag <= 2021, is.na(TPALTA_N)) |>
# Contanto o número de casos por ano de diagnóstico com a função count()
count()#> n
#> 1 171Visualize que obtivemos um output apenas com um único valor,
indicando o total de casos em branco neste conjunto de dados
(n), ou seja, temos 171 casos que ainda estão em tratamento
no Estado do Acre em 2021.
- Mas para o cálculo da prevalência precisamos da população estimada
do Estado do Acre no mesmo ano. Conseguimos essa população fazendo a
tabulação no site do Datasus e você pode importá-la por meio do arquivo
{
pop_ac_09_21.csv}. Lembre-se de copiar o caminho do arquivo localizado no menu lateral “Arquivos”, do módulo.
Acompanhe os comandos escritos no script abaixo e replique:
# Criando o objeto {`pop_ac`} com a população estimada para o Acre
# O argumento `col_types = list("character")` indica que todas as
# colunas serão lidas como strings de texto (character)
pop_ac <- read_csv2('Dados/pop_ac_09_21.csv', col_types = list("character"))Perceba que para a construção do código executado no script
acima incluímos o argumento chamado col_types. Ele foi
necessário para garantir que a primeira coluna do arquivo, chamada
“Codigo”, mantenha-se no formato original do tipo
character.
Agora vamos visualizar como ficou a tabela que criamos para armazenar
a população por ano da estimativa segundo município do Acre. Digite o
script abaixo e replique em seu RStudio:
# Visualizando as linhas e colunas do dataframe {`pop_ac`}
glimpse(pop_ac)#> Rows: 22
#> Columns: 15
#> $ Codigo <chr> "120001", "120005", "120010", "120013", "120017", "120020", …
#> $ Municipio <chr> "Acrelândia", "Assis Brasil", "Brasiléia", "Bujari", "Capixa…
#> $ `2009` <dbl> 12769, 6180, 21758, 8633, 8812, 80978, 15373, 33678, 6653, 1…
#> $ `2010` <dbl> 13081, 6335, 22325, 8838, 9179, 81907, 15754, 33816, 6862, 1…
#> $ `2011` <dbl> 13327, 6457, 22771, 8999, 9468, 82638, 16054, 33925, 7026, 1…
#> $ `2012` <dbl> 13579, 6583, 23231, 9165, 9764, 83389, 16363, 34037, 7195, 1…
#> $ `2013` <dbl> 13821, 6703, 23670, 9324, 10048, 84109, 16658, 34144, 7357, …
#> $ `2014` <dbl> 14069, 6826, 24120, 9486, 10339, 84845, 16960, 34254, 7523, …
#> $ `2015` <dbl> 14318, 6951, 24574, 9650, 10632, 85587, 17265, 34364, 7690, …
#> $ `2016` <dbl> 14551, 7067, 24996, 9803, 10905, 86279, 17550, 34467, 7845, …
#> $ `2017` <dbl> 14781, 7181, 25414, 9954, 11175, 86963, 17830, 34569, 7999, …
#> $ `2018` <dbl> 15020, 7300, 25848, 10111, 11456, 87673, 18122, 34675, 8159,…
#> $ `2019` <dbl> 15256, 7417, 26278, 10266, 11733, 88376, 18411, 34780, 8317,…
#> $ `2020` <dbl> 15490, 7534, 26702, 10420, 12008, 89072, 18696, 34884, 8473,…
#> $ `2021` <dbl> 15721, 7649, 27123, 10572, 12280, 89760, 18979, 34986, 8628,…Nesta tabela, você verá na primeira linha o código do município, na segunda linha o nome do município, e a partir da terceira linha as estimativas populacionais para cada um dos municípios por ano entre 2009 e 2021.
- Como vamos precisar apenas do ano de 2021, do estado todo, vamos
utilizar a função
summarise()para resumir e somar toda a população dos municípios usando a funçãosum(). Perceba que nas colunas da tabela {pop_ac} os anos da população estimada estão organizados em colunas. Para somar somente o ano de 2021, vamos nos referenciar à coluna utilizando crases (assim: `2021`). Isso é necessário para que oRpossa entender que estamos nos referindo a uma coluna de um dataframe e não um valor numérico (o título da coluna é 2021). Abaixo vamos criar a coluna com a população total de 2021. Replique o script abaixo em seuRStudio:
pop_ac |>
# Criando a coluna com a população total do Estado do Acre em
# 2021 com a função summarise() e sum()
summarise(pop_total_ac_2021 = sum(`2021`))#> # A tibble: 1 × 1
#> pop_total_ac_2021
#> <dbl>
#> 1 906876Você deve observar que a população do Acre, portanto, é de 906.876 habitantes em 2021.
- Por fim, calcularemos a prevalência de hanseníase no Acre em 2021 por 10 mil habitantes. Para isto, iremos substituir os valores na expressão matemática a seguir, veja:

Observe como fazemos agora no R, escreva conforme
script abaixo a operação matemática no seu
RStudio.
# Divisão do número de casos pela população do Acre, seguida da multiplicação com
# 10000
(171 / 906876) * 10000#> [1] 1.885594O resultado desta operação é o valor de 1.885594. Desta forma, a prevalência dos casos em tratamento no Estado do Acre em 2021 é de 1,89 pessoas a cada 10 mil habitantes.
- Agora, vamos calcular este indicador para todos os municípios do
estado. Para tal, vamos repetir o comando utilizado anteriormente, mas
indicando dentro da função
count()o município de residência atual (MUNIRESAT). Para fins de reprodutibilidade, iremos salvar o resultado da contagem por municípios em um novo objeto, que chamaremos de {casos_hans_ac_21}. Veja o script abaixo e repita em seuRStudio:
# Criando uma tabela (dataframe) com o nome casos_hans_ac_21
casos_hans_ac_21 <- base_hans |>
# Filtrando os casos na coluna "tipo de saída" (TPALTA_N) com valores em branco
filter(is.na(TPALTA_N)) |>
# Contando o número de casos com valores em branco por município
count(MUNIRESAT)
# Visualizando a tabela resultante
casos_hans_ac_21#> MUNIRESAT n
#> 1 120001 2
#> 2 120005 3
#> 3 120010 8
#> 4 120013 3
#> 5 120017 3
#> 6 120020 12
#> 7 120025 9
#> 8 120030 6
#> 9 120032 1
#> 10 120033 2
#> 11 120035 1
#> 12 120038 3
#> 13 120039 5
#> 14 120040 70
#> 15 120042 2
#> 16 120043 1
#> 17 120045 6
#> 18 120050 7
#> 19 120060 11
#> 20 120070 10
#> 21 120080 5
#> 22 <NA> 1A tabela resultante apresentará na primeira coluna o código do município de residência atual, enquanto a segunda coluna irá indicar o total de casos em cada um destes municípios.
Para seguir nesta etapa precisamos da população de cada município para o cálculo da prevalência. Então, seguiremos os passos abaixo:
Primeiro, utilizaremos novamente o objeto {
pop_ac} criado para armazenar a população estimada para o Acre.Segundo, iremos unir a tabelas população e a tabela de casos em tratamento utilizando a função
left_join(); para uni-las, precisamos indicar qual coluna será chave de ligação entre as duas tabelas utilizando o argumentoby, ou seja, incluímos a variávelMUNIRESATna tabela de casos e a variávelCodigoda tabela de população.
Acompanhe o script abaixo e repita em seu
RStudio:
# Criando uma tabela (dataframe) com o nome {`prev_casos_ac`}
prev_casos_ac <- casos_hans_ac_21 |>
# Juntando a tabela casos_hans_ac_21 com pop_ac, pelas colunas com os códigos de
# município de cada uma
left_join(pop_ac, by = c("MUNIRESAT" = "Codigo"))
# Visualizando a tabela resultante
prev_casos_ac#> MUNIRESAT n Municipio 2009 2010 2011 2012 2013 2014
#> 1 120001 2 Acrelândia 12769 13081 13327 13579 13821 14069
#> 2 120005 3 Assis Brasil 6180 6335 6457 6583 6703 6826
#> 3 120010 8 Brasiléia 21758 22325 22771 23231 23670 24120
#> 4 120013 3 Bujari 8633 8838 8999 9165 9324 9486
#> 5 120017 3 Capixaba 8812 9179 9468 9764 10048 10339
#> 6 120020 12 Cruzeiro do Sul 80978 81907 82638 83389 84109 84845
#> 7 120025 9 Epitaciolândia 15373 15754 16054 16363 16658 16960
#> 8 120030 6 Feijó 33678 33816 33925 34037 34144 34254
#> 9 120032 1 Jordão 6653 6862 7026 7195 7357 7523
#> 10 120033 2 Mâncio Lima 15417 15864 16216 16577 16924 17277
#> 11 120035 1 Marechal Thaumaturgo 14265 14843 15298 15765 16213 16670
#> 12 120038 3 Plácido de Castro 17695 17954 18158 18368 18569 18774
#> 13 120039 5 Porto Walter 9227 9573 9845 10125 10393 10667
#> 14 120040 70 Rio Branco 342445 350589 356998 363589 369899 376348
#> 15 120042 2 Rodrigues Alves 14450 15012 15455 15910 16345 16791
#> 16 120043 1 Santa Rosa do Purus 4658 4894 5080 5271 5454 5642
#> 17 120045 6 Senador Guiomard 20770 21053 21276 21505 21724 21948
#> 18 120050 7 Sena Madureira 38790 39676 40373 41090 41777 42478
#> 19 120060 11 Tarauacá 36351 37131 37745 38377 38981 39599
#> 20 120070 10 Xapuri 16424 16788 17074 17369 17651 17939
#> 21 120080 5 Porto Acre 15096 15524 15861 16207 16538 16877
#> 22 <NA> 1 <NA> NA NA NA NA NA NA
#> 2015 2016 2017 2018 2019 2020 2021
#> 1 14318 14551 14781 15020 15256 15490 15721
#> 2 6951 7067 7181 7300 7417 7534 7649
#> 3 24574 24996 25414 25848 26278 26702 27123
#> 4 9650 9803 9954 10111 10266 10420 10572
#> 5 10632 10905 11175 11456 11733 12008 12280
#> 6 85587 86279 86963 87673 88376 89072 89760
#> 7 17265 17550 17830 18122 18411 18696 18979
#> 8 34364 34467 34569 34675 34780 34884 34986
#> 9 7690 7845 7999 8159 8317 8473 8628
#> 10 17635 17968 18297 18638 18977 19311 19643
#> 11 17132 17563 17988 18430 18867 19299 19727
#> 12 18982 19175 19366 19565 19761 19955 20147
#> 13 10944 11201 11456 11720 11982 12241 12497
#> 14 382864 388932 394924 401155 407319 413418 419452
#> 15 17241 17660 18074 18504 18930 19351 19767
#> 16 5831 6007 6181 6362 6540 6717 6893
#> 17 22174 22385 22593 22810 23024 23236 23446
#> 18 43187 43847 44499 45177 45848 46511 47168
#> 19 40224 40805 41379 41976 42567 43151 43730
#> 20 18230 18501 18769 19048 19323 19596 19866
#> 21 17219 17538 17853 18180 18504 18824 19141
#> 22 NA NA NA NA NA NA NAPerceba que a tabela resultante irá conter, além do código do município de residência atual e o número de casos por município, uma nova coluna indicando o nome do município, e diversas colunas indicando a estimativa do tamanho populacional destes municípios para diferentes anos.
O terceiro passo será calcular a prevalência da hanseníase por município do Estado do Acre, criando uma nova coluna com o resultado do cálculo utilizando a função
mutate().Iremos selecionar somente as variáveis que nos importam nesta tabela (
MUNIRESAT,Municipio,prevalencia_2021). Acompanhe o script abaixo e replique-o em seuRStudio:
prev_casos_ac |>
# Criando uma coluna com o cálculo de prevalência da população por 10 mil
# habitantes em 2021 com a função mutate()
mutate(prevalencia_2021 = (n / `2021`) * 10000) |>
# Selecionando apenas as colunas MUNIRESAT, Municipio e prevalencia_2021 com
# a função select()
select(MUNIRESAT, Municipio, prevalencia_2021)#> MUNIRESAT Municipio prevalencia_2021
#> 1 120001 Acrelândia 1.2721837
#> 2 120005 Assis Brasil 3.9220813
#> 3 120010 Brasiléia 2.9495262
#> 4 120013 Bujari 2.8376844
#> 5 120017 Capixaba 2.4429967
#> 6 120020 Cruzeiro do Sul 1.3368984
#> 7 120025 Epitaciolândia 4.7420834
#> 8 120030 Feijó 1.7149717
#> 9 120032 Jordão 1.1590172
#> 10 120033 Mâncio Lima 1.0181744
#> 11 120035 Marechal Thaumaturgo 0.5069195
#> 12 120038 Plácido de Castro 1.4890554
#> 13 120039 Porto Walter 4.0009602
#> 14 120040 Rio Branco 1.6688441
#> 15 120042 Rodrigues Alves 1.0117873
#> 16 120043 Santa Rosa do Purus 1.4507471
#> 17 120045 Senador Guiomard 2.5590719
#> 18 120050 Sena Madureira 1.4840570
#> 19 120060 Tarauacá 2.5154356
#> 20 120070 Xapuri 5.0337260
#> 21 120080 Porto Acre 2.6121937
#> 22 <NA> <NA> NAObserve que a tabela resultante irá apresentar, além das informações com o código e nome do município, os valores de prevalência para cada um deles no ano de 2021. Observe no output acima que a prevalência da hanseníase no Estado do Acre varia de 0,5 por 10 mil habitantes no município de Marechal Thaumaturgo até 5 por 10 mil habitantes no município de Xapuri.
Agora, vamos avançar um pouco mais e aprender a calcular com o
software R alguns indicadores de mortalidade!


