5. Outros gráficos de interesse da vigilância em saúde
5.1 Gráficos temporais para séries irregulares
As séries temporais irregulares costumam apresentar frequência na
unidade de tempo diferentes durante o período avaliado, ou seja, é
irregular conforme o tempo é modificado. Por exemplo, alguns anos têm 52
semanas e outros 53. Isso é uma irregularidade. Para trabalhar com
séries com esta característica, vamos usar o pacote
xts.
Com este pacote é possível organizar uma série calendário (irregular) baseada na data dos primeiros sintomas, por exemplo, e visualizá-la em um gráfico. Para isso, o processo é similar ao que estamos utilizando: preparamos os dados, atribuímos a um objeto e plotamos em um gráfico.
Então vamos lá. Continuaremos utilizando a base de dados de dengue do estado fictício de Rosas. Acompanhe os passos para preparar os dados:
Primeiro, vamos definir um período utilizando a função
filter().Em seguida, vamos fazer uma contagem de casos notificados de dengue por ano e semana epidemiológica utilizando a função
count(). Note que as variáveisano_epiesem_epiforam criadas logo após a importação do {NINDINET.dbf} quando filtramos os casos de dengue.Agora, vamos extrair o dia de início das semanas utilizando a função
get_date()do pacoteaweek. Vamos definir o argumentostartcomo7, que significa que a semana inicia no domingo, como padrão das semanas epidemiológicas.Vamos organizar a base considerando a ordem do menor ano e menor semana para maior ano e maior semana.Vamos atribuir o resultado desse procedimento ao objeto
dengue_semana.
Veja o script completo a seguir e replique no seu computador:
# Criando o objeto {`dengue_semana`}
dengue_semana <- dengue |>
# Filtrando os registros dentre intervalo de datas
filter(DT_SIN_PRI >= '2007-01-01' & DT_SIN_PRI <= '2012-12-31') |>
# Contando a frequência de notificações por ano epidemiológico e semana
# epidemiológica
count(ano_epi, sem_epi, name = 'n_casos') |>
# Utilizando a função `mutate()` para criar a coluna de data de início
# da semana epidemiológica
mutate(data = get_date(sem_epi, ano_epi, start = 7)) |>
# Ordenando pelo ano e semana epidemiológica
arrange(ano_epi, sem_epi)Atenção
Lembre-se que você deverá sempre verificar se todos os pacotes
necessários foram instalados e carregados no R, caso
encontre algum aviso (warning) ou erro (error) revise seu código
cuidadosamente e rode-o novamente.
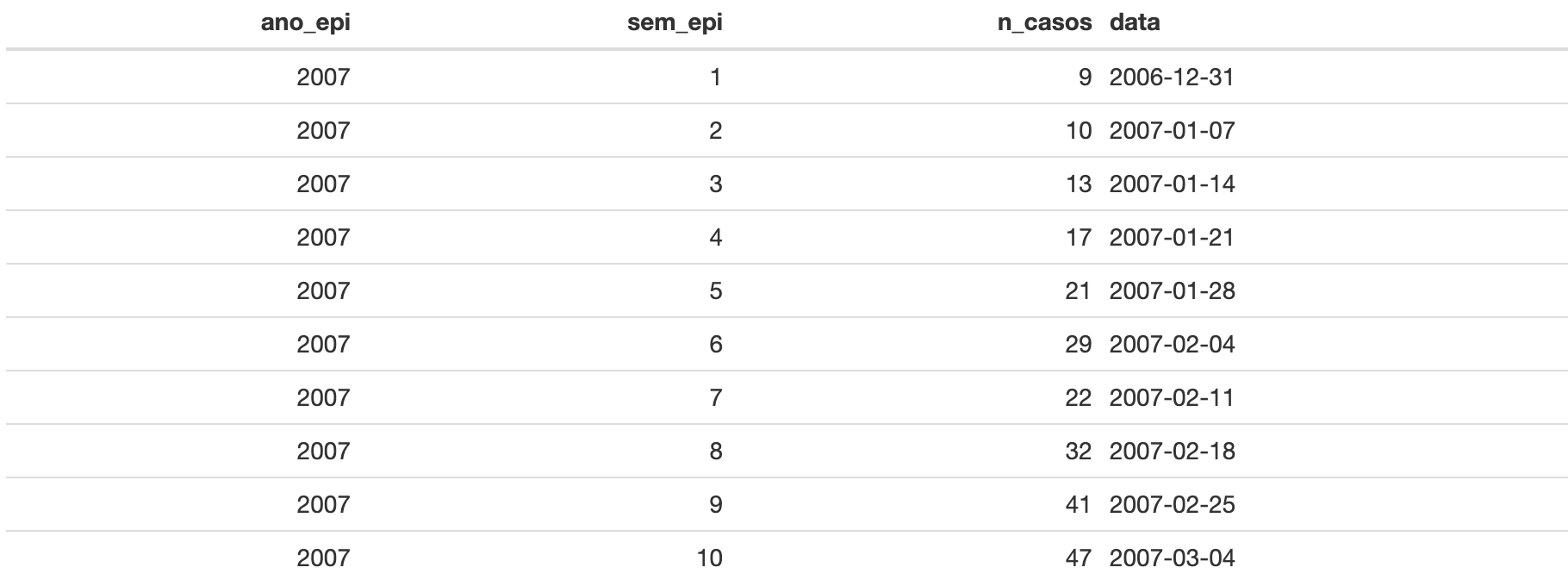
Pronto. Agora, vamos visualizar as primeiras linhas da Tabela 4,
armazenada no objeto {dengue_semana}, que acabamos de criar
utilizando a função head() alinhada à função
kable() do pacote knitr. Essa função cria uma
tabela para ser visualizada em arquivos HTML. Acompanhe o
script a seguir e replique-o em seu RStudio:
kable(head(dengue_semana, 10))Tabela 4: Organização dos dados para criação de um gráfico de linhas (Figura 14).
Pacotes úteis para lidar com datas
Já vimos que o pacote lubridate tem funções bem
interessantes para a manipulação de datas, entre elas
epiweek() e epiyear(), que retornam a semana e
o ano epidemiológico na definição do Centers for Disease
Control (CDC), ou seja, a mesma usada no Brasil.
O pacote aweek também tem funções para lidar com dados por
semana. No exemplo a seguir, estamos calculando a data de início da
semana epidemiológica usando a função aweek(). No entanto,
você tem de estar atento ao valor padrão dessa função, que não é semana
epidemiológica. Por isso, é necessário definir o argumento
start como 7.
# exemplo de como extrair a data de início da semana 1,
# no ano 2020
get_date(1, 2020, start = 7)#> [1] "2019-12-29"O resultado deve ser “2019-12-29” que indica que a primeira semana de 2020 se iniciou em 29/12/2020.
Com os dados prontos, iremos agora utilizar a função xts
do pacote xts. Para isso, será necessário definir dois
argumentos:
x: argumento que deverá indicar os dados a serem plotados. Ou seja, colocaremos noxa coluna da base de dados {dengue_foz} com os valores da incidência de casos (n_casos).order.by: argumento que deverá indicar a data correspondente aos dados indicados no argumentox. Aqui, utilizaremos a coluna com os valores da data de início das semanas epidemiológicas (data).
Perceba que utilizaremos o operador $ (cifrão)
para indicar a seleção das variáveis dentro da base de dados. Observe o
script a seguir e replique-o em seu RStudio:
# criando o no objeto {`dengue_irregular`}
dengue_irregular <- xts(
# selecionando a variável com os dados dos casos
x = dengue_semana$n_casos,
# selecionando a variável que contém as datas correspondentes
order.by = dengue_semana$data)Também poderemos visualizar a série como um gráfico de linhas na
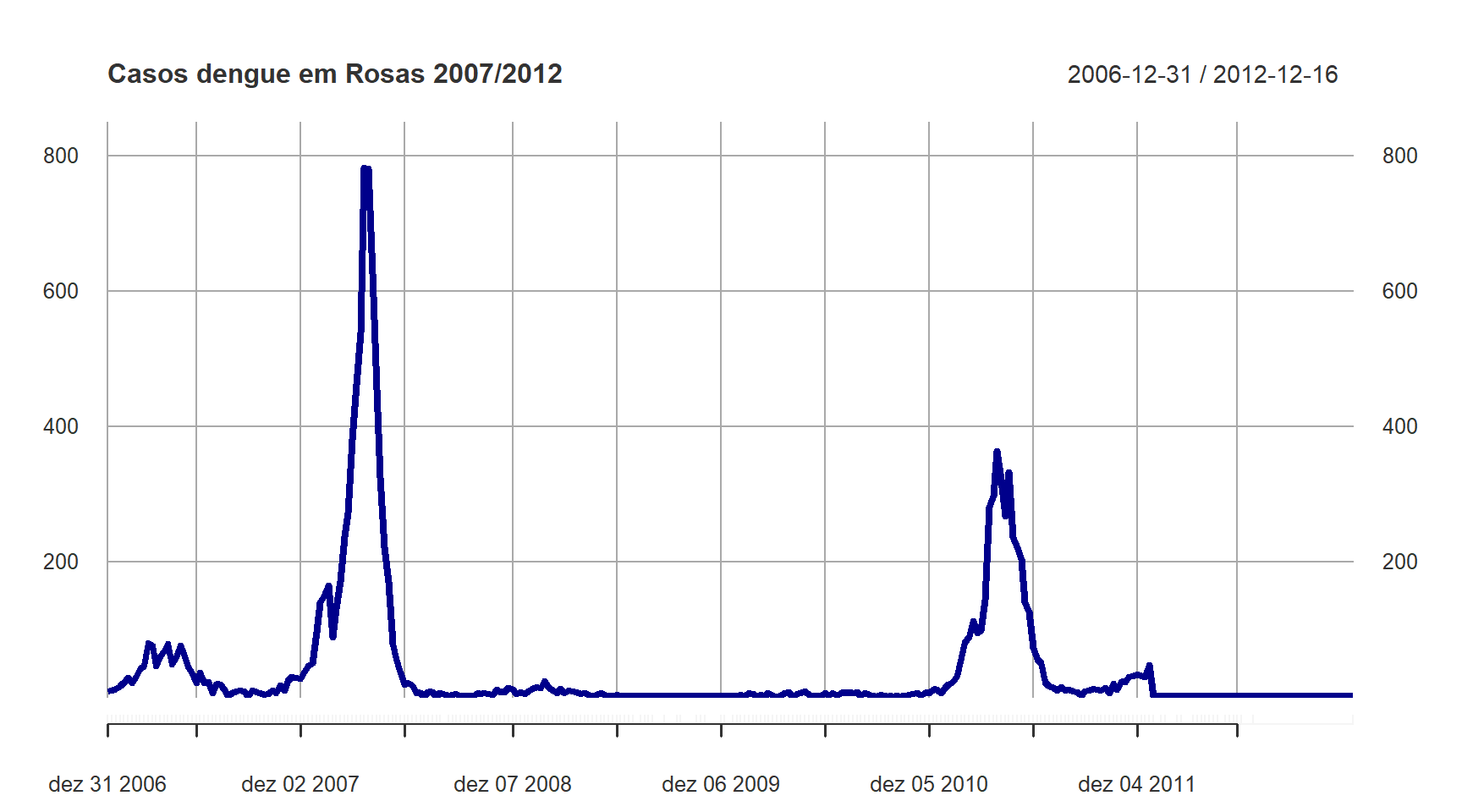
função plot(), apresentado na Figura 14:
par(mfrow=c(1,1))
plot(
# indicando a série temporal
dengue_irregular,
# Definindo o título
main = 'Casos dengue em Rosas 2007/2012',
# Definindo a cor das linhas
col = 'darkblue',
# Definindo a espessura das linhas
lwd = 4,
# Definindo o intervalo do eixo y
ylim = c(0, 850)
)Figura 14: Gráfico de linhas da distribuição dos casos de dengue do Estado de Rosa, entre 2007 e 2012.
5.2 Gráficos temporais de calor (heatmaps)
O gráfico de calor, também conhecido pelo seu nome em inglês heatmap descreve valores para uma variável dividido entre duas outras variáveis em uma grade de quadrados que, conforme o valor da variável principal, a cor fica mais intensa.
Mais uma vez vamos usar os casos de dengue como exemplo:
- em primeiro lugar vamos filtrar os casos de 2007 a 2011;
- em seguida vamos contar o número de casos por dia de primeiros sintomas e atribuir a um objeto dengue_heat.
Acompanhe o script a seguir e replique-o em seu
RStudio:
# Criando o objeto {`dengue_heat`}
dengue_heat <- dengue |>
# Filtrando os registros dentre intervalos de datas
filter(DT_SIN_PRI >= "2007-01-01" & DT_SIN_PRI <= "2011-12-31") |>
# Contando a frequência de notificações por data dos primeiros sintomas
count(DT_SIN_PRI, name = 'casos') |>
# Utilizando a função `mutate()` para criar a coluna de ano de início
# dos primeiros sintomas
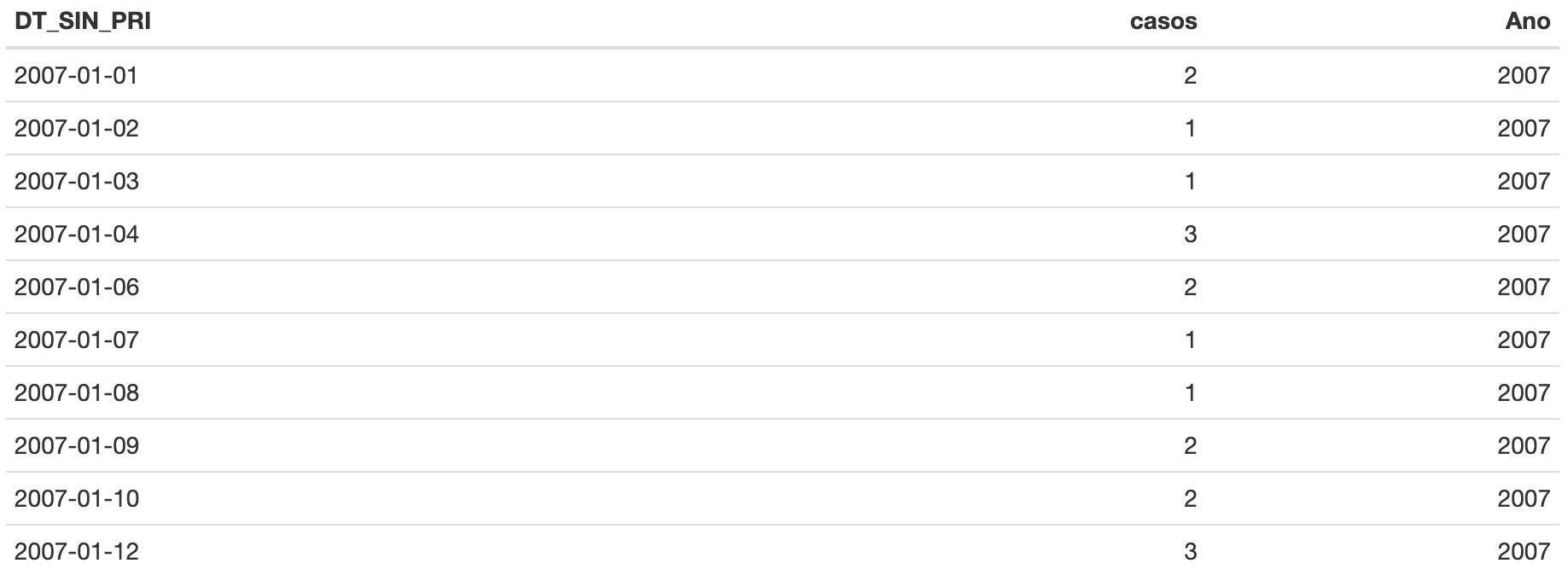
mutate(Ano = year(DT_SIN_PRI))Agora, vamos visualizar as primeiras linhas, conforme Tabela 5, com o
script a seguir. Repita em seu RStudio:
kable(head(dengue_heat, 10))Tabela 5: Tabela {dengue_semana} criada para armazenar os dados que serão utilizados no gráfico temporal (Figura 15).
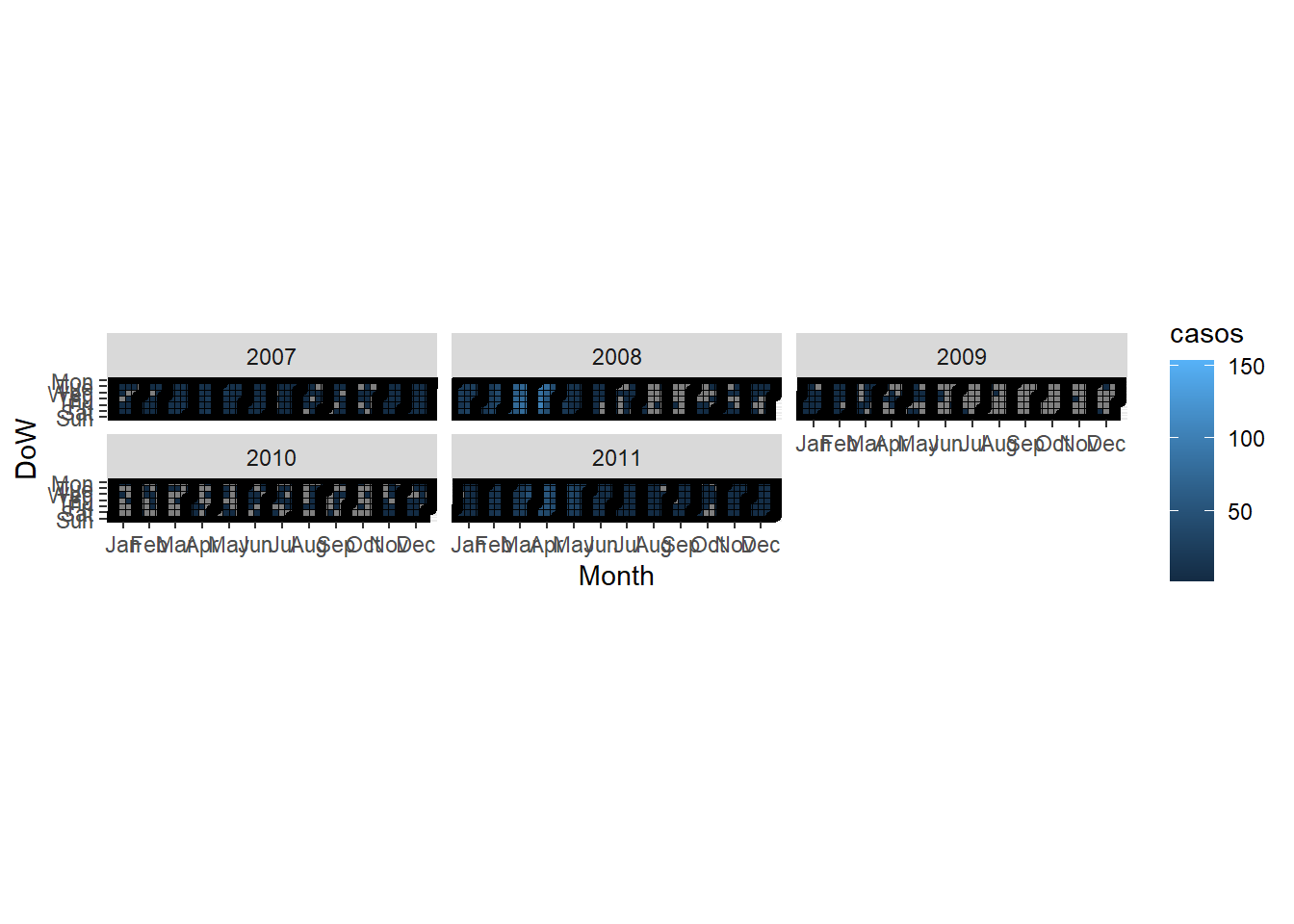
O pacote ggTimeSeries adiciona uma série de funções, entre elas a que vamos utilizar para fazer mapas de calor tipo calendário. Entretanto, as funções básicas do pacote não oferecem um visual que seja fácil de interpretar. Veja a Figura 15 a seguir, observe o padrão no código e replique-o no seu computador:
ggplot_calendar_heatmap(dengue_heat, 'DT_SIN_PRI','casos')Figura 15: Gráfico temporal de calor da distribuição dos casos de dengue do Estado de Rosa, entre 2007 e 2011.
Atenção
Lembre-se que você deverá sempre verificar se todos os pacotes
necessários foram instalados e carregados no R, caso
encontre algum aviso (warning) ou erro (error) revise seu código
cuidadosamente e rode-o novamente.
Fica bem difícil de visualizar os dias, não é mesmo? Vários comandos adicionais podem ser usados para estabelecer os rótulos em meses e dias da semana e também para atribuir uma escala diferente de cores.
Vamos, então, montar um gráfico (Figura 16) com funções do pacote
ggplot2 para deixar mais profissional. Observe o
script a seguir e replique-o em seu RStudio:
ggplot(data = dengue_heat) +
# Definindo argumentos estéticos com as variáveis usadas em x e em y
# e a variável usada para o preenchimento da série
aes(date = DT_SIN_PRI, fill = casos) +
# Adicionando geometria de `heatmap`
stat_calendar_heatmap() +
# Definindo a rampa de cores de preenchimento
scale_fill_gradient2(
low = 'lightblue',
high = 'red',
mid = 'orange',
midpoint = 75
) +
# Arrumando o eixo y, definindo quais os marcadores (`breaks`)
# serão mostrados que, no caso, será uma sequência de 1 a 7, referente aos
# dias da semana. O argumento `expand` ajuda nesse processo.
scale_y_continuous(
breaks = c(1:7),
labels = c("Seg", "Ter", "Qua", "Qui", "Sex", "Sab", "Dom"),
expand = c(0, 0)
) +
# Arrumando o eixo x, definindo quais os marcadores (`breaks`) e
# os rótulos do gráfico `labels`.
scale_x_continuous(
breaks = c(2, 6, 11, 16, 19, 24, 28, 33, 37, 42, 46, 50),
labels = c(
'Jan', 'Fev', 'Mar', 'Abr', 'Mai', 'Jun',
'JUl', 'Ago', 'Set', 'Out', 'Nov', 'Dez'
),
expand = c(0, 0)
) +
# Definindo a estratificação por ano e o visual com uma coluna
facet_wrap( ~ Ano, ncol = 1) +
# Definindo o tema base
theme_light() +
# Definindo título do gráfico
labs(
title = "Mapa de calor dos casos notificados de dengue, estado de Rosas,
2007-2012.",
fill = "Número de casos \nnotificados"
)Figura 16: Gráfico temporal de calor, utilizando ggplot2, da distribuição dos casos de dengue do Estado de Rosa, entre 2007 e 2011.
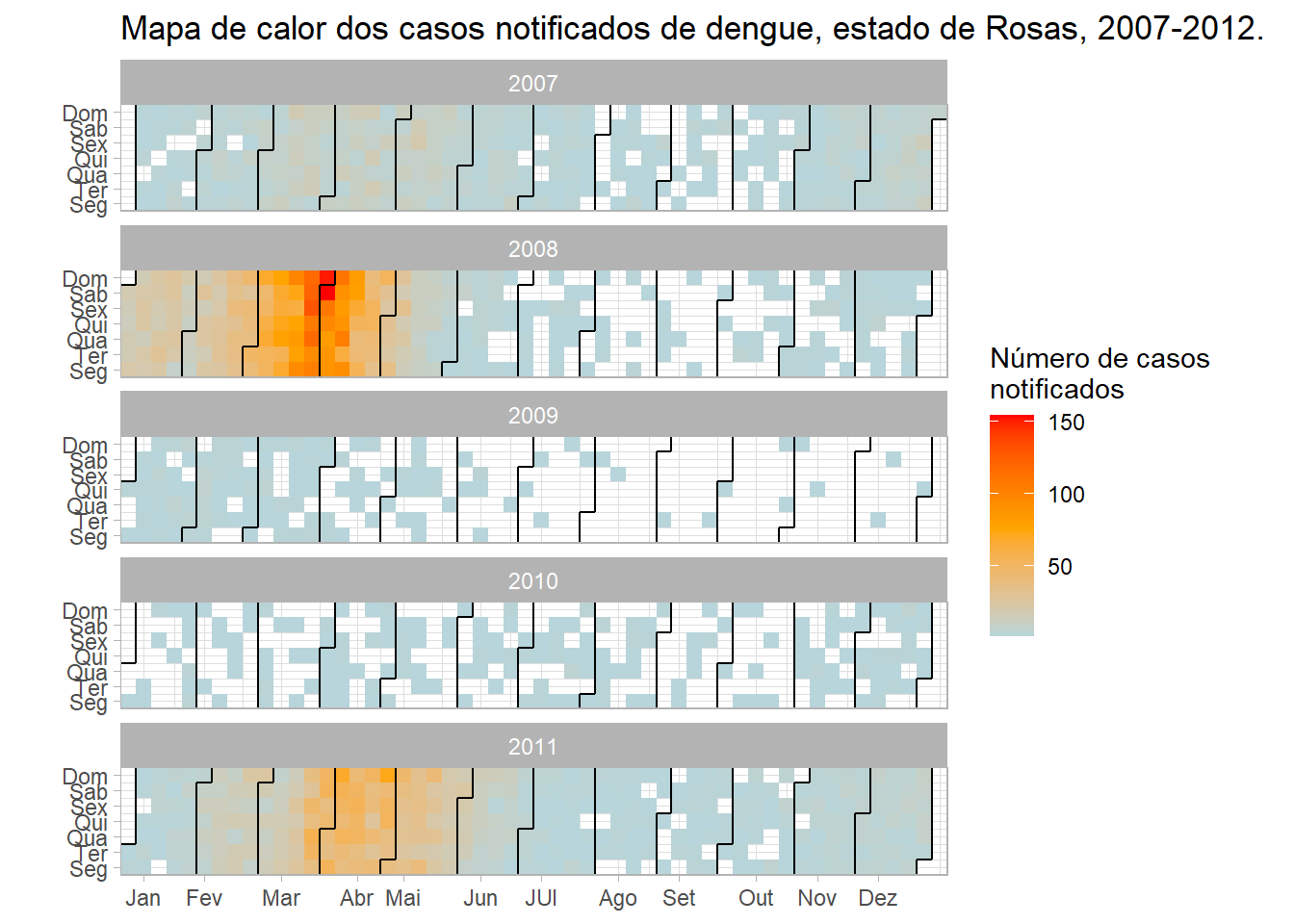
Uau! Perceba no gráfico acima que o conjunto extra de comandos torna o gráfico em algo muito diferente e bem mais fácil de interpretar e visualizar.
5.3 Gráficos temporais com média móvel
Na covid-19 muitos profissionais de vigilância utilizaram gráficos temporais com a inclusão da média móvel de casos e óbitos de covid-19. A média móvel é sempre utilizada quando precisamos incluir no gráfico o cálculo da média aritmética das observações mais recentes, ou seja, quando precisamos saber a tendência média ao longo do tempo. Vamos ver como isso se dá na prática?
Primeiro, organizaremos os dados para o gráfico de linhas padrão,
demonstrando o comportamento da doença que estamos analisando. Acompanhe
o script a seguir e replique-o em seu RStudio:
dengue_semana <- dengue |>
# Filtrando os registros com data de primeiros sintomas maior ou igual
# a data de primeiro de janeiro de 2007.
filter(DT_SIN_PRI >= '2007-01-01') |>
# Utilizando a função `mutate()`pra criar novas colunas
mutate (
# Criando uma nova coluna chamada 'SEM_EPI` referente à semana
# epidemiológica dos primeiros sintomas
SEM_EPI = epiweek(DT_SIN_PRI),
# Criando uma nova coluna chamada 'ANO_EPI` referente ao ano
# epidemiológicos dos primeiros sintomas
ANO_EPI = epiyear(DT_SIN_PRI),
# Criando uma nova coluna chamada 'DT_INI_SEM` referente à
# data de início da semana epidemiológica
DT_INI_SEM = get_date(SEM_EPI, ANO_EPI)
) |>
# Agrupando as notificações pelo ano epidemiológico, semana
# epidemiológica e data de início da semana epidemiológica
group_by(ANO_EPI, SEM_EPI, DT_INI_SEM) |>
# Contando a frequência de notificações
count(name = 'casos')Agora vamos preparar os dados para a média móvel. Observe o
script a seguir e replique-o em seu RStudio:
dengue_sem2 <- dengue_semana |>
# Filtrando os registros com data maior ou igual
# a data de primeiro de janeiro de 2007.
filter(DT_INI_SEM >= '2010-01-01') |>
# Desagrupando os dados
ungroup() |>
# Substituindo os valores faltantes por zero
replace_na(list(casos = 0)) |>
# Utilizando a função `mutate()` para criar colunas
mutate(
# Criando uma nova coluna chamada 'mm14` referente à média
# móvel dos casos notificados de dengue
mm14 = zoo::rollmean(casos, 14, fill = TRUE, align = 'center'),
# Criando uma nova coluna referente ao rótulo que será
# usado no gráfico
rotulo = sprintf('%02d-%s', SEM_EPI, str_sub(ANO_EPI, 3, 4))
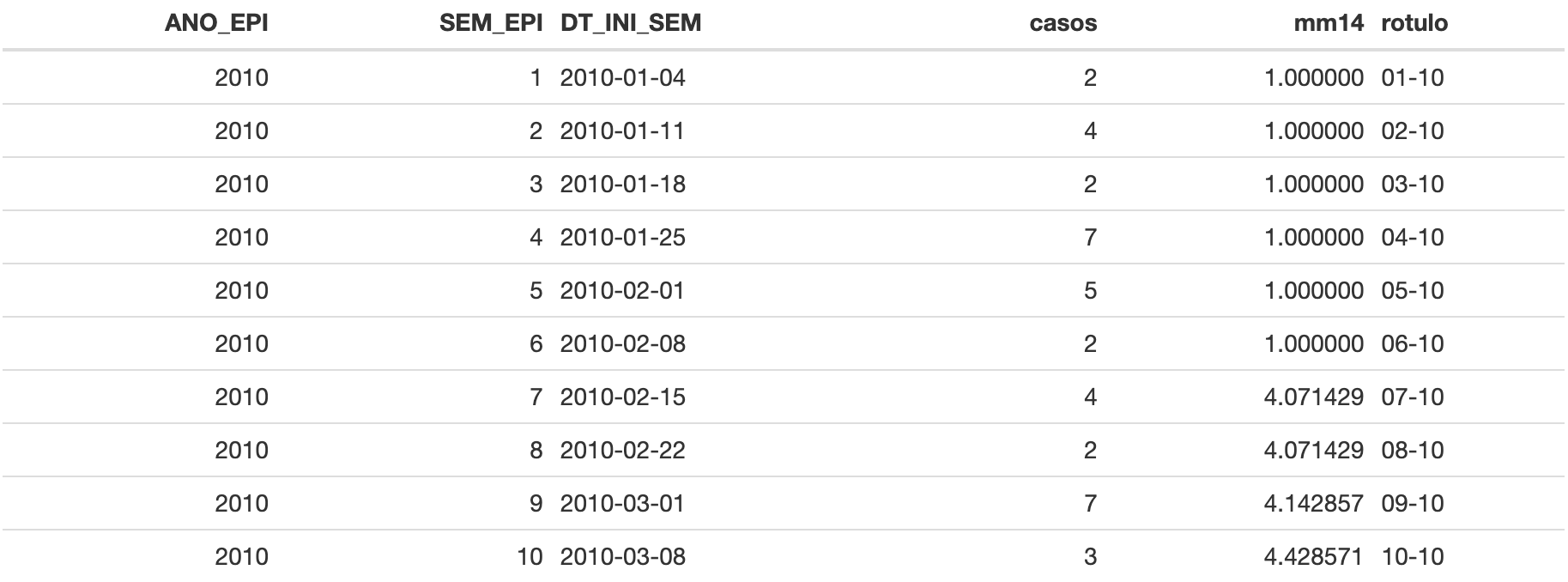
)Agora, vamos visualizar as primeiras linhas, conforme Tabela 6, com o
script a seguir. Repita em seu RStudio:
kable(head(dengue_sem2, 10))Tabela 6: Tabela {dengue_sem2} com dados
organizados para gerar um gráfico de média móvel (Figura 17).
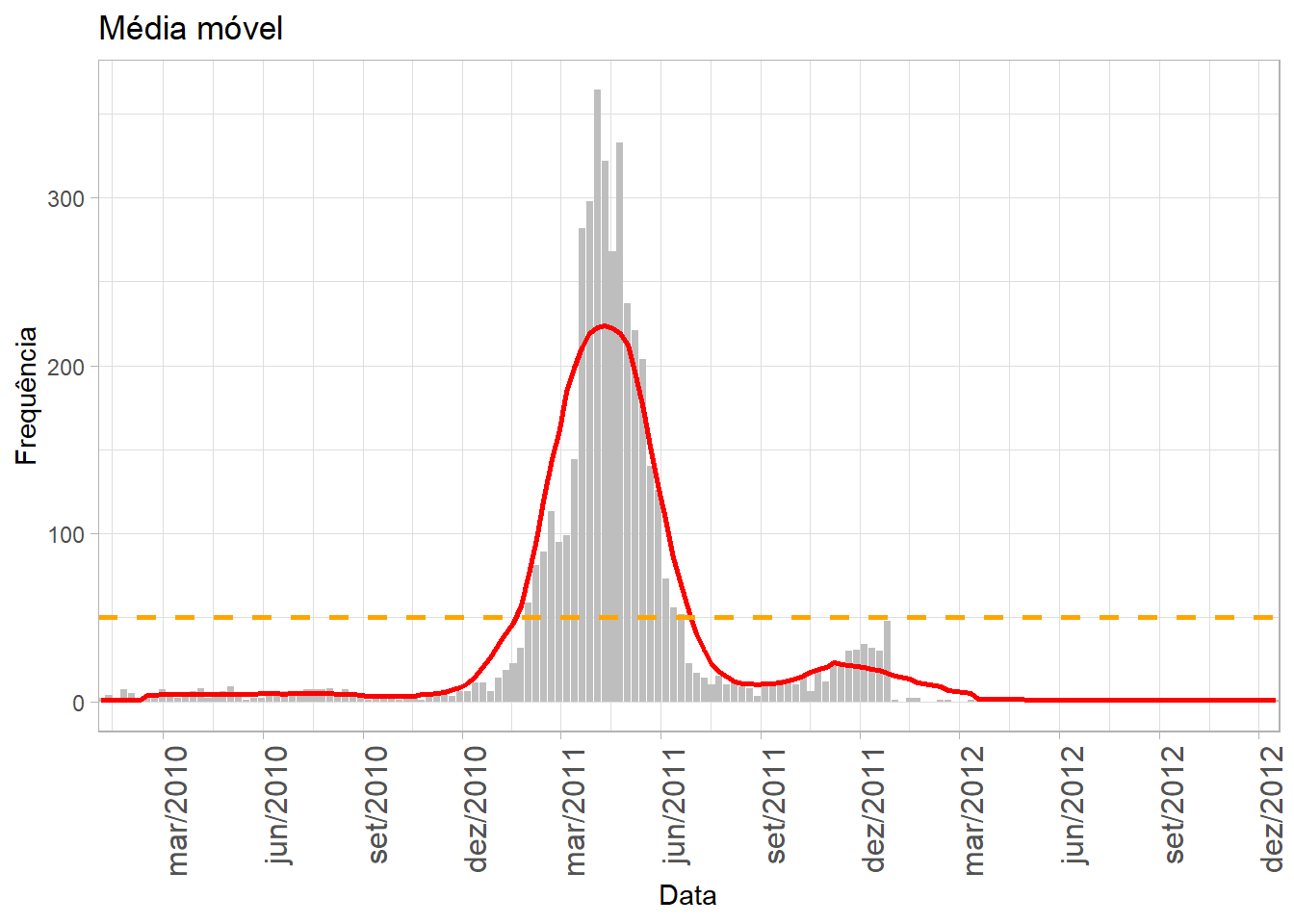
Pronto. Agora, faremos um gráfico de barras em cinza (Figura 17), com
gráfico de linha com a média móvel em vermelho e, por fim, uma linha
pontilhada laranja mostrando 50 casos. Observe o script a
seguir e replique-o em seu RStudio:
# Criando o objeto gráfico {`gsem1`}
gsem1 <- ggplot(data = dengue_sem2) +
# Definindo argumentos estéticos com as variáveis usadas em x e em y
aes(x = DT_INI_SEM, y = casos) +
# Adicionado a geometria de barras e
# definindo a variável usada para o preenchimento
geom_col(fill = "gray") +
# Adicionando a geometria de linhas e definindo a cor e
# a espessura
geom_line(aes(y = mm14), color = 'red', size = 1) +
# Adicionado uma geometria de linha cruzando com o
# eixo y no valor 50 e definindo a linha como
# tracejada (`dashed`), cor laranja e espessura
geom_hline(
yintercept = 50,
linetype = 'dashed',
color = 'orange',
size = 1
) +
# Arrumando a data do eixo x, definindo os intervalos
# (de 3 em 3 meses) e o rótulo
scale_x_date(date_breaks = '3 months',
date_labels = '%b/%Y',
expand = c(0, 0)) +
# Definindo o tema base
theme_light() +
# Alterando o tema do gráfico
theme(
# Alterando o texto do eixo x
axis.text.x = element_text(
angle = 90,
hjust = 1,
size = 12,
color = 'grey32'
)) +
# Definindo o título d gráfico e títulos dos eixos x e y
labs(
title = "Média móvel",
x = "Data",
y = "Frequência"
)
gsem1Figura 17: Gráfico da média móvel e marcação dos 50 casos de dengue em Rosas, entre 2010 e 2012.
Com o objeto gsem1 será possível modificar alguns
aspectos como a escala do eixo X para representar as datas por semanas
epidemiológicas e alterar o tamanho das letras dos respectivos eixos.
Observe essas modificações plotadas na Figura 18 a seguir.
Acompanhe o script e replique-o em seu computador:
gsem1 +
# Definindo os títulos dos eixos x e y
labs(x = "Semana epidemiológica", y = "Casos") +
# Arrumando o eixo x, definindo os marcadores (`breaks`)
# e os rótulos
scale_x_date(date_breaks = '3 weeks',
date_labels = '%W/%Y',
expand = c(0, 0)) +
# Alterando o tema do gráfico
theme(
# Alterando o texto do eixo x
axis.text.x = element_text(
angle = 90,
hjust = 1,
size = 6,
color = 'grey32'
),
# Alterando o texto do eixo y
axis.text.y = element_text(
hjust = 1,
size = 14,
color = 'grey32'
),
# Definindo que as grades do gráfico
# nulas
panel.grid.major.x = element_blank() ,
panel.grid.minor.x = element_blank()
)#> Scale for 'x' is already present. Adding another scale for 'x', which will
#> replace the existing scale.Figura 18: Gráfico da média móvel e marcação dos 50 casos de dengue em Rosas, entre 2010 e 2012, por semana epidemiológica.
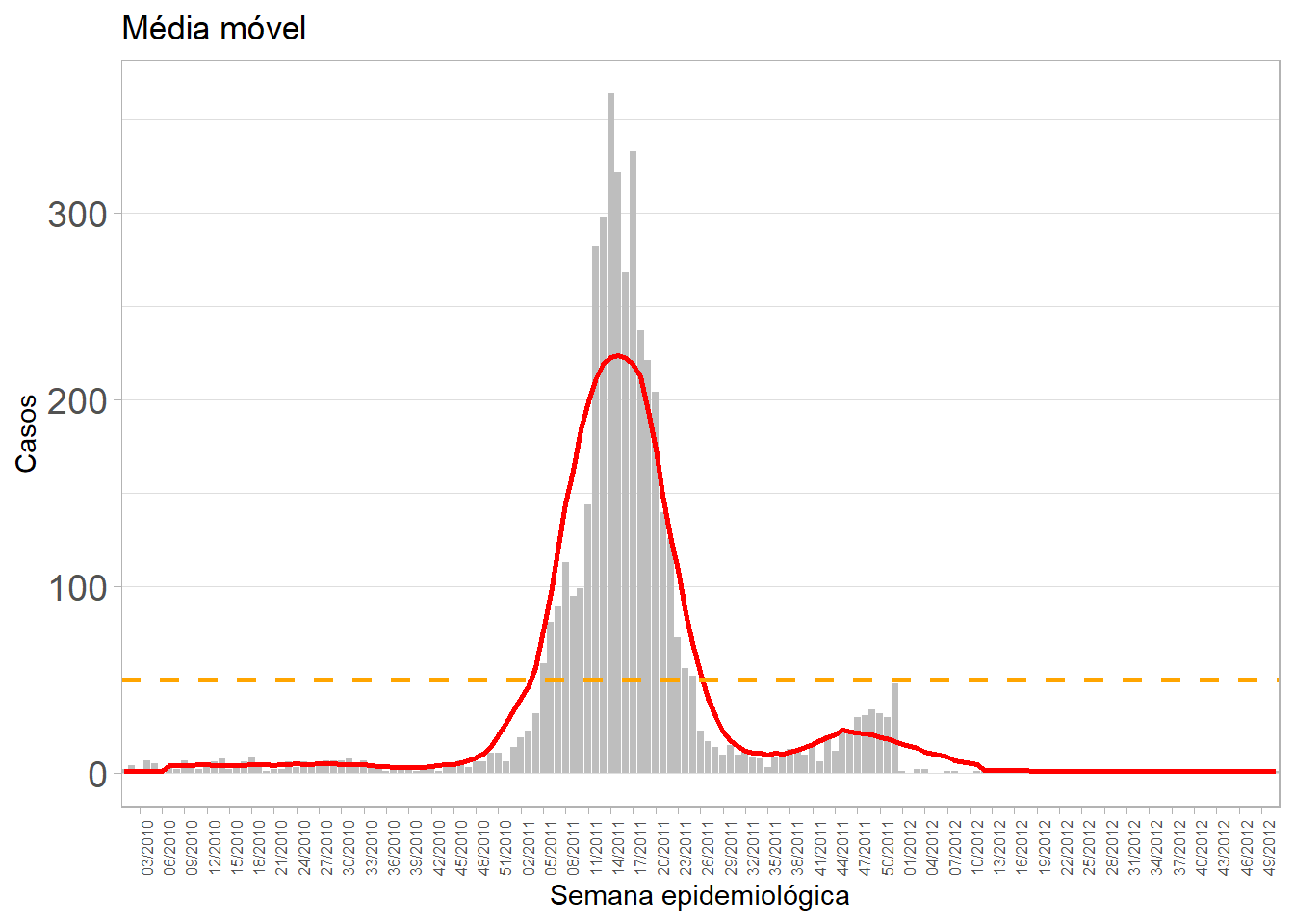
Ficou bacana, não é mesmo? Experimente aplicar o cálculo da média móvel para outras doenças ou agravos analisados em seu dia a dia.
5.4 Pirâmides etárias
Pirâmides etárias são amplamente utilizadas na vigilância em saúde. E
com o apoio da linguagem R será possível produzi-las sempre
que desejar. Aqui iremos construir pirâmide utilizando sexo e idade das
notificações de dengue do Estado de Rosas. O primeiro passo será remover
os poucos casos em que a variável sexo é igual a “I”
(ignorados) e depois criar as faixas etárias com que vamos trabalhar na
pirâmide para isso usaremos a função cut(), que permite
categorizar uma variável contínua (NU_IDE_N, no presente
caso), em categorias. Para isso informamos os pontos de corte (breaks)
e, de forma opcional, os labels.
Em seguida vamos contar os casos por sexo e faixa etária criando uma
variável chamada n e, por fim, vamos multiplicar
n por -1 quando sexo for igual a “F”. Essa configuração
é necessária para espelhar o eixo de frequência do lado oposto ao padrão
e, assim, criarmos as barras para o sexo masculino e feminino (um à
esquerda e outro à direita, como uma pirâmide etária). Acompanhe o
código a seguir e replique-o no seu RStudio:
# Criando o objeto {`piramide`}
piramide <- dengue |>
# Filtrando os registros com sexo diferente de "I" (ignorado)
filter(CS_SEXO != 'I') |>
# Utilizando a função `mutate()` para criar colunas
mutate(
# Criando uma coluna de idade conforme a codificação da variável NU_IDADE_N
idade_anos = if_else(str_sub(NU_IDADE_N, 1, 1) == "4",
as.numeric(str_sub(NU_IDADE_N, 2, 4)), 0),
# Criando uma coluna de faixa etária a partir da variável idade dos casos
# notificados
# utilizando a função `cut()`
fx_etaria = cut(
# Definindo qual variável será classificada em faixas
x = idade_anos,
# Definindo os pontos de corte das classes
breaks = c(-Inf, 10, 20, 30, 40, 50, 60, Inf),
# Definindo o tipo do ponto de corte
right = FALSE,
include.lowest = FALSE,
# Definindo os rótulos das classes
labels = c("0-9", "10-19", "20-29", "30-39", "40-49", "50-59", "60 anos e+")
)
) |>
# Contando a frequência da faixa etária e sexo
count(fx_etaria, CS_SEXO) |>
# Utilizando a função `mutate()` para criar a coluna que
# vai receber as configurações da pirâmide etária
mutate(n = ifelse (CS_SEXO == 'F', n * -1, n))Agora, vamos visualizar as primeiras linhas, conforme Tabela 7, com o
script a seguir. Repita em seu RStudio:
kable(head(piramide, 10))Tabela 7: Tabela {piramide} com dados
organizados para gerar um gráfico no formato pirâmide (Figura 19).
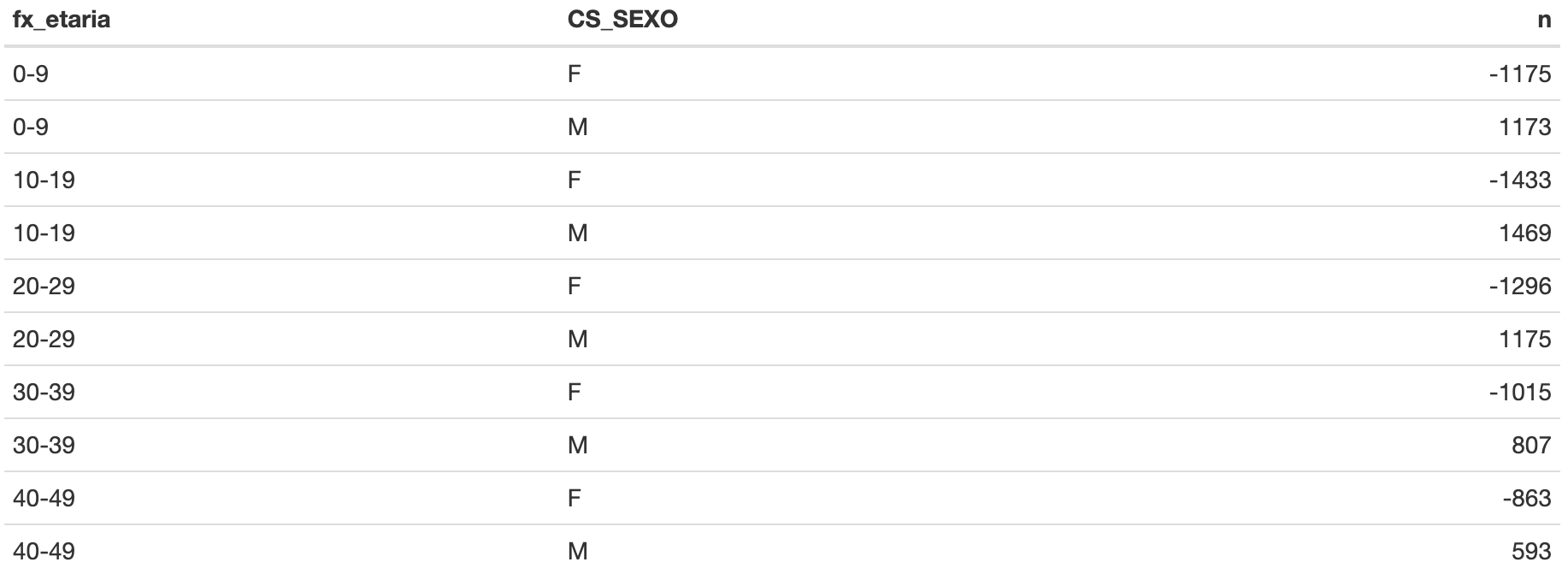
Agora, perceba que vamos configurar o gráfico, Figura 19, para a
visualização da pirâmide etária de casos de dengue em Rosas. Observe o
script a seguir e replique-o em seu RStudio:
ggplot(data = piramide) +
# Definindo argumentos estéticos com as variáveis usadas em x e em y
# e a variável usada para o preenchimento das barras da pirâmide
aes(x = n,
y = factor(fx_etaria),
fill = fct_drop(CS_SEXO)) +
# Adicionando a geometria de colunas, definindo
# a largura e o tipo de colunas
geom_col(width = 0.9, position = "identity") +
# Arrumando o eixo x, definindo os rótulos
# a serem mostrados
scale_x_continuous(labels = abs(c(-200, -100, 0, 100, 200))) +
# Definindo os títulos dos eixos x, y e título da legenda do gráfico
labs(
title = 'Pirâmide etária dos casos notificados de dengue no estado de Rosas,
2007-2012',
x = 'Número de casos notificados',
y = 'Faixa Etária',
fill = 'Sexo'
) +
# Definindo o tema base
theme_light() Figura 19: Gráfico no formato pirâmide com a distribuição dos casos de dengue em Rosas por sexo.
5.5 Gráficos interativos
Com o R você também poderá criar gráficos interativos
que aceitam zoom, seleção ou alguma função com clique do
mouse e, assim, apresentá-los de forma mais dinâmica os
gráficos produzidos na vigilância em saúde. Para isso o pacote
plotly é uma ótima ferramenta. Esse pacote transforma os
gráficos estáticos criados pelo pacote ggplot2 em gráficos
dinâmicos. Essa é uma funcionalidade muito atrativa para páginas
online, painéis de dados e arquivos offline
disponibilizados via e-mail, por exemplo.
Para transformar os gráficos do ggplot2 em gráficos
interativos, basta utilizarmos a função ggploty() do pacote
plotly, utilizando apenas um argumento essencial que é o
objeto gráfico. Vamos adicionar interatividade ao gráfico de linhas
criado na seção anterior.
Acompanhe a linha de código a seguir e repita no seu computador:
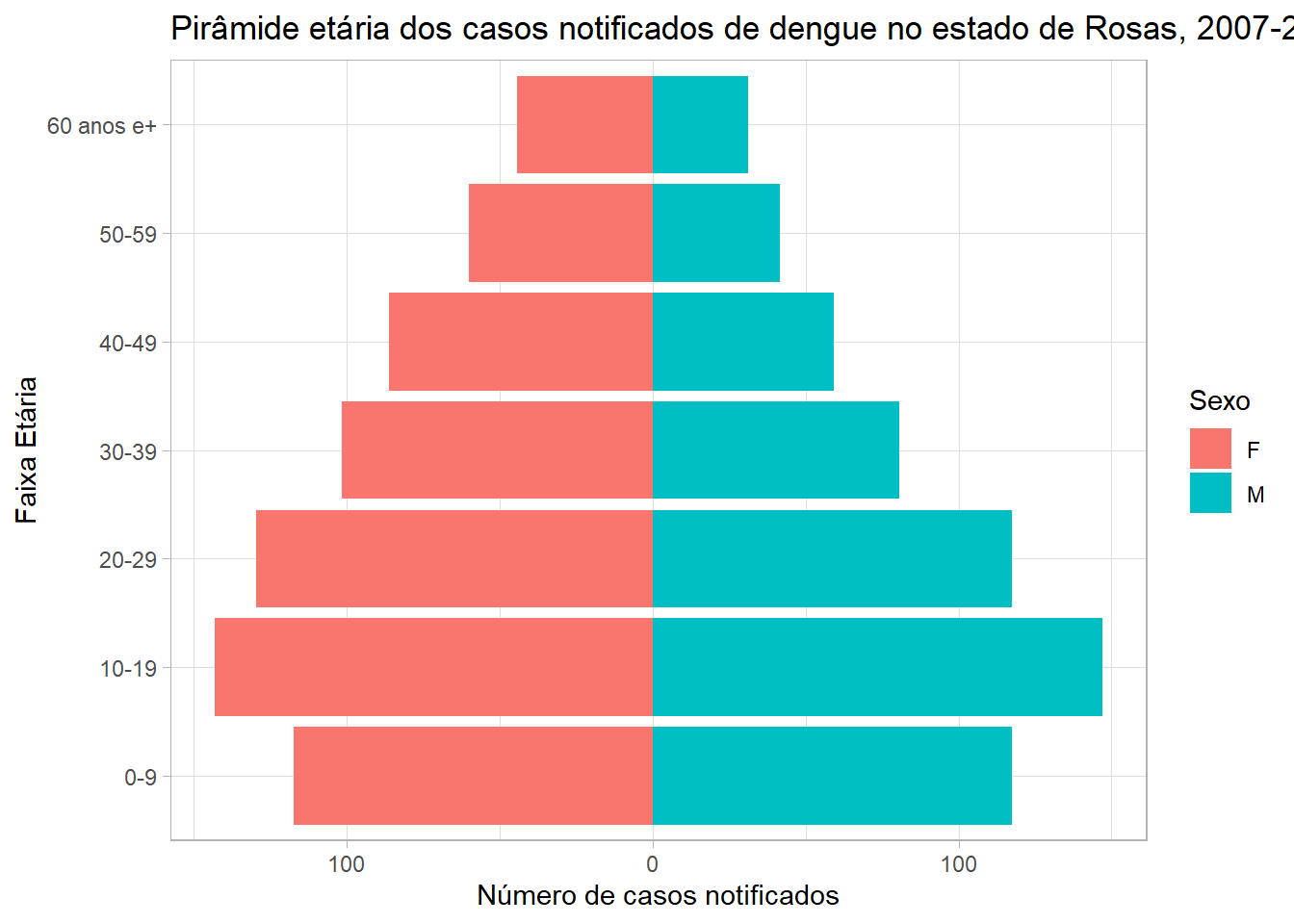
ggplotly(graf_linhas)Agora observe o gráfico apresentado na Figura 20.
Figura 20: Gráfico interativo.
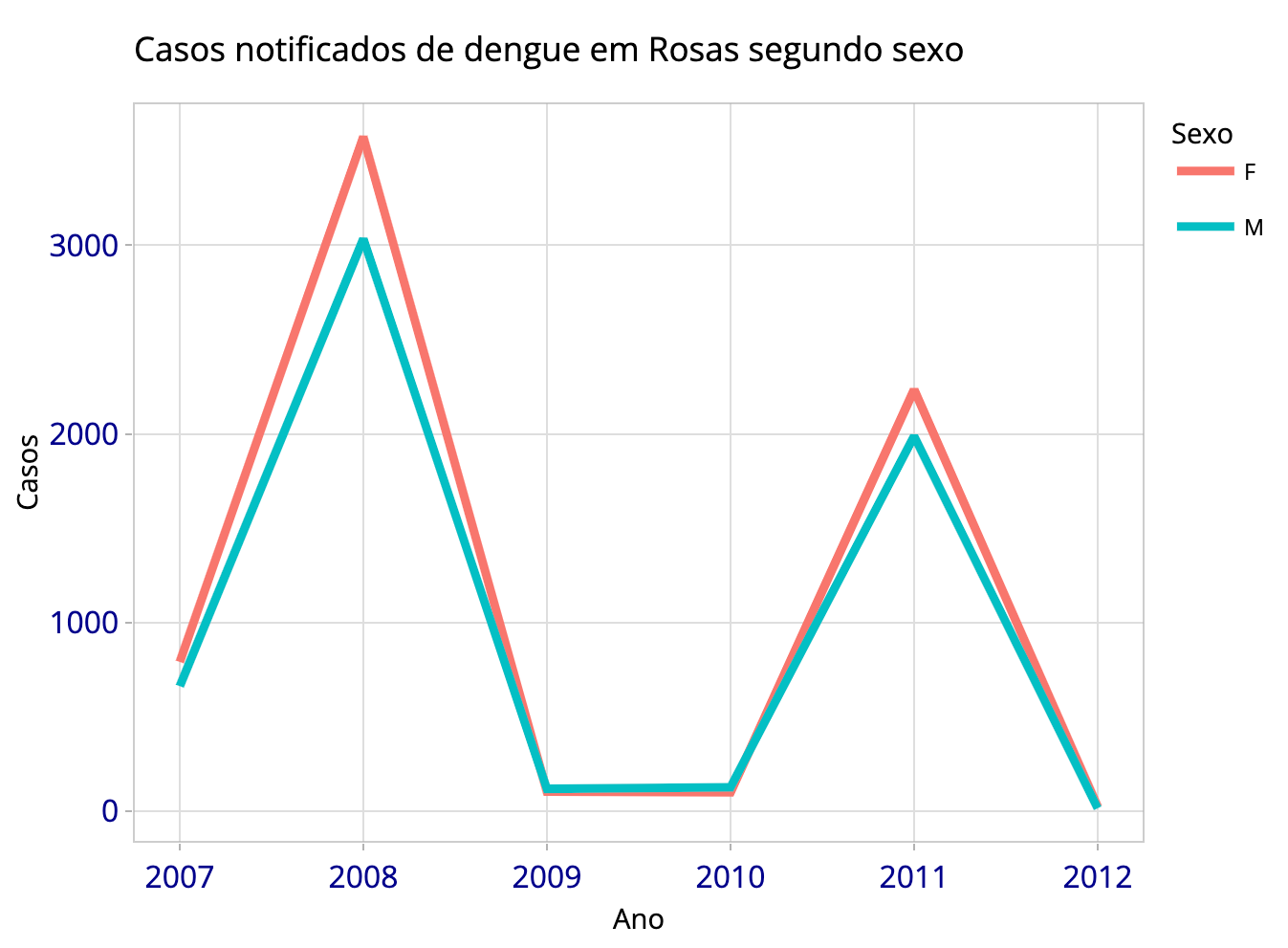
Perceba que o gráfico gerado na Figura 20 será visualizado no painel
Viewer do RStudio. Ao mover o mouse pelo gráfico,
você verá etiquetas que mostram dados da tabela
{graf_linhas} utilizada para montá-lo. No canto direito
deste espaço, você perceberá que há vários comandos de zoom, de mover e
salvar o gráfico como imagem. Explore as possibilidades de interação,
veja como fica na Figura 21 a seguir.
Figura 21: Visualização do gráfico interativo no Painel Viewer.
5.6 Estratificação por variáveis de interesse
Uma opção importante com o pacote ggplot2 é a possibilidade de criar painéis que estratificam um gráfico em sub-gráficos de uma maneira simples podendo controlar vários aspectos desses como arranjo, escalas, cores e outros.
No exemplo a seguir, você pode ver o gráfico de linhas para os 5
distritos administrativos do estado de Rosas. Como sempre, vamos
preparar o dado contando a ocorrência de casos suspeitos de dengue por
semana epidemiológica em cada um dos distritos e, em seguida, criar um
gráfico de linhas onde cada distrito recebe uma cor. Acompanhe os
scripts a seguir e replique-os no seu RStudio:
# Criando o objeto {`dengue_distritos`}
dengue_distritos <- dengue |>
# Filtrando os registros com data de início dos
# sintomas maior ou igual a 1 de janeiro de 2010
filter(DT_SIN_PRI >= '2010-01-01') |>
# Agrupando as notificações pelo ano epidemiológico, semana
# epidemiológica e distritos administrativos
group_by(ano_epi, sem_epi, ID_DISTRIT) |>
# Contando a frequência de notificações
count(name = "casos") |>
# Utilizando a função `mutate()` para criar a coluna
# de data de início da semana epidemiológica
mutate(data_ini = get_date(sem_epi, ano_epi, start = 7))
# Criando o gráfico
ggplot(data = dengue_distritos) +
# Definindo argumentos estéticos com as variáveis usadas em x e em y
# e a variável usada para a cor
aes(x = data_ini, y = casos, color = ID_DISTRIT) +
# Adicionando a linha referente aos casos
geom_line() +
# Definindo o tema base
theme_classic()Figura 22: Gráfico de linhas da distribuição de casos de dengue por distrito de Rosas, entre 2010 e 2013.
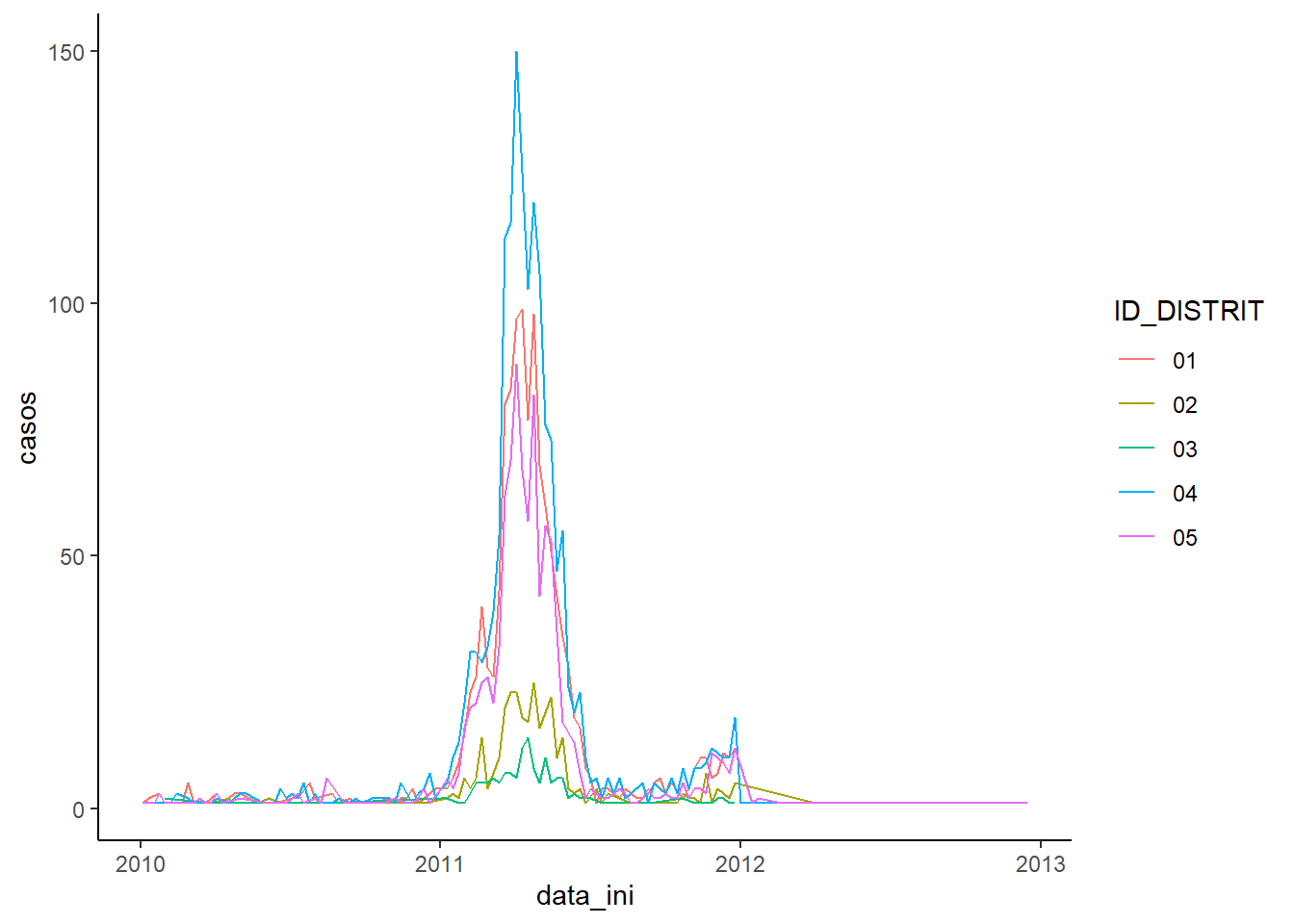
O script acima criou uma tabela do tipo dataframe
com dados de dengue notificados a partir de janeiro de 2010, agrupados e
sumarizados por ano e semana epidemiológica em cada distrito
administrativo do Estado de Rosas, no objeto
{dengue_distritos}. Em seguida, conforme Figura 22, plotou
um gráfico de linhas representa cada distrito apresentado.
Mas perceba que na Figura 22 as linhas se sobrepõem e fica um pouco difícil visualizar o comportamento da doença em cada distrito. Ainda mais porque alguns tiveram muitos casos e outros bem menos. Para melhorar esta visualização, conforme Figura 23, vamos criar sub-gráficos para cada distrito.
No ggplot2 há duas funções para criar sub-gráficos
estratificados por alguma variável:
facet_grid(),facet_wrap().
A primeira, facet_grid(), utiliza como argumento
principal o operador til (~) seguido da variável que será
estratificada em sub-gráficos. Isso significa que a função irá espalhar
as categorias da variável por uma linha, um ao lado do outro.
Acompanhe o script a seguir e replique-os no seu
RStudio:
ggplot(data = dengue_distritos) +
# Definindo argumentos estéticos com as variáveis usadas em x e em y
# e a variável usada para a cor
aes(x = data_ini, y = casos, color = ID_DISTRIT) +
# Adicionando a linha referente aos casos
geom_line() +
# Estratificando pelo distrito
facet_grid(~ID_DISTRIT) +
# Definindo o tema base
theme_light() +
# Definindo o título do gráfico.
labs(title = "Número de casos notificados de dengue no estado de Rosas segundo
o distrito de residência do paciente, 2010-2012.")Figura 23: Gráfico de linhas da distribuição de casos de dengue por distrito de Rosas, entre 2010 e 2013, segundo distrito de residência.
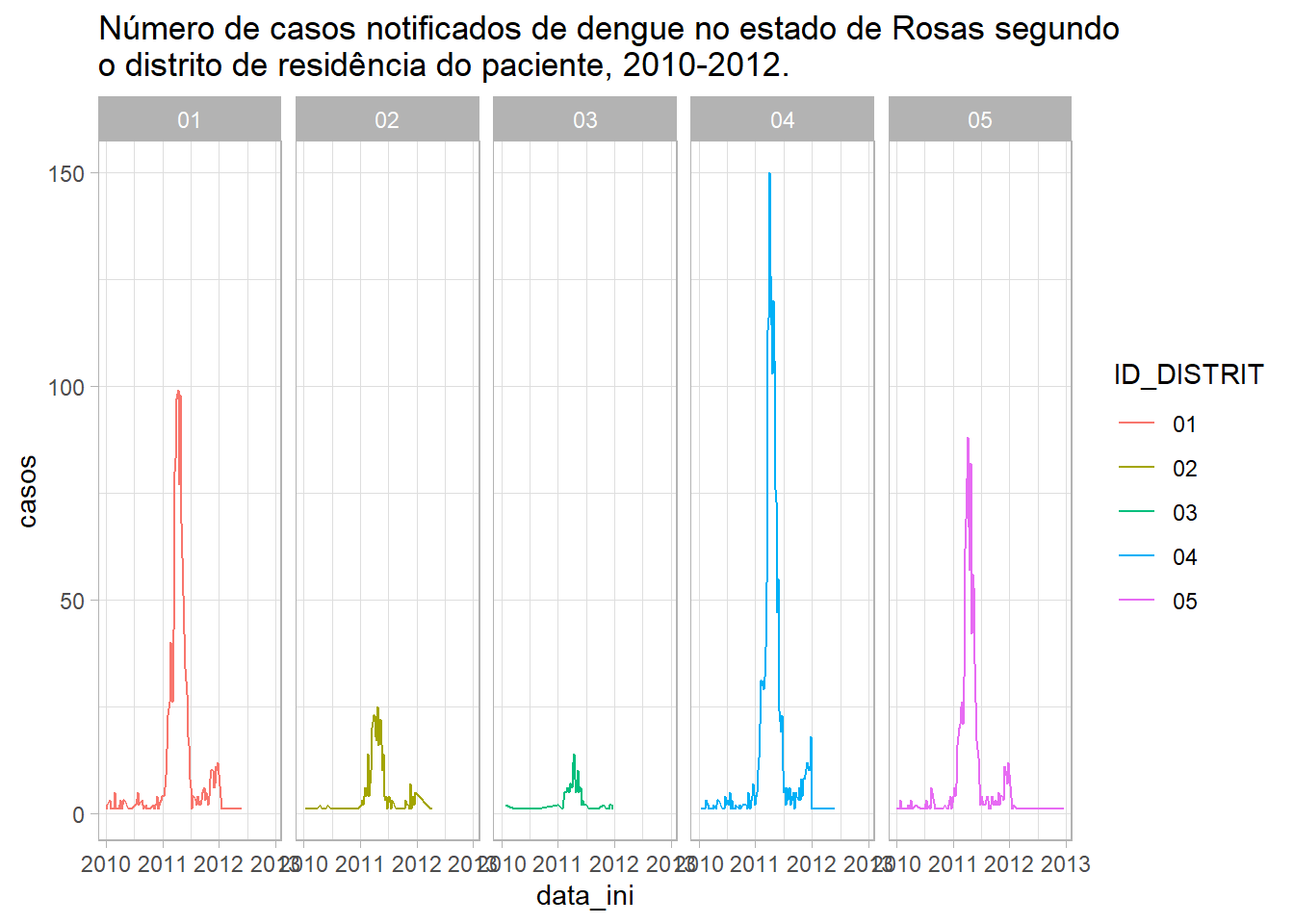
Percebeu o quanto a visualização melhorou? O título dos sub-gráficos são as categorias da variável distrito, o eixo x repete para cada sub-gráfico, mas o eixo y é o mesmo para todos.
A função facet_wrap() funciona da mesma forma e tem o
mesmo tipo de argumento. Uma das diferenças entre as duas é o
output da função. Utilizando a mesma tabela criada
anteriormente, {dengue_distritos}, vamos criar o mesmo
gráfico anterior, só mudando para facet_wrap().
Acompanhe o script a seguir e replique-os no seu
RStudio:
ggplot(data =dengue_distritos) +
# Definindo argumentos estéticos com as variáveis usadas em x e em y
# e a variável usada para a cor
aes(x = data_ini, y = casos, color = ID_DISTRIT) +
# Adicionando a linha referente aos casos
geom_line() +
# Estratificando pelo distrito
facet_wrap( ~ ID_DISTRIT) +
# Definindo o tema base
theme_light() +
# Definindo o título do gráfico.
labs(
title = "Número de casos notificados de dengue no estado de Rosas segundo
o distrito de residência do paciente, 2010-2012."
)Figura 24: Gráfico de linhas, com a função facet_wrap(), da distribuição de casos de dengue por distrito de Rosas, entre 2010 e 2013.
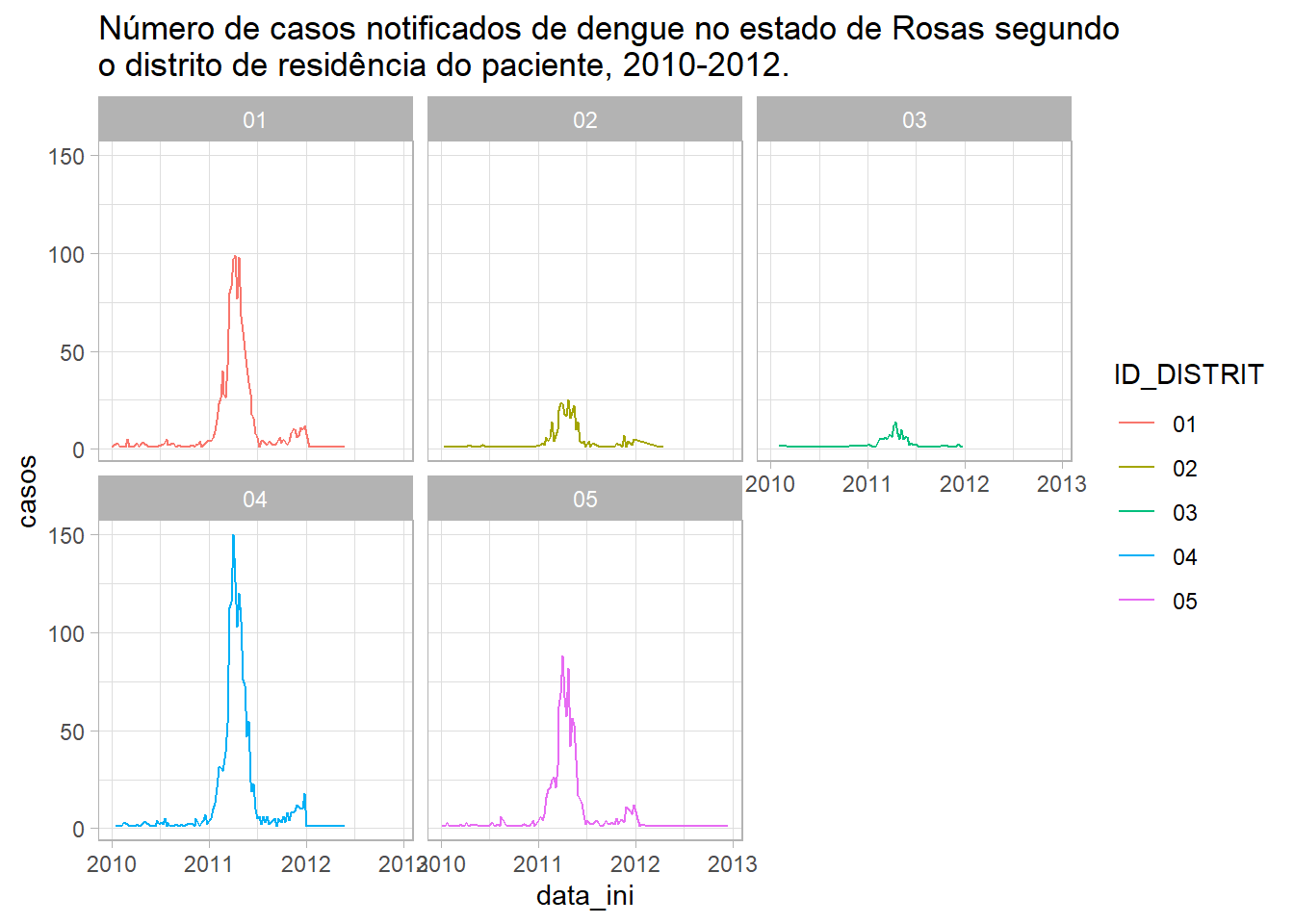
Percebeu a diferença? Os sub-gráficos foram posicionados em três colunas, sendo a linha “quebrada” para outra, na qual foram posicionados mais dois. Outra diferença é que agora o eixo x é compartilhado com alguns sub-gráficos e o eixo y se repete quando troca de linha de sub-gráficos.
A quantidade de colunas e linhas pode ser controlada. Perceba o código a seguir, no qual definimos que a disposição dos sub-gráficos (Figura 25) será em duas colunas, deixando livre o número de linhas. Além disso, vamos posicionar a legenda a seguir de todos os sub-gráficos e definir uma escala de cores diferentes do padrão dado pelo pacote, ou seja, vamos criar uma rampa de cores utilizando códigos hexadecimais.
Acompanhe o código a seguir e repita no seu computador:
# Criando uma rampa de cores utilizando códigos hexadecimais
cores <- c("#66C2A5" , "#FC8D62" , "#8DA0CB" , "#E78AC3", "#A6D854")
ggplot(data =dengue_distritos) +
# Definindo argumentos estéticos com as variáveis usadas em x e em y
# e a variável usada para a cor
aes(x = data_ini, y = casos, color = ID_DISTRIT) +
# Adicionando geometria de linha e definindo a espessura
geom_line(size = 0.8) +
# Estratificando pelo distrito e definindo 2 colunas de disposição dos
# sub-gráficos
facet_wrap(~ ID_DISTRIT, ncol = 2) +
# Definindo os títulos dos eixos x e y
# # Definindo o título do gráfico
labs(
title = "Número de casos notificados de dengue no estado de Rosas segundo
o distrito de residência do paciente, 2010-2012.",
x = 'Data ',
y = 'Casos dengue')+
# Arrumando a rampa de cores conforme o vetor criado (`cores`)
scale_color_discrete(type = cores, name = 'Distritos') +
# Definindo o tema base
theme_light() +
# Alterando o tema do gráfico, posicionando a legenda para baixo
theme(legend.position = "bottom")Figura 25: Gráfico de linhas, com duas colunas e legenda inferior central, da distribuição de casos de dengue por distrito de Rosas, entre 2010 e 2013.
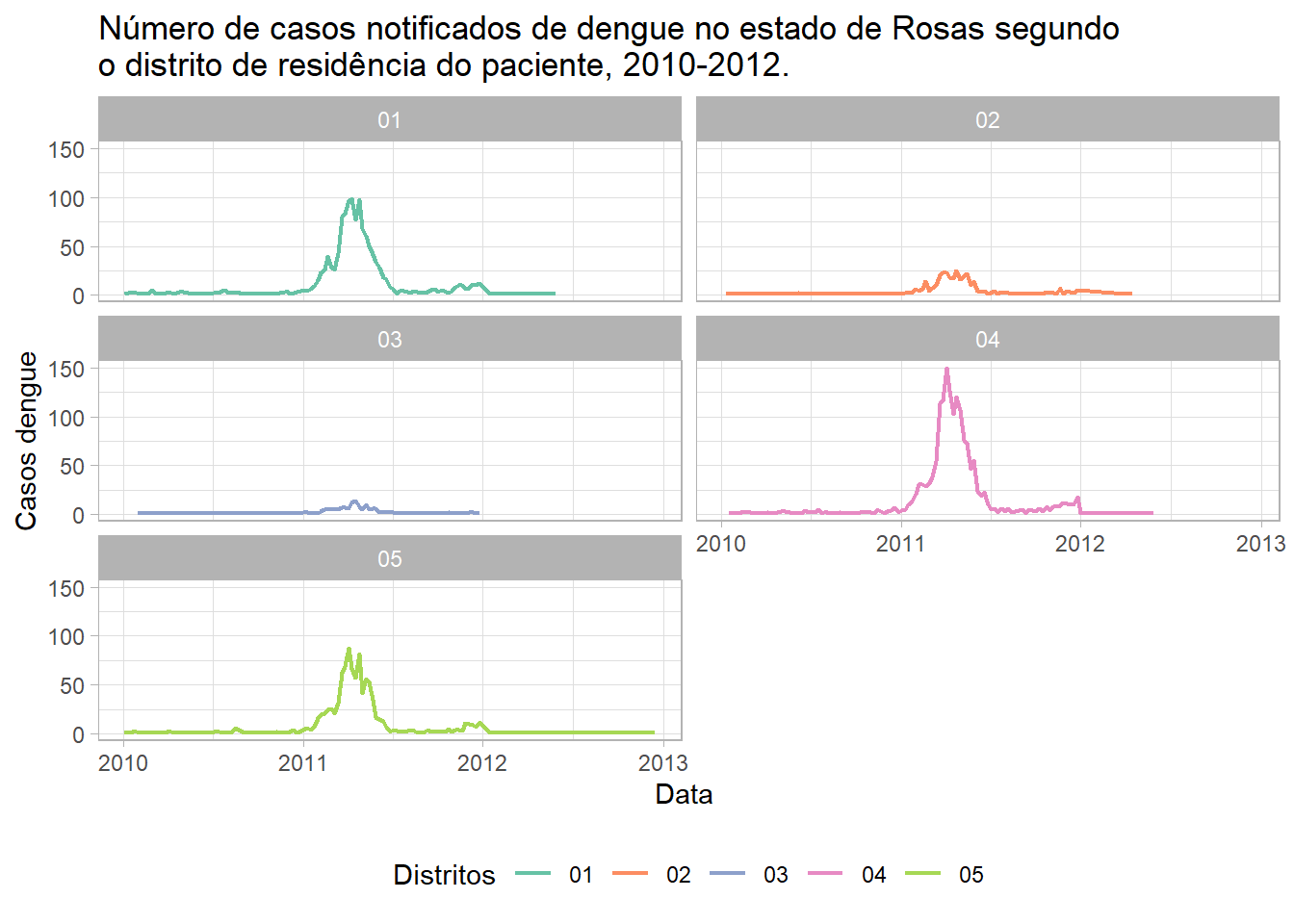
A melhor disposição dos sub-gráficos depende da forma que você deseja representar o evento. Por exemplo, para comparação entre distritos, é de grande importância ter a mesma escala no eixo x. Isso permite que a magnitude do acometimento da doença em cada região seja representada. Mas, considere que o eixo x dos sub-gráficos não esteja representando os distritos administrativos que possuem poucos casos notificados e você decida alterar a escala do eixo x (Figura 26).
Uma das formas de adequar o gráfico é definir o argumento
scales como free_y dentro da função
facet_wrap(). Acompanhe o script a seguir e repita
no seu RStudio, fazendo a comparação entre os
sub-gráficos:
# Criando uma rampa de cores utilizando códigos hexadecimais
cores <- c("#66C2A5" , "#FC8D62" , "#8DA0CB" , "#E78AC3", "#A6D854")
# Criando o objeto gráfico {`painel`}
painel <- ggplot(data =dengue_distritos) +
# Definindo argumentos estéticos com as variáveis usadas em x e em y
# e a variável usada para a cor
aes(x = data_ini, y = casos, color = ID_DISTRIT) +
# Adicionando a geometria de linha e definindo
# a espessusra
geom_line(size = 0.8) +
# Estratificando pelo distrito e definindo
# mudança no eixo y conforme cada sub-gráfico
facet_wrap(~ ID_DISTRIT, ncol = 2, scales = "free_y") +
# Definindo os títulos dos eixos x, y e título do gráfico
labs(
title = "Número de casos notificados de dengue nos distritos administrativos
do estado de Rosas, 2010-2012.",
x = 'Data ',
y = 'Casos dengue') +
# Arrumando a rampa de cores
scale_color_discrete(type = cores, name = 'Distritos') +
# Definindo o tema base
theme_light() +
# Alterando o tema do gráfico
theme(legend.position = "bottom")
# Plotando o objeto `painel`
painelFigura 26: Gráfico de linhas, alterando a escala do eixo x, da distribuição de casos de dengue por distrito de Rosas, entre 2010 e 2013.
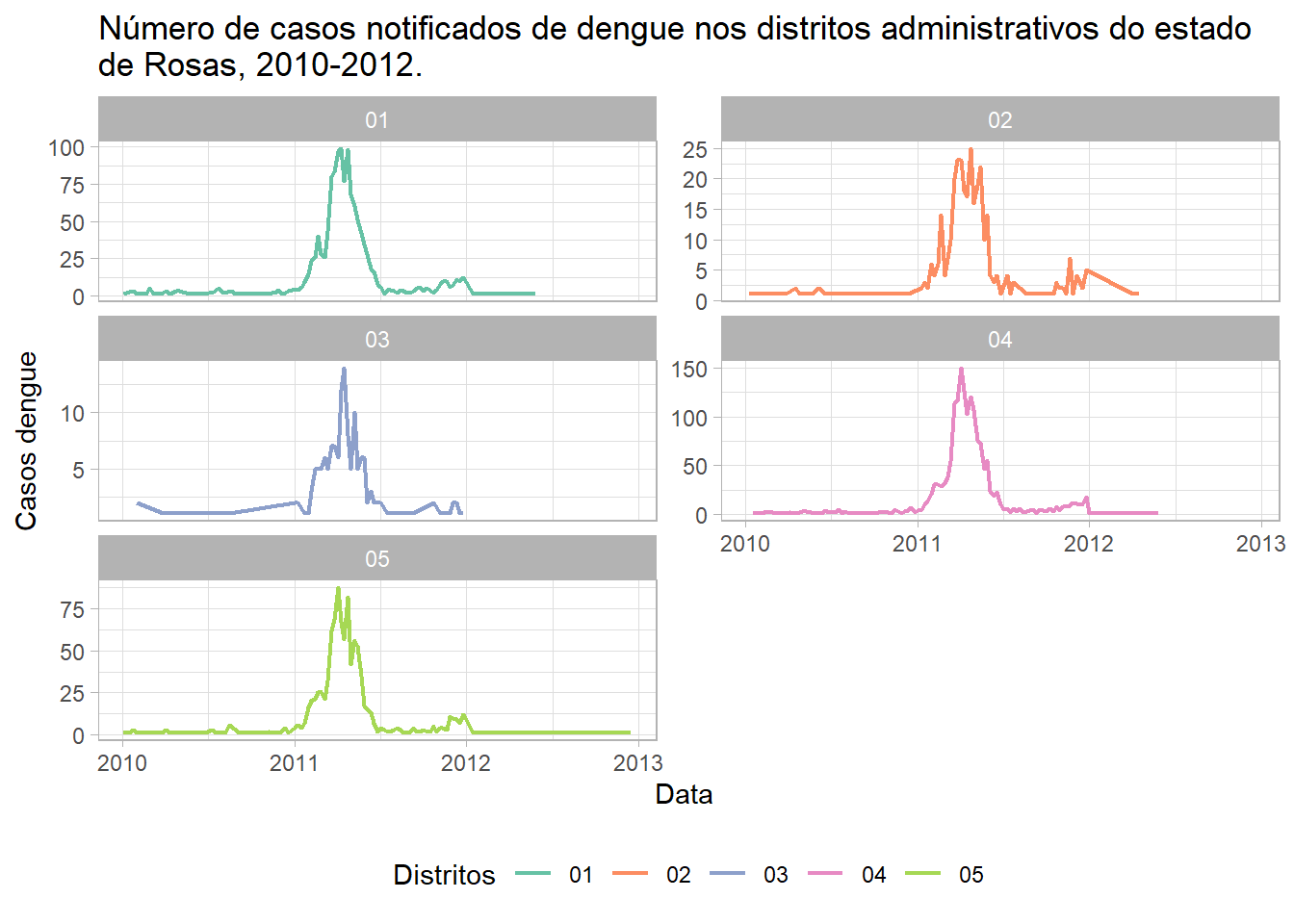
Observe que agora cada sub-gráfico possui um eixo y diferente. Dessa forma, é possível visualizar o comportamento da doença em cada distrito.
Interessante, não é mesmo? Na próxima seção, você aprenderá como salvar estes gráficos no seu computador.


