3. Criando gráficos
O ggplot2 é um pacote do R criado por
Hadley Wickham, que faz parte do universo do tidyverse. Esse
pacote implementa um sistema de gráficos para visualização de dados,
permitindo, inclusive, a criação de gráficos avançados.
O ggplot2 é diferente dos demais pacotes gráficos
existentes no R, por simplificar algumas etapas de produção
gráfica. Em um primeiro contato você pode encontrar dificuldades para
utilizá-lo, mas após se familiarizar com ele é possível obter gráficos
completos, com multicamadas e com excelente qualidade visual de forma
rápida e efetiva. Acredite, por meio da combinação de seus diversos
elementos em poucas linhas de código ele vai revolucionar a sua rotina
de trabalho.
Atualmente, podemos encontrar mais de 70 pacotes do R
que estendem ou implementam funções gráficas da ggplot2.
Clique aqui e conheça alguns
exemplos.
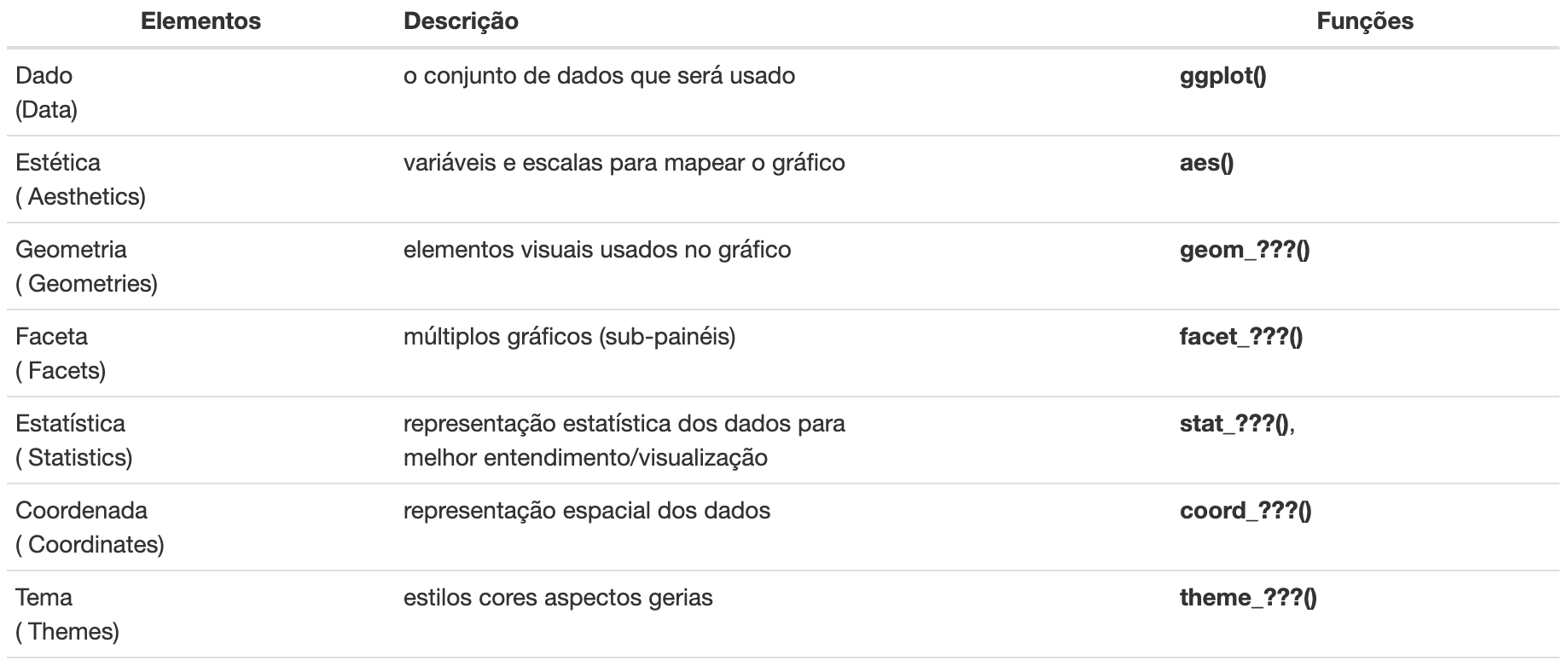
Observe na Tabela 2 a seguir os principais elementos (camadas) que
utilizaremos aplicando funções do ggplot2.
Tabela 2: Principais camadas, sua descrição e as respectivas funções correspondentes do ggplot2.
Atenção
Para fazer um gráfico com o ggplot2 precisamos ao menos
das camadas de Dados, Estética e Geometria.
As diversas camadas são aplicadas concomitantemente quando utilizamos
o operador + (soma). Neste caso, não devemos aplicar o
pipe ( |> ou %>%) .
Atualmente, diversos pacotes atuam como extensão ao
ggplot2, adicionando novas funcionalidades. Neste curso
usaremos alguns desses pacotes complementares.
3.1 Organizando os dados
O primeiro passo para se fazer um gráfico é conhecer o dado a ser apresentado e adequá-lo à visualização proposta. Cada tipo de gráfico exigirá uma organização específica dos dados para se obter o resultado desejado. Como segundo exercício prático neste curso você construirá gráficos que visualizem os dados para avaliação das notificações de dengue do Estado de Rosas (fictício). Para isso você precisará organizar informações que possam apresentar a situação das notificações do estado entre os anos 2007 e 2012 para o secretário de saúde.
Para isso, primeiro precisaremos exportar o nosso banco de dados. Os
dados disponibilizados foram exportados do Sinan Net do Estado de Rosas:
é o arquivo de nome {NINDNET.dbf} que está disponível no
menu lateral “Arquivos” do Ambiente Virtual do curso. Clique e faça o
download para seguirmos com as análises.
Vamos lá?! Carregue e instale os pacotes necessários para produção
gráfica. Escreva as linhas de código a seguir no script do seu
RStudio:
# Esta primeira linha verifica se o pacote está instalado.
# Caso não esteja, irá prosseguir com a instalação
if(!require(foreign)) install.packages("foreign");library(foreign)
if(!require(knitr)) install.packages("knitr");library(knitr)
if(!require(lubridate)) install.packages("lubridate");library(lubridate)
if(!require(aweek)) install.packages("aweek");library(aweek)
if(!require(xts)) install.packages("xts");library(xts)
if(!require(plotly)) install.packages("plotly");library(plotly)
if(!require(ggTimeSeries)) install.packages("ggTimeSeries");library(ggTimeSeries)Em seguida, importaremos a base de dados {NINDINET.DBF},
que se refere às notificações realizadas entre 2007 e 2012 de um estado
fictício chamado Rosas. Para isso, vamos utilizar a função
read.dbf() do pacote foreign. Note que o
argumento as.is marcado como TRUE na função
transforma as variáveis da base de dados no tipo character.
Acompanhe o script a seguir e replique-o em seu
RStudio:
# criando objeto do tipo dataframe (tabela) {`nindi`} com o banco de dados
# {`NINDINET.dbf`}
nindi <- read.dbf('Dados/NINDINET.dbf', as.is = TRUE)Pronto. Com a base importada, vamos filtrar os registros cujo agravo
de notificação foi dengue (campo ID_AGRAVO com registro
“A90”). Além disso, vamos transformar a variável data dos primeiros
sintomas (DT_SIN_PRI), armazenada como character,
para date utilizando a função ymd do pacote
lubridate. Essa função reconhece os separadores típicos de
datas e atribui um formato adequado à variável.
A partir da variável DT_SIN_PRI, vamos criar três novas
variáveis também utilizando funções do pacote lubridate.
São elas:
epiweek(): retorna a semana epidemiológica a partir de uma variável do tipo data.epiyear(): retorna o ano conforme o calendário epidemiológico.month(): retorna o mês referente ao calendário normal.
Também será necessário alterar o tipo da variável NU_ANO
para o tipo numeric. Veja a seguir o script completo e
replique-o em seu RStudio:
# Criando a tabela {`dengue`}
dengue <- nindi |>
# Filtrando os registros de casos de dengue (CID = A90)
filter(ID_AGRAVO == 'A90') |>
# Criando novas colunas
mutate(
# Transformando a variável `DT_SIN_PRI` para data
DT_SIN_PRI = ymd(DT_SIN_PRI),
# Criando uma nova coluna chamada 'sem_epi', referente à semana
# epidemiológica dos primeiros sintomas
sem_epi = epiweek(DT_SIN_PRI),
# Criando uma nova coluna chamada 'ano_epi', referente ao ano epidemiológico
# dos primeiros sintomas
ano_epi = epiyear(DT_SIN_PRI),
# Criando uma nova coluna chamada 'mes', referente ao mês dos primeiros sintomas
mes = month(DT_SIN_PRI),
# Transformando a coluna `NU_ANO` no tipo numérico
NU_ANO = as.numeric(NU_ANO)
)Pronto já temos os dados necessário para criar as nossas visualizações de dados (DataViz).


