6. Avaliando a distribuição dos dados
Já estudamos que todos os cálculos apresentados podem ser feitos com
comandos específicos no R. Alguns destes cálculos podem ser
obtidos com apenas um comando utilizando a função
summary(). Ela é muito utilizada, pois
automatiza a visão geral das variáveis do banco de dados analisado
permitindo ao profissional de vigilância conhecer as medidas resumo de
uma só vez, ou seja, agiliza o trabalho.
Agora, em todas as avaliações que você fizer da distribuição dos
dados analisados, o comando summary() se tornará um
argumento essencial para análise. Ele resulta em uma espécie de
“sumarização” de cada variável conforme seu tipo.
Observe no script abaixo a avaliação que faremos da nossa
tabela {dados}, oriunda do banco de dados
{NINDINET.dbf} disponível no menu lateral “Arquivos”, do
módulo, vamos analisar apenas as variavéis: ID_AGRAVO,
DT_NOTIFIC, DT_SIN_PRI,
IDADE_ANOS e CS_SEXO. As variáveis numéricas
serão apresentadas segundo as métricas mínimo, máximo, média, mediana,
primeiro e terceiro quartis. Entretanto, lembre-se que no caso das
variáveis do tipo fator (fct), o argumento
summary() retornará apenas a frequência de cada
categoria.
Acompanhe os script abaixo com três exemplos para praticar
junto ao seu RStudio:
- No primeiro exemplo selecionamos algumas variáveis utilizadas anteriormente, mas sem agrupar ou filtrar agravos.
- No segundo exemplo adicionamos ao filtro, a seleção dos registros notificados de dengue.
- E, por fim no terceiro exemplo filtramos apenas os casos notificados por hepatite viral.
Vamos lá!
Observe o output do código abaixo quando não filtramos um agravo:
# 1º exemplo: sem utilização de filtro pelo agravo
dados |>
# criando uma nova coluna chamada IDADE_ANOS com a função mutate() e, nela,
# calculando a idade em anos. Primeiro, fazendo a diferença em dias entre
# data de primeiros sintomas e data de notificação e transformando em
# número inteiro com a função as.integer(), seguido da divisão por 365.25,
# e, no final, arredondamento para o menor número inteiro com uso da função
# floor()
mutate(IDADE_ANOS = floor(as.integer(DT_SIN_PRI - DT_NASC) / 365.25)) |>
# selecionando as colunas ID_AGRAVO, DT_NOTIFIC, DT_SIN_PRI, IDADE_ANOS
# e CS_SEXO
select(ID_AGRAVO, DT_NOTIFIC, DT_SIN_PRI, IDADE_ANOS, CS_SEXO) |>
# sumarizando o banco utilizando a função summary()
summary()#> ID_AGRAVO DT_NOTIFIC DT_SIN_PRI IDADE_ANOS
#> A90 :12781 Min. :2007-01-01 Min. :1939-04-24 Min. : 0.00
#> W64 : 5438 1st Qu.:2008-03-28 1st Qu.:2008-03-17 1st Qu.: 15.00
#> A169 : 2347 Median :2009-06-18 Median :2009-04-23 Median : 28.00
#> X58 : 1606 Mean :2009-09-24 Mean :2009-08-01 Mean : 30.26
#> B19 : 955 3rd Qu.:2011-04-20 3rd Qu.:2011-04-09 3rd Qu.: 44.00
#> B24 : 675 Max. :2012-12-30 Max. :2012-12-29 Max. :101.00
#> (Other): 3819 NA's :4304
#> CS_SEXO
#> F:13567
#> I: 56
#> M:13998
#>
#>
#>
#> Perceba que no primeiro exemplo acima, a tabela visualizada apresenta
as variáveis ID_AGRAVO e CS_SEXO com suas
respectivas frequências e as variáveis DT_NOTIFIC,
DT_SIN_PRI e IDADE_ANOS com as estatísticas
descritivas básicas (mínimo, 1º quartil, mediana, média, 3º quartil e
máxima).
Agora, acompanhe o output do código abaixo quando filtramos
os registros notificados com código CID10 = A90, replique os passos em
seu RStudio:
# 2º exemplo: filtro pelo agravo dengue ("A90")
dados |>
# filtrando os agravos de dengue (código "A90") com a função filter()
filter(ID_AGRAVO == "A90") |>
# utilizando a função mutate() para modificar a coluna ID_AGRAVO, removendo as
# categorias (levels) em branco após o filtro usando a função droplevels()
mutate(ID_AGRAVO = droplevels(ID_AGRAVO)) |>
# criando uma nova coluna chamada IDADE_ANOS com a função mutate() e, nela,
# calculando a idade em anos. Primeiro, fazendo a diferença em dias entre
# data de primeiros sintomas e data de notificação e transformando em
# número inteiro com a função as.integer(), seguido da divisão por 365.25,
# e, no final, arredondamento para o menor número inteiro com uso da função
# floor()
mutate(IDADE_ANOS = floor(as.integer(DT_SIN_PRI - DT_NASC) / 365.25)) |>
# selecionando as colunas ID_AGRAVO, DT_NOTIFIC, DT_SIN_PRI, IDADE_ANOS
# e CS_SEXO
select(ID_AGRAVO, DT_NOTIFIC, DT_SIN_PRI, IDADE_ANOS, CS_SEXO) |>
# sumarizando o banco utilizando a função summary()
summary()#> ID_AGRAVO DT_NOTIFIC DT_SIN_PRI IDADE_ANOS CS_SEXO
#> A90:12781 Min. :2007-01-03 Min. :1993-06-23 Min. : 0.00 F:6827
#> 1st Qu.:2008-03-13 1st Qu.:2008-03-06 1st Qu.:12.00 I: 11
#> Median :2008-04-20 Median :2008-04-11 Median :24.00 M:5943
#> Mean :2009-04-03 Mean :2009-03-24 Mean :26.93
#> 3rd Qu.:2011-03-31 3rd Qu.:2011-03-27 3rd Qu.:39.00
#> Max. :2012-12-23 Max. :2012-12-21 Max. :98.00
#> NA's :3364Você conseguiu executar? A tabela apresentada em seu
Rtudio no painel console, deve ser igual
ao que visualizamos aqui. Perceba que estamos analisando agora as
estatísticas descritivas básicas dos casos de Dengue notificados em
Rosas:
- Temos 12.781 casos notificados.
- A menor data de notificação foi em 03/01/2007 e a maioria destes casos foram notificados em 20/04/2008.
- A última data de início de sintomas registrada no sistema foi em 21/12/2012, mas a média dos casos foi notificada em 24/03/2009.
- Quanto as idades dos casos, a média (26,9 anos) e a mediana (24 anos), sendo 98 anos a maior idade entre os notificados, e 3.364 dos casos não tiveram a idade relatada.
- A maioria dos notificados eram do sexo feminino (6.827 casos).
Agora vamos praticar com um último exemplo. Observe o output
do código abaixo em que filtraremos os registros notificados com código
CID10 = B19, replique em seu RStudio:
# 3º exemplo: filtro pelo agravo hepatite viral ("B19")
dados |>
# filtrando os agravos de hepatite viral (código "B19") com a função filter()
filter(ID_AGRAVO == "B19") |>
# utilizando a função mutate() para modificar a coluna ID_AGRAVO, removendo as
# categorias (levels) em branco após o filtro usando a função droplevels()
mutate(ID_AGRAVO = droplevels(ID_AGRAVO)) |>
# criando uma nova coluna chamada IDADE_ANOS com a função mutate() e, nela,
# calculando a idade em anos. Primeiro, fazendo a diferença em dias entre
# data de primeiros sintomas e data de notificação e transformando em
# número inteiro com a função as.integer(), seguido da divisão por 365.25,
# e, no final, arredondamento para o menor número inteiro com uso da função
# floor()
mutate(IDADE_ANOS = floor(as.integer(DT_SIN_PRI - DT_NASC) / 365.25)) |>
# selecionando as colunas ID_AGRAVO, DT_NOTIFIC, DT_SIN_PRI, IDADE_ANOS
# e CS_SEXO
select(ID_AGRAVO, DT_NOTIFIC, DT_SIN_PRI, IDADE_ANOS, CS_SEXO) |>
# sumarizando o banco utilizando a função summary()
summary()#> ID_AGRAVO DT_NOTIFIC DT_SIN_PRI IDADE_ANOS CS_SEXO
#> B19:955 Min. :2007-01-04 Min. :1951-08-13 Min. : 0.00 F:418
#> 1st Qu.:2008-11-09 1st Qu.:2008-06-25 1st Qu.:31.00 I: 0
#> Median :2009-10-23 Median :2009-07-14 Median :45.00 M:537
#> Mean :2009-12-25 Mean :2009-07-01 Mean :42.92
#> 3rd Qu.:2011-04-12 3rd Qu.:2011-01-11 3rd Qu.:56.00
#> Max. :2012-12-27 Max. :2012-12-20 Max. :89.00
#> NA's :55Observe que visualizamos a tabela com as estatísticas descritivas básicas dos casos notificados com hepatites virais do Estado de Rosas. Tente descrever os dados que visualiza, será um bom exercício para memorização e aprendizagem!
Se houver uma grande quantidade de variáveis contidas na base de
dados analisada, a visualização dos outputs (resultados) da
função summary() poderá ficar comprometida.
Nestes casos, voê pode utilizar a função de forma individualizada, ou seja, escrevemos os comandos para cada variável de interesse da seguinte forma:
# utilizando a função summary() para
# visualizar a coluna CS_SEXO da tabela {`dados`}
summary(dados$CS_SEXO)#> F I M
#> 13567 56 13998Você também poderá utilizar o pacote summarytools para
melhorar a visualização do comando executado. Este pacote possui a
função dfSummary(). Ela apresenta um resumo dos dados em
forma de tabela, contendo os nomes, tipos, frequência, resumo numérico,
gráfico histograma, a porcentagem de registros em branco e registros
preenchidos (válidos). Isso torna a leitura dos resultados mais
agradável.
Para utilizá-la você deve utilizar o argumento view(),
pois ele permitirá a visualização do relatório elaborado pelo comando na
aba Viewer do RStudio, no formato de
relatório em HTML (página da web). A função também é simples, tendo como
argumento apenas a base de dados.
Acompanhe o script abaixo e replique em seu
RStudio:
dados |>
# criando uma nova coluna chamada IDADE_ANOS com a função mutate() e, nela,
# calculando a idade em anos. Primeiro, fazendo a diferença em dias entre
# data de primeiros sintomas e data de notificação e transformando em
# número inteiro com a função as.integer(), seguido da divisão por 365.25,
# e, no final, arredondamento para o menor número inteiro com uso da função
# floor()
mutate(IDADE_ANOS = floor(as.integer(DT_SIN_PRI - DT_NASC) / 365.25)) |>
# selecionando as colunas ID_AGRAVO, DT_NOTIFIC, DT_SIN_PRI, IDADE_ANOS
# e CS_SEXO
select(ID_AGRAVO, DT_NOTIFIC, DT_SIN_PRI, IDADE_ANOS, CS_SEXO) |>
# criando o sumário com a função dfSummary()
dfSummary() |>
# visualizando o relatório em formato HTML com função view()
view()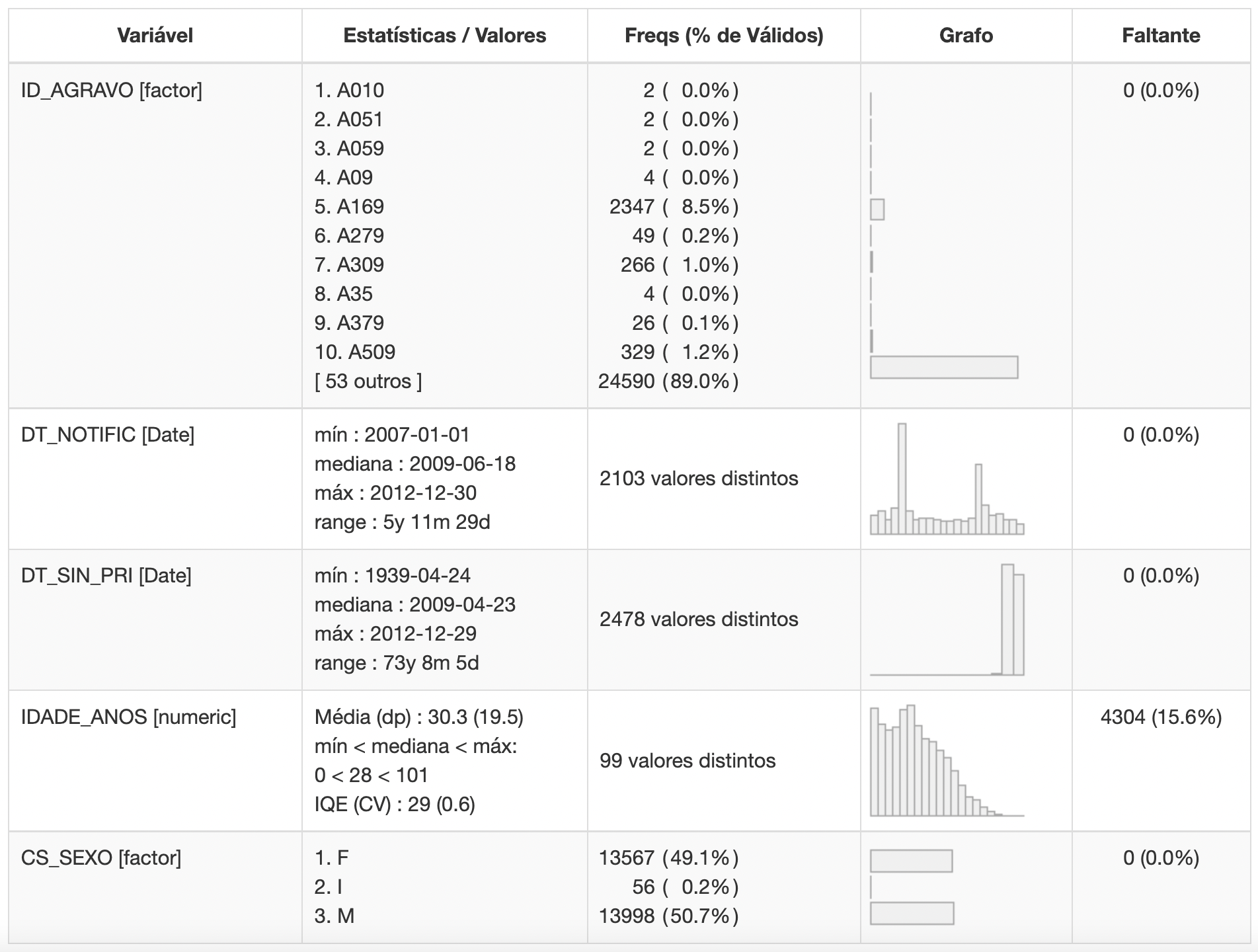
O relatório gerado é apresentado no formato HTML, e pode ser
visualizado em um navegador de internet, clicando no ícone do painel
Viewer conforme a Figura 4:
Figura 4: Tela de visualização da aba Viewer.
Observe que a tabela acima apresentou a visualização das seguintes análises em cada coluna:
- Variável: indicando qual é a variável estudada;
- Estatísticas ou Valores: indicando a frequência absoluta de cada registro da variável estudada,
- Freqs (% de válidos): indicando a frequência
absoluta e relativa de cada registro da variável estudada,
- Graph: visualização gráfica da distribuição dos dados da variável estudada,
- Valid: indicando a frequência absoluta e relativa dos valores considerados validos da variável estudada e
- Faltante: indicando a frequência absoluta e relativa dos valores considerados nulos ou em branco da variável estudada.
O que você achou depois de executar os comandos? Esta visualização é mais agradável, não é mesmo? Pratique com os bancos de dados que você utiliza na vigilância.
Para geração de tabelas que possuam variáveis calculadas incluindo
cálculos de porcentagens e médias, o pacote gtsummary é
muito útil. Afinal, com poucas linhas de comando, será possível gerar
uma bela tabela para análises. Veja e acompanhe os passos abaixo:
Primeiro, filtramos com a função
filter()os casos notificados de dengue e hepatite virais, notificados no Estado de Rosas.Segundo, eliminamos os valores nulos com a função
droplevels().No terceiro passo, selecionamos com a função
select()as variável (CS_SEXO) que utilizaremos para a análise dos agravos.Por fim, utilizamos a função
tbl_summary(), incluindo o argumentoby = CS_SEXOque permitirá que os agravos selecionados no passo 1, sejam cruzados com a variável sexo.
Assim, teremos como resultado uma tabela com estatísticas descritivas já calculadas. Observe o script abaixo:
dados |>
# filtrando os agravos de dengue (código "A90") e
# hepatite viral
# (código "B19") com a função filter()
filter(ID_AGRAVO %in% c("A90", "B19")) |>
# utilizando a função mutate() para modificar a
# coluna ID_AGRAVO, removendo as
# categorias (levels) em branco após o filtro usando
# a função droplevels()
mutate(ID_AGRAVO = droplevels(ID_AGRAVO)) |>
# selecionando apenas as colunas de agravo (ID_AGRAVO)
# e sexo (CS_SEXO)
select(ID_AGRAVO, CS_SEXO) |>
# gerando uma tabela com resumo das informações
# cruzando
# do agravo (ID_AGRAVO) pelo sexo (CS_SEXO)
tbl_summary(by = CS_SEXO)
A tabela gerada poderá ser visualizada no painel
Viewer do seu RStudio (ela não será
visualizada no painel Console). Agora vamos interpretar os
resultados:
Né o número de valores totais por categorias de sexo, está localizado no cabeçalho da tabela.né o número total de casos por agravo, está sinalizado com¹no rodapé da tabela.(%)é a porcentagem total por agravo, está sinalizado com¹no rodapé da tabela.
Para a visualização dessas informações apresentadas na tabela no idioma português, precisamos definir o idioma português como padrão. Siga o script com o comando abaixo configure esse padrão.
# definindo o idioma das tabelas geradas em pacote
# "gtsummary" como português ("pt")
theme_gtsummary_language("pt")Próximo módulo
Parabéns, você chegou ao final do nosso quarto módulo! Agora você já
conhece todas as funções básicas para manipular banco de dados e fazer
cálculos epidemiológicos utilizando a linguagem R. No
próximo módulo você irá colocar em prática cálculos de indicadores de
saúde necessários para estabelecer rotinas de trabalho para o seu dia a
dia na vigilância em saúde.
Até lá.


