3. Construindo o diagrama de controle de Hepatite A
É possível construir um diagrama de controle para qualquer doença que tenha seus casos suspeitos notificados com registro do início dos sintomas. Nesta subseção vamos analisar a situação da Hepatite A no município São Paulo (SP), utilizando como referência a avaliação do ano de 2017.
A vigilância das Hepatites Virais visa monitorar os casos da doença no território, recomendando ações que controlem e previnam em tempo oportuno as infecções. Dessa forma, analisar a ocorrência de casos ou surtos é estratégico para mitigar a doença. Aqui utilizaremos um diagrama de controle para avaliar um dos agravos de rotina para a vigilância em saúde.
Vamos lá! Considere que a coordenadoria de vigilância epidemiológica em saúde do Estado de São Paulo recebeu um alerta de aumento de casos de Hepatite A no início de março de 2017 no município de SP. Rapidamente, a equipe realizou a exportação dos dados do Sistema de Informação de Agravos de Notificação (Sinan Net) para avaliação e enviou os dados para seu e-mail.
Aqui neste exemplo vamos supor que você é membro da equipe técnica municipal de vigilância e agravos de doenças transmissíveis.
Você recebeu dois arquivos do tipo .dbf:
- o primeiro contendo os casos confirmados de Hepatite A entre os anos de 2007 e 2016, e
- e uma segunda exportação do Sinan Net apenas com os casos confirmados de Hepatite A de janeiro e fevereiro de 2017.
Agora como profissional da vigilância epidemiológica você iniciará a
avaliação utilizando a linguagem de programação R!
Todos os bancos de dados utilizados para análises neste módulo se
encontram no menu lateral “Arquivos” do curso. Lembre-se de fazer o
download do material do curso diretamente do Ambiente Virtual
de Aprendizagem do curso e arquivá-los em um diretório que deverá
indicar para que o R o localize.
Primeiro, importaremos os dois arquivos para o ambiente do
RStudio. Observe o código abaixo e replique-o em seu
computador:
# criando objeto do tipo dataframe (tabela) {sinan_hep_sp_2007_2016} com o
# banco de dados {sinan_hep_sp_2007_2016.dbf}
sinan_hep_sp_2007_2016 <- read.dbf("Dados/sinan_hep_sp_2007_2016.dbf", as.is = TRUE)
# criando objeto do tipo dataframe (tabela) {sinan_hep_sp_fev_2017} com o
# banco de dados {sinan_hep_sp_fev_2017.dbf}
sinan_hep_sp_fev_2017 <- read.dbf("Dados/sinan_hep_sp_fev_2017.dbf", as.is = TRUE)Pronto, seus dados já podem ser utilizados pelo R! Agora
será necessário calcular a frequência de casos por semana epidemiológica
e ano dos primeiros sintomas. Para isso, considere criar as variáveis
correspondentes utilizando as funções do pacote lubridate
para transformar datas: epiweek() e year().
Ambas as funções possuem apenas um argumento obrigatório, que é a
variável no formato date (data). Você precisará utilizar a
função mutate() do pacote dplyr para realizar
as transformações.
Acompanhe o script abaixo e replique-o em seu
RStudio:
# Criando a tabela {`sinan_hep_sp_cont_07_16`}
sinan_hep_sp_cont_07_16 <- sinan_hep_sp_2007_2016 |>
# Utilizando a função `mutate()` para criar as novas colunas
mutate(
# Criando uma nova coluna chamada 'sem_epi', referente à semana
# epidemiológica dos primeiros sintomas
sem_epi = epiweek(x = DT_SIN_PRI),
# Criando uma nova coluna chamada 'ano', referente ao ano dos primeiros sintomas
ano = year(x = DT_SIN_PRI)) |>
# Contando a frequência de notificações por ano e semana epidemiológica
count(ano, sem_epi)Agora que você já criou as colunas, visualize as primeiras linhas
desta nova tabela utilizando a função head(). A nova tabela
{sinan_hep_sp_cont_07_16} deverá possuir três colunas:
ano, sem_epi e n. Reproduza o
script abaixo em seu computador:
head(sinan_hep_sp_cont_07_16)#> ano sem_epi n
#> 1 2007 1 2
#> 2 2007 2 2
#> 3 2007 3 7
#> 4 2007 4 2
#> 5 2007 5 7
#> 6 2007 6 3Pronto. Agora você deverá aplicar os mesmos passos à base de dados
{sinan_hep_sp_fev_2017}. Não se preocupe, o código abaixo é
praticamente o mesmo do mostrado anteriormente, apenas com a modificação
da base de dados. Acompanhe abaixo o script e repita em seu
RStudio:
# Criando o objeto {`sinan_hep_sp_cont_fev17`} e
# realizando a contagem dos casos segundo a
# semana epidemiológica e ano dos primeiros sintomas
sinan_hep_sp_cont_fev17 <- sinan_hep_sp_fev_2017 |>
mutate(sem_epi = epiweek(DT_SIN_PRI),
ano = year(DT_SIN_PRI)) |>
count(ano, sem_epi)Pronto! Já temos duas tabelas com as mesmas variáveis:
{sinan_hep_sp_cont_07_16} e
{sinan_hep_sp_cont_fev17}. Agora, vamos visualizar a
variação dos casos confirmados de Hepatite A do Estado de São Paulo
entre os anos 2007 e 2016. O objetivo dessa análise é identificar em
quais anos ocorreram surtos ou epidemias. Queremos, portanto,
identificar os anos epidêmicos para, eventualmente, retirá-los da
estatística necessária para o diagrama de controle.
Para avaliação da distribuição de dados e análise dos anos epidêmicos
vamos plotar um gráfico do tipo boxplot utilizando a função
boxplot(). Com ela realizaremos o cruzamento da variável
n (o número de casos confirmados de Hepatite A entre 2007 e
2016) com a variável ano (anos do início dos sintomas).
Para ter algum parâmetro de análise vamos utilizar a mediana geral dos casos (linha laranja) para visualizar aqueles anos que se apresentaram com número maior de casos.
Acompanhe o script abaixo:
# Definindo a mediana geral de casos confirmados entre 2007 e 2016
mediana_geral <- median(sinan_hep_sp_cont_07_16$n)
# utilizando a função `boxplot()` para criar o gráfico
boxplot(
# Definindo o cruzamento número de casos por ano
# Aqui utilizamos o símbolo "~" para sinalizar o cruzamento das variáveis
sinan_hep_sp_cont_07_16$n ~ sinan_hep_sp_cont_07_16$ano,
# Definindo os títulos dos eixos x e y
ylab = 'Número de casos confirmados de Hepatite A',
xlab = 'Ano dos primeiros sintomas',
# Definindo o título do boxplot
main = 'Número de casos confirmados de Hepatite A em São Paulo/SP entre 2007-2016'
)
# Criando linhas de análise
abline(
h = mediana_geral,
lty = 2,
lwd = 2,
col = "red"
)Figura 7: Gráfico boxplot da distribuição de casos de HepatiteA, por ano, em São Paulo/SP.
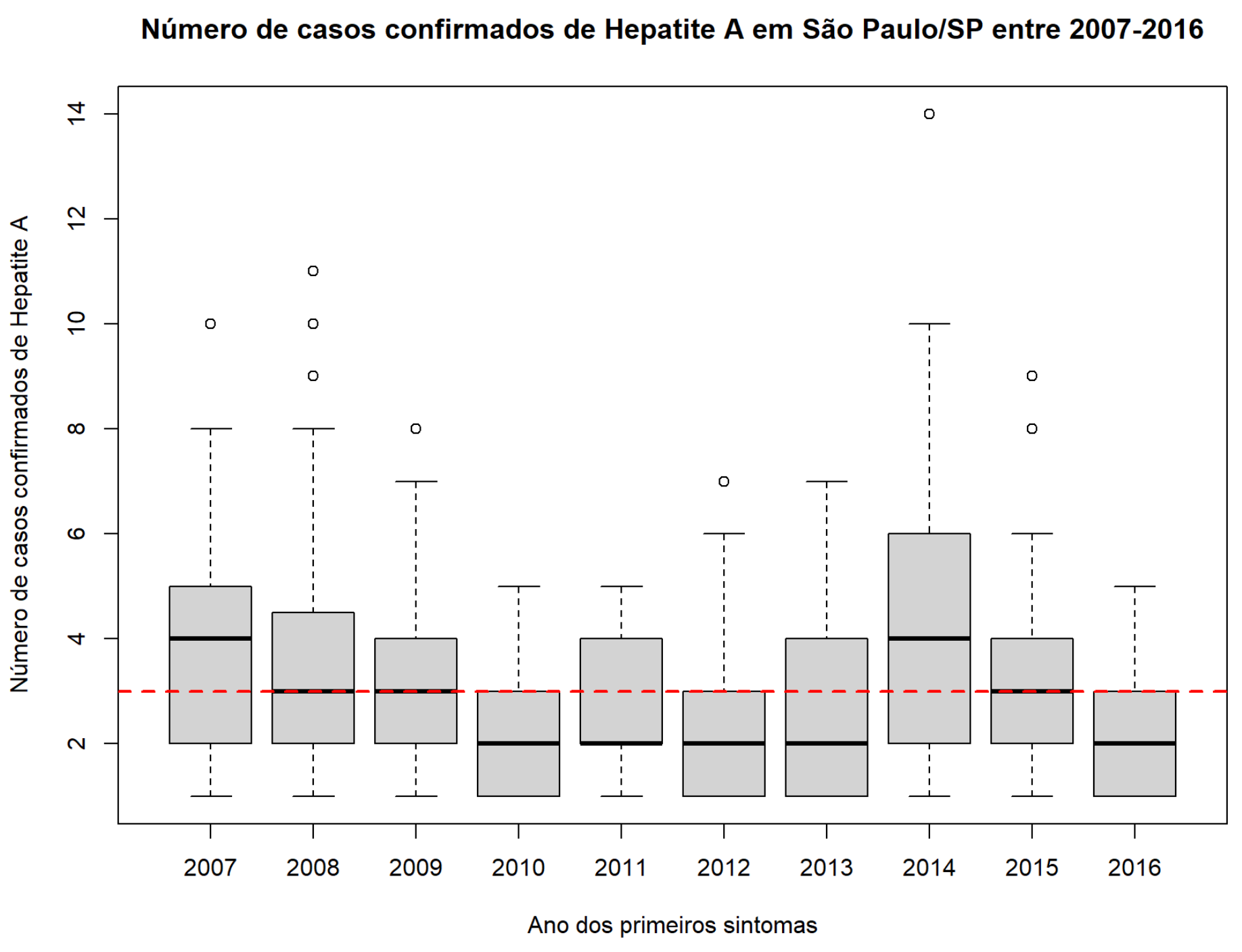
Após plotar o gráfico da Figura 7, você visualizará o boxplot. Agora vamos analisá-lo juntos:
- cada retângulo (box) representa a variação do número de casos dentro de cada ano,
- a linha mais grossa dentro do retângulo (box) representa a mediana de casos de cada ano,
- a linha reta tracejada na cor preta acima do retângulo (box) representa o limite superior de cada ano,
- os círculos ao alto (outliers) representam valores muito altos, além dos limites em cada ano, e
- a linha tracejada na cor vermelha representa a mediana geral de todos os anos (2007 a 2016).
Percebemos que 2007 e 2014 registraram número de casos de Hepatite A maior, pois suas medianas (linha mais grossa dentro do retângulo) são mais altas que os demais boxs. Além disso, é possível observar que 2014 registrou um pico de casos, se destacando na série. Já sabemos, portanto, que 2007 e 2014 apresentaram aumento nos casos de Hepatite A e por isso serão considerados para análise anos epidêmicos.
Agora, criamos um objeto que armazenará os anos que definimos
como não epidêmicos para Hepatite A: 2008, 2009, 2010, 2011,
2012, 2013, 2015 e 2016. Perceba no script abaixo que
utilizamos uma sequência de 2006 até 2013 com o auxílio do símbolo
: (dois pontos) e adicionamos os anos de 2015 e de 2016
separados por , (virgula). Com isso, escreveremos o código
de forma mais curta.
Replique o código abaixo e execute os comandos em seu
RStudio:
# criando o objeto {nao_epidemic} para armazenar os anos não epidêmicos
nao_epidemic <- c(2008:2013, 2015, 2016)Pronto, já temos armazenados os anos não epidêmicos no objeto
{nao_epidemic} para nossa avaliação. Vamos agora filtrar os
casos confirmados de Hepatite A cujo ano dos primeiros sintomas estão
contidos nestes anos. Execute os passos a seguir:
- crie um novo objeto do tipo data.frame que armazenará os
dados: {
hep_stat}, - utilize a função
filter()para os anos não epidêmicos, - agrupe os dados pela semana epidemiológica com a função
group_by(), e - realize o cálculo das métricas necessárias para construção gráfica
do diagrama de controle: média, desvio padrão e limites superior e
inferior. Utilize para isso a função
summarise()e as funçõesmean(),sd()e as fórmulas dos limites.
Acompanhe abaixo o script e reproduza-o em seu
RStudio:
# Criando o objeto {`hep_stat`}
hep_stat <- sinan_hep_sp_cont_07_16 |>
# Filtrando os anos contidos no grupo de anos não epidêmicos
filter(ano %in% nao_epidemic) |>
# Agrupando os dados pela semana epidemiológica
group_by(sem_epi) |>
# Criando medidas-resumo e limites superior e inferior
summarise(
media = mean(n, na.rm = TRUE),
desvio = sd(n, na.rm = TRUE) ,
sup = media + 2 * desvio,
inf = media - 2 * desvio
)Feito! Agora chegou a hora de criar a visualização gráfica para
avaliar o número de casos de Hepatite A confirmados até fevereiro de
2017, comparando-o aos demais anos. Essa avaliação é muito similar à
realizada para construir o diagrama de controle de dengue. Assim será
utilizado novamente o pacote ggplot2. Vamos lá. Siga o
passo a passo abaixo:
- Salve o gráfico inicial em um novo objeto:
{
grafico_base}. Necessitaremos dele como base estrutural para toda a análise, evitando assim retrabalho. - Adicione uma legenda ao gráfico tornando-o autoexplicativo. Para
isso, use a função
aes()aninhada à funçãogeom_line()e o seu argumentocolor. - Personalize ainda mais o gráfico com a função
scale_color_manual()e use os seguintes argumentos:
Values, que define os valores e suas respectivas cores.Labels, que define o rótulo de dados para a legenda.Name, que define o título da legenda que, nesse caso, ficará em branco (" ").
Colocaremos em prática essas três etapas e construiremos o nosso
diagrama de controle. Observe o script abaixo e replique-o com
muita atenção em seu RStudio:
# Criando um novo objeto {`grafico_base`}
grafico_base <- ggplot(data = hep_stat) +
# Definindo as variáveis usadas no eixo x e em y do gráfico
aes(x = sem_epi, y = media) +
# Adicionando uma geometria de linha com largura de 1.2 pixel
# referente ao número médio de casos confirmados.
# Além disso, inserindo um argumento estético para a cor,
# que será convertida na legenda.
geom_line(aes(color = "cor_media_casos"), size = 1.2) +
# Adicionando uma geometria de linha referente ao limite superior.
# Além disso, inserindo um argumento estético para o eixo y e
# para a cor, que será convertida na legenda.
geom_line(aes(y = sup, color = 'cor_limite'), size = 1.2) +
# Arrumando o eixo x, definindo o intervalo de tempo que será utilizado (`breaks`)
# uma sequência de semanas epidemiológicas de 1 a 53
# o argumento `expand` ajuda nesse processo.
scale_x_continuous(breaks = 1:53, expand = c(0, 0)) +
# Definindo os títulos dos eixos x e y
labs(x = '',
y = '',
title = 'Diagrama de controle') +
# Definindo o tema do gráfico
theme_classic() +
# Criando a legenda das linhas
scale_color_manual(
name = "",
values = c('cor_media_casos' = 'darkblue', 'cor_limite' = 'red'),
labels = c("Média de casos confirmados", "Limite superior")
)
#visualizando o gráfico criado
grafico_baseFigura 8: Gráfico da distribuição de casos de Hepatite A, por ano, em São Paulo/SP.
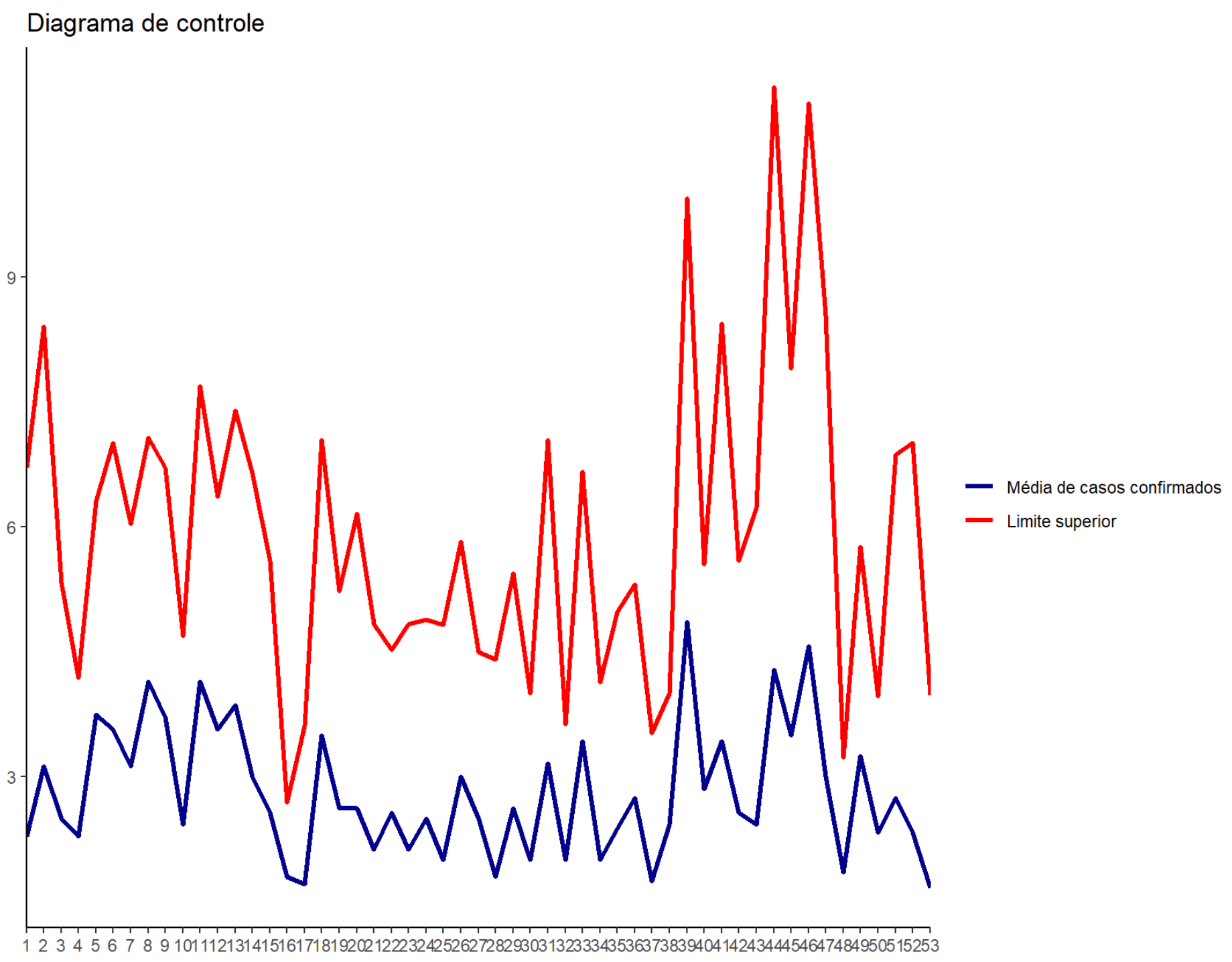
Observe que na Figura 8 mais uma vez existe variação aleatória das medidas calculadas, chamadas em séries temporais de ruídos. Vimos no exemplo anterior que a variação aleatória manifesta-se visualmente na forma de rugosidade nas linhas dos gráficos de séries temporais. O termo ‘ruído’ é efetivamente empregado para referenciar a variação aleatória na análise de séries temporais.
Lembre-se que uma forma de melhorar a visualização gráfica e
evidenciar seus componentes de variação é suavizando as curvas, como
fizemos na avaliação da dengue em Foz do Iguaçu/PR. Assim, aplicaremos
ao gráfico feito acima a função stat_smooth() para avaliar
se há a evidência da variação.
Observe o script abaixo e replique-o em seu
RStudio:
grafico_base_suavizado <- ggplot(data = hep_stat) +
# Definindo as variáveis usadas no eixo x e em y do gráfico
aes(x = sem_epi, y = media) +
# Suavizando a linha referente ao número médio de casos.
# o argumento `size` para definir largura da linha = 1.2 pixel
# O argumento `se` = FALSE desabilita o intervalo de confiança
# e o argumento `span` definindo o valor da suavização
stat_smooth(
aes(color = 'cor_media_casos'),
size = 1.2,
se = FALSE,
span = 0.2
) +
# Suavizando a linha referente ao limite superior
stat_smooth(
aes(y = sup, color = 'cor_limite'),
size = 1.2,
se = FALSE,
span = 0.2
) +
# Arrumando o eixo x, definindo o intervalo de tempo que será utilizado (`breaks`)
# uma sequência de semanas epidemiológicas de 1 a 53
# o argumento `expand` ajuda nesse processo.
scale_x_continuous(breaks = 1:53, expand = c(0, 0)) +
# Definindo os títulos dos eixos x e y
labs(x = '',
y = '',
title = 'Diagrama de controle') +
# Definindo o tema do gráfico
theme_classic() +
# Criando a legenda das linhas
scale_color_manual(
name = "",
values = c('cor_media_casos' = 'darkblue', 'cor_limite' = 'red'),
labels = c("Média suavizada de casos confirmados", "Limite superior suavizado")
)
#visualizando o gráfico criado
grafico_base_suavizado#> `geom_smooth()` using method = 'loess' and formula 'y ~ x'
#> `geom_smooth()` using method = 'loess' and formula 'y ~ x'Figura 9: Gráfico alisado da distribuição de casos de Hepatite A, por ano, em São Paulo/SP.
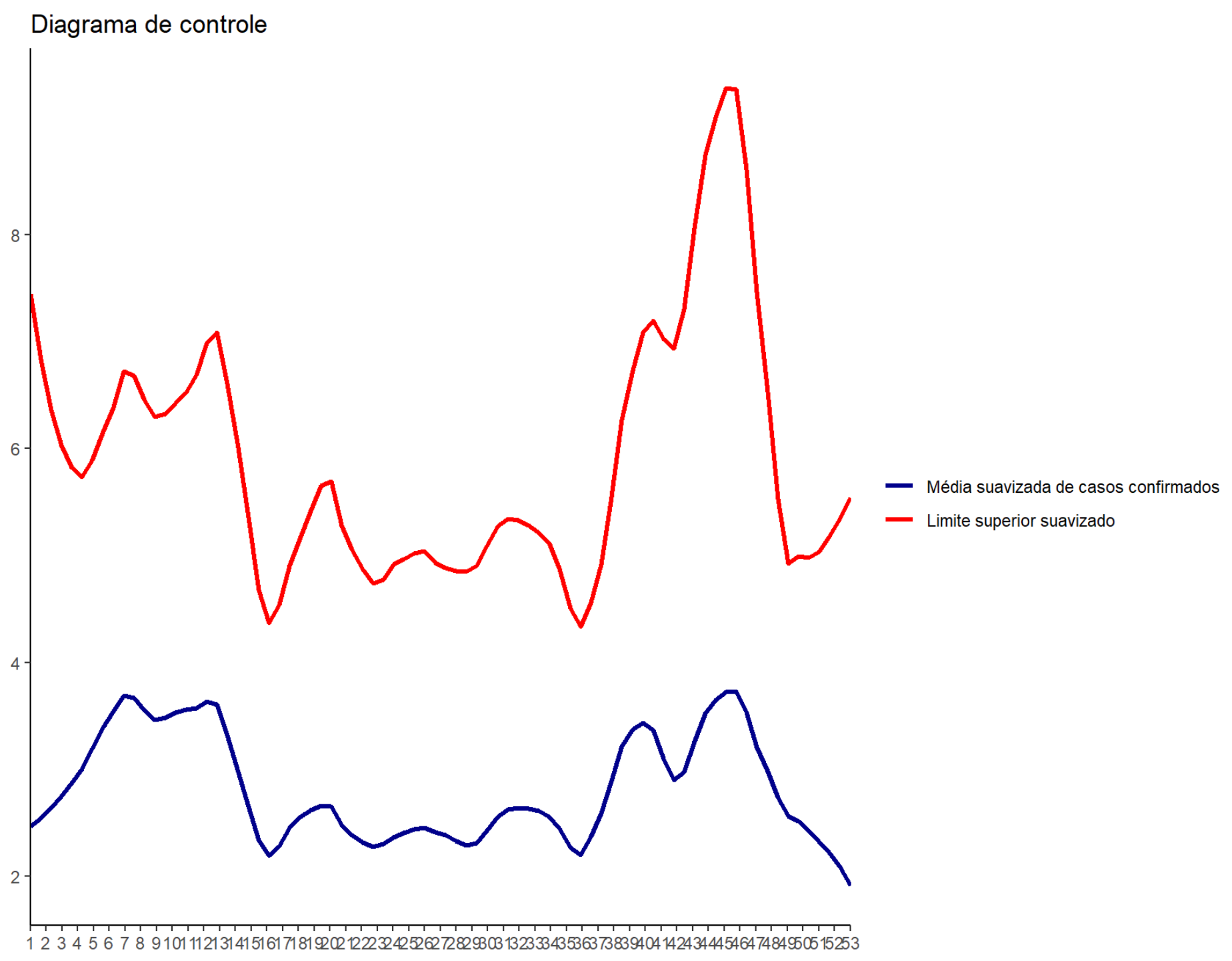
Pronto. Percebeu a diferença na Figura 9? Ao optar pela suavização de curvas é preciso ficar atendo às análises. Em nosso exemplo, além de excluir anos com alto número de casos, também incluímos o desvio padrão para construção da média anual, o que pode mascarar uma epidemia. Não é raro que os surtos epidêmicos ocorram de forma brusca e inesperada.
Ao se analisar uma série temporal, o que diferenciaria um surto ou epidemia de uma variação aleatória? Fique atento, pois epidemias não ocorrem ao acaso e o esforço de analisar as séries temporais vem justamente no sentido de antecipar os problemas e preveni-los.
Por sua magnitude, os surtos epidêmicos não podem ser confundidos com as variações irregulares. Todas e quaisquer mudanças de padrão no acompanhamento de uma doença são importantes e devem ser investigados!
Perceba que o gráfico ainda não contém os dados de 2017, pois é
apenas o gráfico com linhas básicas de análise que você poderá a
qualquer momento incluir ou excluir anos a ele. Agora precisaremos somar
a ele os dados de casos de Hepatite A de 2017. Para isso utilizaremos
apenas uma ação de somar incluindo o operador +(mais) e a
função geom_col do pacote ggplot2.
Acompanhe o passo a passo a seguir para fazer a inclusão de dados de casos de 2017:
- calcularemos a frequência de casos e plotaremos no gráfico como
sendo as barras, ou seja, eixo y (
y = n), - incluiremos uma legenda para o número de casos
(
fill = 'cor_n_casos'), - modificaremos a opacidade das cores do gráfico para 0.4
(
alpha = 0.4), e - incluiremos a legenda das barras utilizando a função
scale_fill_manual().
Observe o script abaixo com atenção e acrescente as
modificações em seu código no RStudio:
# Gráfico criado anteriormente
grafico_base_suavizado +
# Adicionando (+) uma geometria de colunas utilizando a base de dados
# {`sinan_hep_sp_cont_fev17`}
geom_col(data = sinan_hep_sp_cont_fev17,
# O eixo y é definido como a frequência de casos (n).
# As barras serão preenchidas com valor textual para definição da
# legenda.
aes(y = n, fill = 'cor_n_casos'), alpha = 0.4) +
# Definindo a legenda das barras, convertendo o valor textual em um nome de cor
# e definindo o rótulo
scale_fill_manual(
name = "",
values = c('cor_n_casos' = 'deepskyblue'),
labels = "Número de casos até fev/2017"
)#> `geom_smooth()` using method = 'loess' and formula 'y ~ x'
#> `geom_smooth()` using method = 'loess' and formula 'y ~ x'Figura 10: Gráfico do diagrama de controle de Hepatite A, em São Paulo/SP até fevereiro de 2017.

Observe a Figura 10 e perceba que o número de casos nas primeiras semanas do ano de 2017 tem ultrapassado a média dos últimos anos estudados. A semana epidemiológica 4 (entre os dias 22/01/2017 e 28/01/2017) apresentou uma magnitude que ultrapassou o limite esperado. Além disso, outras semanas epidemiológicas estão com o número de casos confirmados acima da média (semanas 6 e 9), mas abaixo do limite superior.
Lembre-se que para uma boa avaliação de situação de saúde será preciso descrever de forma detalhada todos os casos: caracterizando-o em tempo, lugar e pessoa. Será preciso exaurir todas as razões que estão levando ou podem levar a um aumento repentino dos casos de Hepatite A.
Considere agora que você acabou de receber por e-mail os dados
atualizados de Hepatite A incluindo também o mês de março de 2017 no
formato .dbf. Você precisará atualizar o seu diagrama de
controle e enviá-lo novamente às áreas afins! Para isso, você irá
aproveitar as análises realizadas até aqui e apenas incluir dados mais
atualizados. Vamos lá, siga o passo a passo:
- Primeiro, importe os dados para o utilizarmos no
R.
Digite o script abaixo em seu computador:
# Importando o banco de dados {`sinan_hep_sp_mar_2017.dbf`} para o `R`
sinan_hep_sp_mar_2017 <- read.dbf("Dados/sinan_hep_sp_mar_2017.dbf", as.is = TRUE)- Segundo, você deve realiza o cálculo de frequência dos casos de Hepatite A.
Replique o script abaixo em seu RStudio:
# Criando o objeto {`sinan_hep_sp_cont_mar17`} e
# realizando a contagem dos casos segundo a
# semana epidemiológica e ano dos primeiros sintomas
sinan_hep_sp_cont_mar17 <- sinan_hep_sp_mar_2017 |>
mutate(
sem_epi = epiweek(DT_SIN_PRI),
ano = year(DT_SIN_PRI)
) |>
count(ano, sem_epi)- Por fim, sobreponha o gráfico base com os dados atualizados até março de 2017.
Acompanhe os códigos abaixo e reproduza-os em seu
RStudio:
# Gráfico criado anteriormente
grafico_base_suavizado +
# Adicionando uma geometria de colunas utilizando a base de dados
# {`sinan_hep_sp_cont_mar17`}
geom_col(data = sinan_hep_sp_cont_mar17,
aes(y = n, fill = 'cor_n_casos'), alpha = 0.4) +
# Definindo a legenda das barras, convertendo o valor textual em um nome de cor
# e definindo o rótulo
scale_fill_manual(
name = "",
values = c('cor_n_casos' = 'deepskyblue'),
labels = "Número de casos até março/2017"
)#> `geom_smooth()` using method = 'loess' and formula 'y ~ x'
#> `geom_smooth()` using method = 'loess' and formula 'y ~ x'Figura 11: Gráfico do diagrama de controle de Hepatite A, em São Paulo/SP até março de 2017.
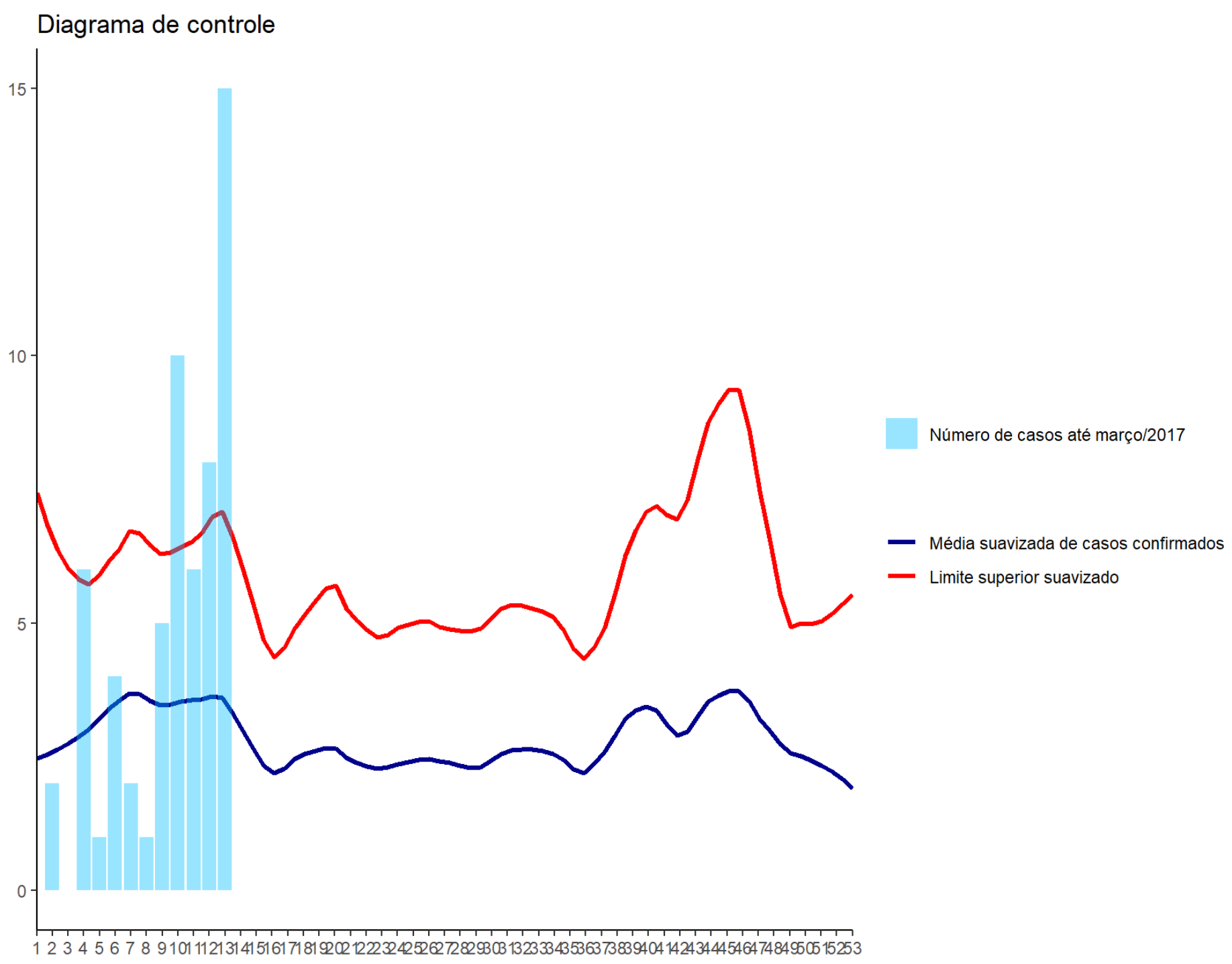
Pronto. Plotando o novo gráfico, conforme Figura 11, foi possível notar que o munícipio de São Paulo apresenta um surto epidêmico. Perceba que há um comportamento irregular do número de casos confirmados de Hepatite A nos três primeiros meses de 2017, apresentando um grave problema que deverá ser enfrentado. O número de casos confirmados de Hepatite A foi, durante todo o mês de março, acima da média e, quase sempre, acima do limite superior esperado.
A linguagem de programação R lhe apoiará como uma
ferramenta poderosa para avaliação de mudanças de cenários
epidemiológicos, oferecendo uma visão rápida da magnitude do problema.
Em São Paulo os casos continuaram aumentando, deflagrando a posteriori
como uma grande epidemia de Hepatite A no estado.
Observe abaixo o script para importação dos dados de 2017
completos: de janeiro até dezembro. Replique os códigos abaixo em seu
RStudio:
# Importando o banco de dados {`sinan_hep_sp_dez_2017.dbf`} para o `R`
sinan_hep_sp_dez_2017<- read.dbf("Dados/sinan_hep_sp_dez_2017.dbf", as.is = TRUE)
sinan_hep_sp_cont_17 <- sinan_hep_sp_dez_2017 |>
mutate(
sem_epi = epiweek(DT_SIN_PRI),
ano = year(DT_SIN_PRI)
) |>
count(ano, sem_epi)
grafico_base_suavizado +
geom_col(data = sinan_hep_sp_cont_17,
aes(y = n, fill = 'cor_n_casos'), alpha = 0.4) +
scale_fill_manual(
name = "",
values = c('cor_n_casos' = 'deepskyblue'),
labels = "Número de casos até dez/2017"
) +
ggtitle("Diagrama de controle de Hepatite A em São Paulo/SP entre 2006-2017")#> `geom_smooth()` using method = 'loess' and formula 'y ~ x'
#> `geom_smooth()` using method = 'loess' and formula 'y ~ x'Figura 12: Gráfico do diagrama de controle de Hepatite A, em São Paulo/SP até dezembro de 2017.
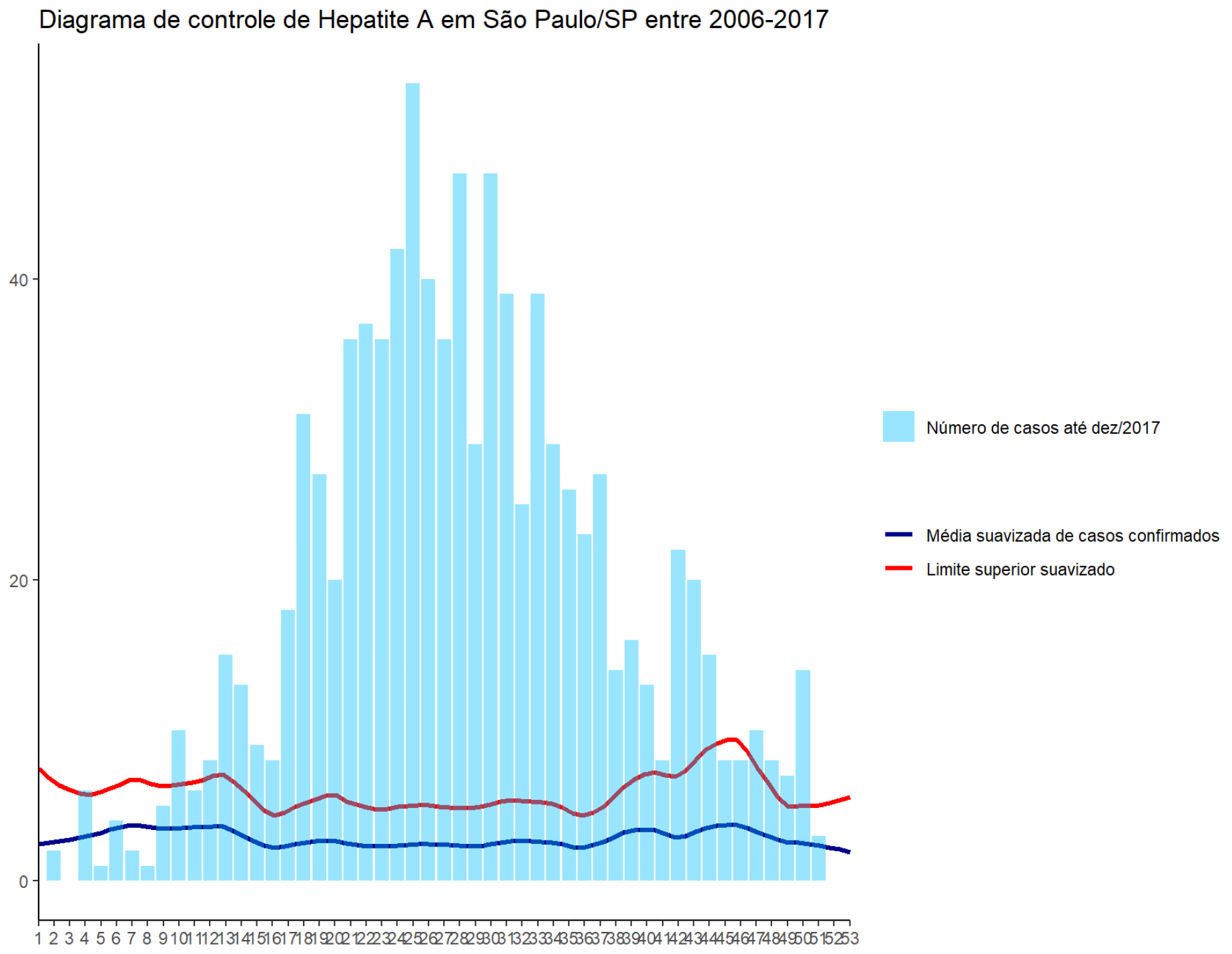
Observe na Figura 12 a existência de uma epidemia no Município de São Paulo, com número de casos maior que o limite superior.
Apesar da típica transmissão da infecção de Hepatite A por via fecal-oral, geralmente por meio de água e alimentos contaminados, nos últimos anos a disseminação tem sido associada às práticas sexuais, observada mais frequentemente entre homens que fazem sexo com homens (HSH).
Ocorreu que, em 2016, mais de 20 países da União Europeia foram acometidos com aumento na notificação de casos de Hepatites A e logo surgiram casos na Américas. Naquele momento a predominância era na população HSH. A cidade de São Paulo agiu à época com uma forte campanha para uso de preservativos e a prática do sexo seguro, além de investir na divulgação de informação para evitar a exposição fecal-oral durante a atividade sexual e cuidados de higiene. Outra medida importante foi reforçar principalmente a vacinação contra Hepatite A em pessoas com critérios já definidos para vacinação (pessoas vivendo com HIV/Aids, portadores crônicos de VHB e VHC e outras hepatopatias crônicas). Para saber mais sobre o surto em São Paulo clique aqui
Estas medidas foram determinantes para a redução da doença nos anos seguintes. A vacina contra a Hepatite A é fornecida pelo SUS de forma gratuita e é obrigatória no calendário vacinal do Programa Nacional de Imunização (PNI) para crianças de 15 meses a 4 anos, 11 meses e 29 dias.
Agora utilize seus novos conhecimentos na sua prática na vigilância. Será excelente, temos certeza!
Existem outros algoritmos para o cálculo do diagrama de controle. Veja um exemplo de uso no boletim InfoDengue para a semana 29 de 2022 em Foz do Iguaçu/PR.
Além disso, o R possui alguns pacotes que podem ajudar
na aplicação de outras metodologias para o acompanhamento de doenças e a
detecção de surtos. Alguns deles são MEM, qcc
e surveillance.
Nossos cursos
Pronto, chegamos ao final deste curso! Agora você já conhece as
principais ações para automatizar o diagrama de controle de doenças com
o apoio da linguagem de programação R. Quer seguir a diante
no aprendizado? Você encontrará outras etapas para aprofundamento das
análises de dados em vigilância em saúde nos outros cursos. Aproveite e
já faça sua inscrição nos cursos abaixo clicando nos links:
- Análises de dados para Vigilância em Saúde - curso básico.
- Visualização de dados de interesse para a vigilância em saúde.
- Produção automatizada de relatórios na vigilância em saúde.
- Linkage de bases de dados de saúde.
- Análise espacial de dados para a vigilância em saúde.
- Construção de painéis (dashboards) para monitoramento de indicadores de saúde.
Aproveite!


