2. Construindo o diagrama de controle de dengue
Agora, vamos praticar?
Utilizaremos dados de dengue como exemplo para demonstrar o método mais aplicados para monitoramento: o diagrama de controle semanal. As arboviroses são doenças constantemente monitoradas e de relevância epidemiológica em vários municípios do país, principalmente dengue, zika e chikungunya. O Brasil é o país que mais contribui com suas magnitudes nas Américas. Assim acompanhar suas trajetórias é uma atividade realizada com frequência pelas vigilâncias e pode se tornar muito difícil em anos epidêmicos.
Para essa prática, vamos demonstrar a coleta de dados de dengue e a
elaboração do diagrama de controle com apoio da linguagem
R, de forma a automatizar este processo de avaliação que
nos é rotineiro. Vamos lá?
Atenção
Uma série temporal que possua, por exemplo, apenas três anos não é indicada para ser monitorada via diagrama de controle por possuir poucos dados. O mesmo se aplica a uma série temporal de quatro anos seguidos com uma epidemia, pois não é possível estabelecer uma avaliação segura com poucos anos não epidêmicos!
Lembre-se que o diagrama de controle pode se tornar muito sensível a pequenas variações. Assim, cálculos inadequados podem levar o profissional de vigilância a realizar conclusões inadequadas da realidade como, por exemplo, indicar endemia quando há epidemia. Isso pode ocorrer se o canal epidêmico ficar com limite superior elevado pela incorporação de série histórica com anos epidêmicos.
2.1 Obtendo os dados
O primeiro passo é obter os dados que serão analisados. Utilizaremos aqui como fonte de dados o sistema InfoDengue. O InfoDengue é um projeto criado pela Fiocruz e pela Fundação Getúlio Vargas que emite alertas e faz o monitoramento de arboviroses para todo o Brasil. No seu site é possível acessar boletins epidemiológicos de alerta e obter séries temporais de casos de dengue, chikungunya ou zika notificados por grande parte dos municípios pelo Brasil.
O InfoDengue é uma fonte de dados bastante interessante pois além da incidência de casos oferece dados de temperatura e umidade dos municípios.
O InfoDengue é um sistema de alerta para arboviroses baseado em dados híbridos gerados por meio da análise integrada de dados minerados a partir da web social e de dados climáticos e epidemiológicos. Implementado em 2015, o sistema foi desenvolvido por pesquisadores do Programa de Computação Científica (Fundação Oswaldo Cruz, RJ) e da Escola de Matemática Aplicada (Fundação Getúlio Vargas) com a forte colaboração da Secretaria Municipal de Saúde do Rio de Janeiro e o Observatório da Dengue/UFMG.
Em 2021 o sistema ganhou amplitude nacional com o apoio do Ministério da Saúde, realizando análises complexas em nível estadual.
O InfoDengue disponibiliza relatórios semanais, sendo também possível baixar dados via API com séries históricas das arboviroses, variáveis climatológicas e correções de atraso de notificação (os chamados nowcast).
Clique AQUI para obter os boletins e diversos dados diretamente do site.
Para a nossa análise vamos utilizar a série histórica de dengue da cidade de Foz do Iguaçu, no Paraná, com dados entre o período de 2012 até 2022, organizados por semana epidemiológica. Para acessar esses dados do InfoDengue utilizaremos a sua API e, assim, além de automatizar nossa análise automatizaremos também a coleta de dados.
API se refere ao termo em inglês Application
Programming Interface, que pode ser traduzido como “Interface de
Programação de Aplicativos”. Ela funciona como uma espécie de porta que
garante em tempo real que haja conexão com diversas aplicações e
softwares, como o R. API’s são muito importantes na área da
saúde, e são amplamente utilizadas na atualidade por permitir a
integração de diferentes funcionalidades entre sistemas em um único
local.
A API torna o acesso às informações que queremos muito mais práticas, rápidas e é claro automatizadas. OS sistemas de informação do Ministério da Saúde estão em constantes transformações de digitais e alguns já é possível o acesso através de uma API como o e-SUS AB PEC e o e-SUS Notifica.
Por meio da API vamos conectar o seu R à base de dados
do InfoDengue e, assim, efetuar o download dos dados que
utilizaremos. Siga os passos abaixo para aprender a consultar a API e
obter os dados necessários em seu computador:
- Primeiro vamos instalar e carregar os pacotes necessários para
importar a API e criar o diagrama de controle de dengue. Serão o
tidyverse, olubridatee oxts. Replique o código abaixo em seuRStudio:
# Instalando os pacotes necessários
if(!require(tidyverse)) install.packages("tidyverse"); library(tidyverse)
if(!require(lubridate)) install.packages("lubridate"); library(lubridate)
if(!require(foreign)) install.packages("foreign"); library(foreign)
if(!require(xts)) install.packages("xts"); library(xts)- Em seguida, vamos lá no
RStudiodefinir os objetos necessários para gerar uma consulta à API do InfoDengue:
url: o endereço eletrônico (URL) da API,geocode: o código do município segundo IBGE de Foz do Iguaçu, que é 4108304,format: o formato dos dados; utilizaremos o arquivo do tipo.csv,disease: a arbovirose de interesse, que será a dengue,ew_starteew_end: o período que será estudado, a semana epidemiológica de início (1) e fim (53); aqui, em geral, pode-se selecionar parte de um ano, mas quando lidamos com séries anuais basta deixar 52 ou 53, eey_starteey_end: os anos de início e fim de nossa série histórica: 2012 e 2022 no nosso exemplo.
Veja abaixo como construímos o script no R com
essas informações necessárias. Replique essas linhas de código em seu
RStudio.
# Atribuição de dados para consultar a API do InfoDengue
url <- "https://info.dengue.mat.br/api/alertcity?"
# inserimos o endereço do InfoDengue
geocode <- 4108304 # indicamos código do IBGE de Foz do Iguaçu
disease <- "dengue" # selecionamos a doença
format <- "csv" # indicamos o formato de arquivo que será baixado
ew_start <- 1 # indicamos o início da semana epidemiológica
ew_end <- 53 # indicamos o final da semana epidemiológica
ey_start <- 2012 # indicamos o início do ano a ser exportado
ey_end <- 2022 # indicamos o final do ano a ser exportado- O terceiro passo é concatenar, ou seja, unir em uma única
string os objetos escritos no passo 2. Para
concatenar todos os objetos, vamos utilizar a função
paste0(). Dessa forma, criamos uma URL (um endereço como um link para acessar sites) de consulta à API que acessará o repositório e retornará os dados solicitados no formato.csv. O nosso objetivo aqui será programar o próprioRpara que ele se encarregue de trazer os dados usando a internet, de forma automática para o objeto que chamaremos de {consulta}.
Replique o código abaixo no seu RStudio e visualize o
objeto consulta que armazenará o link como de um
site qualquer, mas, no nosso caso, é também um caminho para fazermos o
download dos dados de dengue.
# programando o R para ele trazer os dados diretamente da internet
consulta <- paste0(url,
"geocode=", geocode,
"&disease=", disease,
"&format=", format,
"&ew_start=", ew_start,
"&ew_end=", ew_end,
"&ey_start=", ey_start,
"&ey_end=", ey_end)
# visualizando o link armazenado no objeto {`consulta`}
consulta#> [1] "https://info.dengue.mat.br/api/alertcity?geocode=4108304&disease=dengue&
#format=csv&ew_start=1&ew_end=53&ey_start=2012&ey_end=2022"Ao executar o código acima, é possível visualizar um link,
ou seja, a URL que criamos para consultar a API do InfoDengue.
Você poderá inspecionar esse caminho de acesso web clicando AQUI.
OU, se preferir, copie e cole o link (exibido em seu
output do RStudio) em seu navegador de internet e
visualize dos dados que foram solicitados. Aproveite e perceba como os
dados estão armazenados no InfoDengue.
Você também poderá obter esses dados manualmente. Veja na Figura 2 como é a tela para a seleção e download dos dados de forma manual. Clique AQUI para acessá-los do seu navegador de internet.
Figura 2: Tela da API do InfoDengue para download manual dos dados.
- Agora que armazenamos os dados no objeto {
consulta}, precisaremos executar a funçãoread_csv()para que oRcarregue o arquivo que é do tipo.csvcom os dados da dengue. Nesta etapa iremos armazenar estes dados em um novo objeto que nomearemos de {dengue_foz} para que possamos iniciar nossa análise sem alterar o banco de dados original. Lembre-se que esta é uma boa prática para quem utiliza linguagem de programação!
Vamos lá. Observe o script abaixo e replique-o em seu
RStudio:
# Armazenando o banco de dados do InfoDengue {consulta} no objeto {dengue_foz} para
# analisá-lo
dengue_foz <- read_csv(consulta)#> Rows: 562 Columns: 25
#> ── Column specification ────────────────────────────────────────────────────────
#> Delimiter: ","
#> dbl (23): SE, casos_est, casos_est_min, casos_est_max, casos, p_rt1, p_inc1...
#> date (2): data_iniSE, versao_modelo
#>
#> ℹ Use `spec()` to retrieve the full column specification for this data.
#> ℹ Specify the column types or set `show_col_types = FALSE` to quiet this message.Atenção
Pode ser que demore alguns segundos para que a conexão seja estabelecida e o download seja realizado. Verifique o sinal de internet de sua rede local e, caso encontre problemas, procure o serviço de apoio à informática da sua localidade.
Caso não consiga acessar a API do Infodengue você poderá utilizar o
arquivo localmente em seu computador. Para isso, acesse o menu lateral
“Arquivos” do curso e faça download do banco de dados de nome
consulta.csv.
Agora, replique o script abaixo em seu RStudio
e importe o arquivo para o R:
# Esta primeira linha verifica se o pacote está
# instalado.
# Caso não esteja, o código abaixo irá prosseguir com
# a instalação do pacote readr
if(!require(readr)) install.packages("readr")
#Carregando o pacote readr
library(readr)
# Utilize a função read_csv para importar os dados
# do arquivo consulta.csv para um objeto chamado "
# dengue_foz"
dengue_foz <- read_csv(file = "Dados/consulta.csv")
# visualizando o arquivo `consulta.csv` no objeto
# {`dengue_foz`}
dengue_foz#> # A tibble: 562 × 25
#> data_iniSE SE casos…¹ casos…² casos…³ casos p_rt1 p_inc…⁴ Local…⁵ nivel
#> <date> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
#> 1 2022-10-02 202240 163 115 633 111 6.74e-1 63.1 0 1
#> 2 2022-09-25 202239 161 148 236 146 6.30e-1 62.3 0 1
#> 3 2022-09-18 202238 156 150 185 149 3.47e-1 60.4 0 1
#> 4 2022-09-11 202237 145 141 157 141 4.49e-2 56.1 0 1
#> 5 2022-09-04 202236 169 166 179 166 1.41e-1 65.4 0 1
#> 6 2022-08-28 202235 167 165 175 166 5.38e-2 64.7 0 1
#> 7 2022-08-21 202234 199 197 206 197 4.08e-1 77.1 0 2
#> 8 2022-08-14 202233 202 201 209 201 1.65e-1 78.2 0 2
#> 9 2022-08-07 202232 196 195 201 196 1.05e-3 75.9 0 2
#> 10 2022-07-31 202231 210 210 215 210 1.96e-6 81.3 0 2
#> # … with 552 more rows, 15 more variables: id <dbl>, versao_modelo <date>,
#> # tweet <dbl>, Rt <dbl>, pop <dbl>, tempmin <dbl>, umidmax <dbl>,
#> # receptivo <dbl>, transmissao <dbl>, nivel_inc <dbl>, umidmed <dbl>,
#> # umidmin <dbl>, tempmed <dbl>, tempmax <dbl>, notif_accum_year <dbl>, and
#> # abbreviated variable names ¹casos_est, ²casos_est_min, ³casos_est_max,
#> # ⁴p_inc100k, ⁵Localidade_id
#> # ℹ Use `print(n = ...)` to see more rows, and `colnames()` to see all variable
# namesSiga em frente no curso.
Pronto, agora já importamos para o R um data.frame com dados de dengue da cidade de Foz do Iguaçu/PR no objeto {dengue_foz}. Vamos agora desenhar nosso diagrama de controle..
Como o número de notificações podem sofrer inclusões e alterações constantemente, é possível que os valores podem divergir de quando você importar. Isso é normal.
2.2 Montando o diagrama de controle
Até aqui já possuímos os dados que precisamos para a construção do nosso diagrama de controle do agravo dengue para estudar se há uma epidemia ou endemia município de Foz do Iguaçu/PR em 2022. Nesse momento agora precisamos escolher as variáveis necessárias para nossa análise. Esta etapa é fundamental!
Para esta escolha você necessitará estudar o dicionário de dados do InfoDengue, que se encontra disponível no site clicando AQUI. Observe a Tabela 1 abaixo e escolha as variáveis necessárias para a construção do diagrama de controle:
Tabela 1: Dicionário de dados da API do InfoDengue.
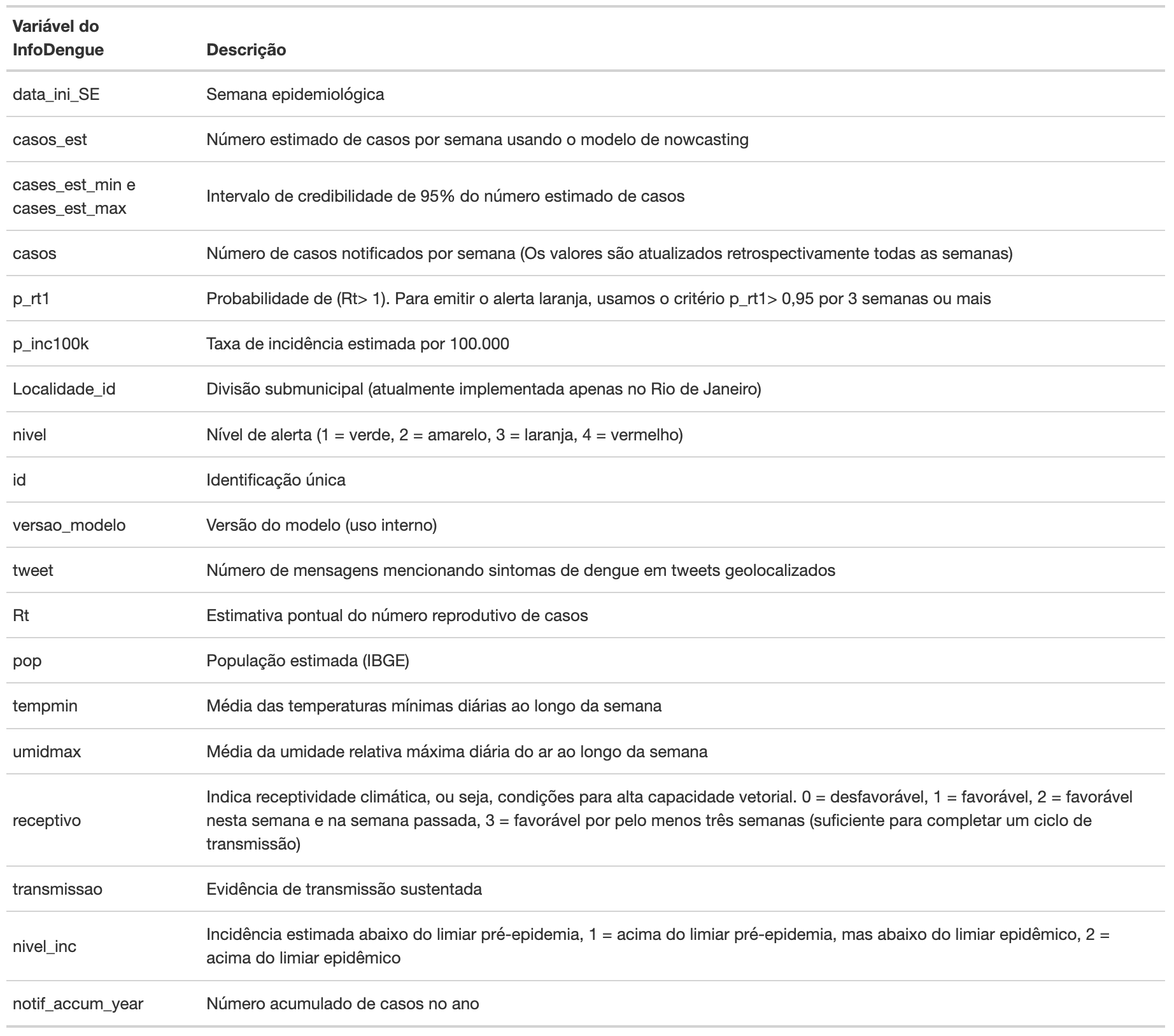
Agora que já conhecemos todas as variáveis disponíveis é possível verificar que utilizaremos nesse exemplo apenas três variáveis da tabela que fizemos download. Abaixo listamos as colunas necessárias:
data_iniSE: data de início da semana epidemiológica (SE),casos: número de casos de dengue na semana epidemiológica, ep_inc100k: incidência de dengue por 100 mil habitantes na semana epidemiológica.
Existem ainda várias outras variáveis que podem ser bastante
interessantes para avaliação da situação de dengue fornecidas pelo
InfoDengue tais como: temperatura mínima, umidade máxima e número de
twittes realizados sobre o tema no município (monitoramento
de rumores). Você pode visualizá-las e incluí-las para ampliar sua
análise.
Para o próximo passo criaremos um novo objeto, o
{foz_ts}. Ele será do tipo série temporal. Para
isso, utilizaremos a função xts() do pacote
xts. Para utilização da função será necessário definir dois
argumentos:
x: argumento que deverá indicar os dados a serem plotados. Ou seja, colocaremos noxa coluna da base de dados {dengue_foz} com os valores da incidência de casos (p_inc100k).order.by: argumento que deverá indicar a data correspondente aos dados indicados no argumentox. Aqui, utilizaremos a coluna com os valores da data de início das semanas epidemiológicas (data_iniSE).
Perceba que utilizaremos o operador $ (cifrão)
para indicar a seleção das variáveis dentro da base de dados. Observe o
script abaixo e replique-o em seu RStudio:
# criando o objeto {`foz_ts`}
foz_ts <- xts(
# selecionando a variável com os dados da incidência de dengue
x = dengue_foz$p_inc100k,
# selecionando a variável que contém as datas correspondentes
order.by = dengue_foz$data_iniSE)Agora que criamos uma tabela apenas com as variáveis
p_inc100k e data_iniSE, vamos transformá-las
em uma visualização gráfica utilizando a função plot().
Assim, criaremos a estrutura gráfica para a distribuição da incidência
do agravo dengue por 100 mil habitantes no período entre 2012 e 2022.
Para isso precisaremos utilizar os seguintes argumentos da função
plot():
x: indicar qual o objeto que está armazenando a série temporal. No nosso exemplo será a tabela {foz_ts}.main: para incluir o título do gráfico. Vamos utilizar “Distribuição da incidência de dengue em Foz do Iguaçu/PR”.ylab: para incluir título do eixo y do gráfico. Vamos colocar “Incidência p/ 100.000 hab.”.
Observe o script abaixo a estrutura do código com a
parametrização acima e replique-o em seu RStudio:
# Plotando o diagrama de controle
plot(
# indicando a série temporal
x = foz_ts,
# colocando o título do gráfico
main = 'Distribuição da incidência de dengue em Foz do Iguaçu/PR',
# escrevendo o título do eixo y
ylab = 'Incidência p/ 100.000 hab.'
)Figura 3: Gráfico de linha da distribuição da incidência de dengue por 100 mil habitantes, no período entre 2012 e 2022, em Foz do Iguaçu/PR.
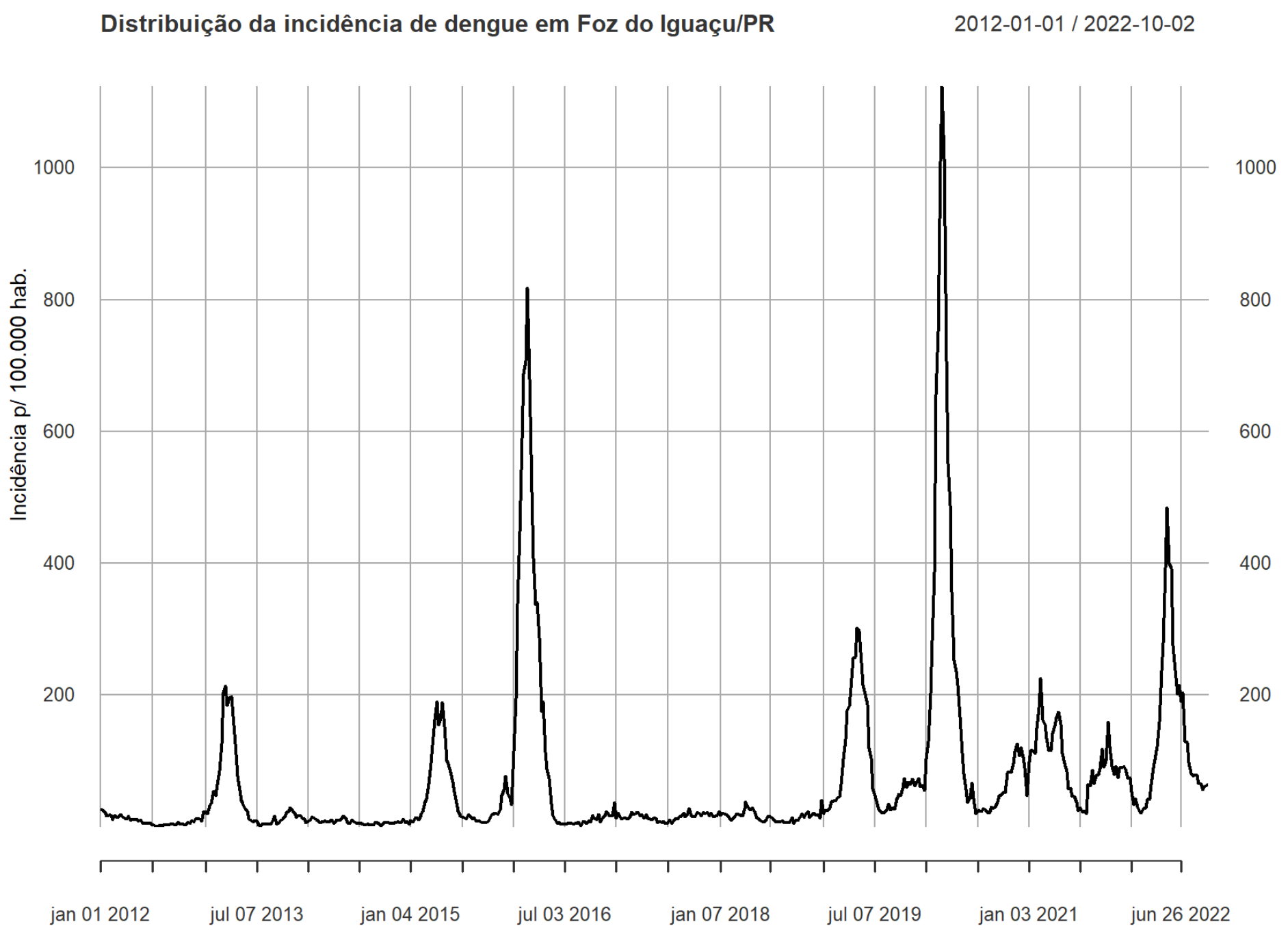
Você deverá obter um gráfico como o indicado na Figura 3 acima.
Precisamos analisar a distribuição dos valores de incidência do
agravo dengue em toda a série história escolhida (anos entre 2012 e
2022), observando-se especialmente se há anos epidêmicos desta série
temporal. Utilizaremos para isso a função summary().
Observe o script abaixo e replique-o em seu
RStudio:
# Analisando as estatísticas básicas do banco de dengue importado
summary(dengue_foz$p_inc100k)#> Min. 1st Qu. Median Mean 3rd Qu. Max.
#> 0.000 9.777 21.104 71.871 76.864 1124.500Observe o output! A média de incidência (mean)
é um pouco maior que 70 casos por 100 mil habitantes e a mediana é um
pouco maior que 20 casos por 100 mil habitantes. Essa diferença entre
média e mediana é um bom indicador de que há valores muito altos e
extremos puxando a média para cima, ou seja, outliers que estão
demonstrando uma distribuição assimétrica dos dados. Na verdade, podemos
observar isso também na Figura 3.
Os outliers são valores que estão fugindo do que se espera da normalidade dos dados e que podem, e provavelmente irão, causar distorções às suas análises.
Outliers são também comumente chamados em análise de dados de:
- valores que aparecem fora da curva,
- valores anômalos,
- valores discrepantes ou
- valores atípicos.
No nosso exemplo eles podem indicar surtos ou epidemias.
Para continuar a avaliar a distribuição dos dados que estamos analisando observaremos o comportamento da incidência de dengue ao longo dos dez anos (2012-2022). Para conseguir esta avaliação faremos (plotaremos) um gráfico do tipo boxplot.
Com o boxplot será possível visualizar a taxa de incidência em cada um dos anos exportados observando quais os anos se comportaram como outliers nesta série. Para isso, definiremos como parâmetro a classificação utilizada como critério pelo Programa Nacional de Controle de Dengue para avaliação da incidência de casos:
- Baixa incidência: até 100 casos por 100 mil habitantes;
- Média incidência: entre 101 e 299 casos por 100 mil habitantes, aqui utilizaremos como referência 200 casos;
- Alta incidência: 300 casos ou mais por 100 mil habitantes ou mais.
Representaremos estes parâmetros sobrepondo algumas linhas no boxplot:
- uma linha tracejada azul (no gráfico chamaremos de blue) para representar a baixa incidência,
- uma linha tracejada laranja (orange) para representar a média incidência, e
- uma linha tracejada vermelha (red) para representar a alta incidência.
Atenção
Apesar de serem parâmetros muito utilizados no âmbito da vigilância e monitoramento de dengue, a definição de outros valores é válida a partir da realidade do seu município ou estado.
Agora executaremos a função boxplot() para plotar o
gráfico. Vamos utilizar a variável p_inc100k da base de
dados {dengue_foz} estratificada pelo ano da data de início
da semana epidemiológica (data_iniSE). Fique atento ao
observar o script, pois esta união é indicada na linguagem
R pelo uso do símbolo ~(til). Aqui também
definiremos os títulos dos eixos x e y utilizando os argumentos
xlab e ylab, respectivamente.
Para plotar as linhas vamos utilizar também a função
abline() definindo os argumentos:
h: os valores que as linhas devem cruzar no eixo y . No nosso exemplo vamos colocar linhas naqueles valores que definem, baixa, média e alta incidência de dengue,lty: tipo da linha. Aqui o número dois (2) representa linhas tracejadas,lwd: largura da linha, ecol: cores das linhas. Como serão três linhas, utiliza-se a funçãoc()para definir as três cores.
Acompanhe o script abaixo e reproduza-o em seu
RStudio:
# Plotando gráfico boxplot com incidência por (`~`) ano
boxplot(dengue_foz$p_inc100k ~ year(dengue_foz$data_iniSE),
ylab = 'Incidência por 100.000 hab.',
xlab = "Ano de início da semana epidemiológica",
# Inserindo o título do boxplot
main = "Distribuição da incidência anual de dengue em Foz do Iguaçu-PR
entre 2012-2022.")
# Criando linhas de análise a partir dos parâmetros que definimos
abline(
h = c(300, 200, 70),
lty = 2,
lwd = 2,
col = c('red', 'orange', 'blue')
)Figura 4: Gráfico boxplot da distribuição da incidência de dengue por 100 mil habitantes, por ano, em Foz do Iguaçu/PR.

Visualize o gráfico que retornou em seu RStudio. Ele
deve ser semelhante ao observado aqui acima na Figura 4.
Precisaremos analisá-lo com atenção para indicar quais dos anos plotados são anos epidêmicos. Observe que, embora tenhamos em 2013 taxas mais altas que os demais anos, apenas seus outliers tocam a linha de cor laranja (média incidência). Ou seja, 2013 possui magnitude menor que os demais anos e o seu valor do terceiro quartil é inferior a 50%, por isso o incluiremos como ano não epidêmico. Assim, selecionaremos para a construção do diagrama de controle os anos de 2012, 2013, 2014, 2017 e 2018, pois são anos não epidêmicos - estão abaixo da baixa incidência de casos.
Agora que já escolhemos os anos não epidêmicos que
levaremos em conta na criação do diagrama de controle, precisamos criar
o objeto {nao_epidemic} que armazenará (receberá) esses
anos.
Muitas séries históricas não possuem anos epidêmicos e você não precisará excluir qualquer valor. Essa decisão irá variar de acordo com a doença/agravo, o local e o período considerados na análise.
Vamos lá! Acompanhe o script abaixo e replique-o em seu
RStudio:
# Criando o objeto {`nao_epidemic`} com anos não epidêmicos
nao_epidemic <- c(2012, 2013, 2014, 2017, 2018)Pronto! Dados armazenados no objeto {nao_epidemic}.
Agora podemos utilizá-los para criar mais um passo da nossa análise.
Lembre-se que estamos analisando o comportamento de dengue em Foz do
Iguaçu/PR em 2022 e, para isso, estamos comparando os dados deste ano
com os demais anos não epidêmicos escolhidos (2012, 2013, 2014, 2017 e
2018) em um diagrama de controle. Continue sua análise seguindo os
passos abaixo:
- criaremos o objeto {
dengue_2022} que armazenará os dados, - utilizaremos a função
filter()do pacotedplyre a funçãoyear()do pacotelubridatepara a seleção do ano de 2022, e - criaremos uma coluna contendo as semanas epidemiológicas com a
função
mutate().
É bem simples. Observe o script abaixo e replique-o em seu
RStudio:
# Criando o objeto {`dengue_2022`} com ano de 2022
dengue_2022 <- dengue_foz |>
# Filtrando o ano para 2022
filter(year(data_iniSE) == 2022) |>
# Criando uma nova coluna chamada 'sem_epi', referente à semana epidemiológica
mutate(sem_epi = epiweek(data_iniSE))Pronto! Agora utilizaremos o objeto {dengue_2022} para
outra etapa da criação do diagrama. Acompanhe o passo a passo abaixo
escrito no script. Com ele iremos construir a estrutura básica
do diagrama de controle.
Esta etapa é importante, pois criaremos a tabela
{dengue_stat} com cinco variáveis calculadas: a média,
desvio padrão e limites superior e inferior da taxa de incidência por
semana epidemiológica para plotar no gráfico. Para isso será
necessário:
- Filtrar utilizando as funções
filter()eyear(), os anos não epidêmicos utilizando o operador %in% que retorna TRUE quando o ano está contido no vetor nao_epidemic. - Utilizar a função
mutate()para extrair da data de início da semana epidemiológica o número da semana e atribuir seu valor a sem_epi. - Agrupar com a função
group_by()os dados por semana, ou seja, teremos os dados por semana para cada ano selecionado. - Utilizar a função
summarise()e criar a média, desvio padrão e os limites máximo e mínimo para cada uma das semanas epidemiológicas dos períodos não epidêmicos.
Vamos lá, observe o script abaixo e replique o código em seu
RStudio:
# Criando o gráfico com o diagrama de controle
dengue_stat <- dengue_foz |>
# Filtrando os dados da série em que o ano não é epidêmico
filter(year(data_iniSE) %in% nao_epidemic) |>
# Criando uma nova coluna chamada 'sem_epi', referente à semana epidemiológica
mutate(sem_epi = epiweek(data_iniSE)) |>
# Agrupando os dados pela semana epidemiológica
group_by(sem_epi) |>
# Criando medidas-resumo e limites superior e inferior
summarise(
n = n(),
media = mean(p_inc100k, na.rm = TRUE),
desvio = sd(p_inc100k, na.rm = TRUE) ,
sup = media + 2 * desvio,
inf = media - 2 * desvio
)Visualize agora as primeira linhas da tabela
{dengue_stat} utilizando a função head().
Observe o script abaixo e replique-o em seu
RStudio:
# Visualizando a tabela {`dengue_stat`}
head(dengue_stat)#> # A tibble: 6 × 6
#> sem_epi n media desvio sup inf
#> <dbl> <int> <dbl> <dbl> <dbl> <dbl>
#> 1 1 6 21.4 11.8 45.0 -2.24
#> 2 2 5 18.5 6.51 31.5 5.49
#> 3 3 5 17.3 5.07 27.4 7.13
#> 4 4 5 18.7 7.90 34.5 2.86
#> 5 5 5 19.4 8.94 37.2 1.49
#> 6 6 5 20.9 18.1 57.2 -15.4Pronto, agora já temos as medidas necessárias para a construção gráfica do diagrama de controle de dengue. Assim, já poderemos fazer seu gráfico e incluir os valores de incidência da dengue às semanas epidemiológicas do ano de 2022.
Será necessário utilizar as funções do pacote ggplot2,
um pacote construído para visualização de dados. Com o
ggplot2 os gráficos podem ser construídos de camada em
camada, detalhando a visualização dos dados que queremos analisar.
Acompanhe o script abaixo com atenção e replique-o em seu
RStudio. Nesse curso não teremos a oportunidade de detalhar
os comandos para gerar os gráficos, assim, se tiver interesse, clique aqui para saber mais sobre o
ggplot2. Aliás, vale muito a pena conhecer melhor o
ggplot2 para fazer gráficos muito interessantes. Mas para
fazer os seus diagramas de controle basta seguir o script
abaixo e adaptar as informações às suas necessidades.
# Definindo a base a ser utilizada
ggplot(data = dengue_stat) +
# Definindo argumentos estéticos com as variáveis usadas em x e em y
aes(x = sem_epi, y = media) +
# Adicionando a linha referente à incidência média de dengue.
# Adicionando uma geometria de linha na cor azul e largura de 1.2 pixel
geom_line(aes(color = 'cor_media_casos'), size = 1.2) +
# Adicionando uma geometria de linha na cor laranja. Além disso, inserindo
# um argumento estético para o eixo y que, no caso, é a variável de limite
# superior
geom_line(aes(y = sup, color = 'cor_limite'), size = 1.2) +
# Adicionando uma geometria de colunas utilizando a base de dados
# {`dengue_2022`} e y como a incidência de dengue em 2022. O argumento
# `fill` refere-se à cor das barras e `alpha` à transparência.
geom_col(data = dengue_2022,
aes(y = p_inc100k, fill = 'cor_incidencia'), alpha = 0.4) +
# Arrumando o eixo x, definindo o intervalo de tempo que será utilizado (`breaks`)
# uma sequência de semanas epidemiológicas de 1 a 53
# o argumento `expand` ajuda nesse processo.
scale_x_continuous(breaks = 1:53, expand = c(0, 0)) +
# Definindo os títulos dos eixos x e y
labs(x = 'Semana Epidemiológica',
y = 'Incidência por 100 mil hab.',
title = 'Diagrama de controle de dengue em Foz do Iguaçu/PR no ano de
2022.') +
# Definindo o tema do gráfico
theme_classic() +
# Criando a legenda das linhas
scale_color_manual(
name = "",
values = c('cor_media_casos' = 'darkblue', 'cor_limite' = 'red'),
labels = c("Incidência média", "Limite superior")
) +
# Criando a legenda das barras
scale_fill_manual(
name = "",
values = c('cor_incidencia' = 'deepskyblue'),
labels = "Incidência de dengue em 2022"
)Figura 5 Gráfico do Diagrama de Controle de dengue em Foz do Iguaçu/PR.
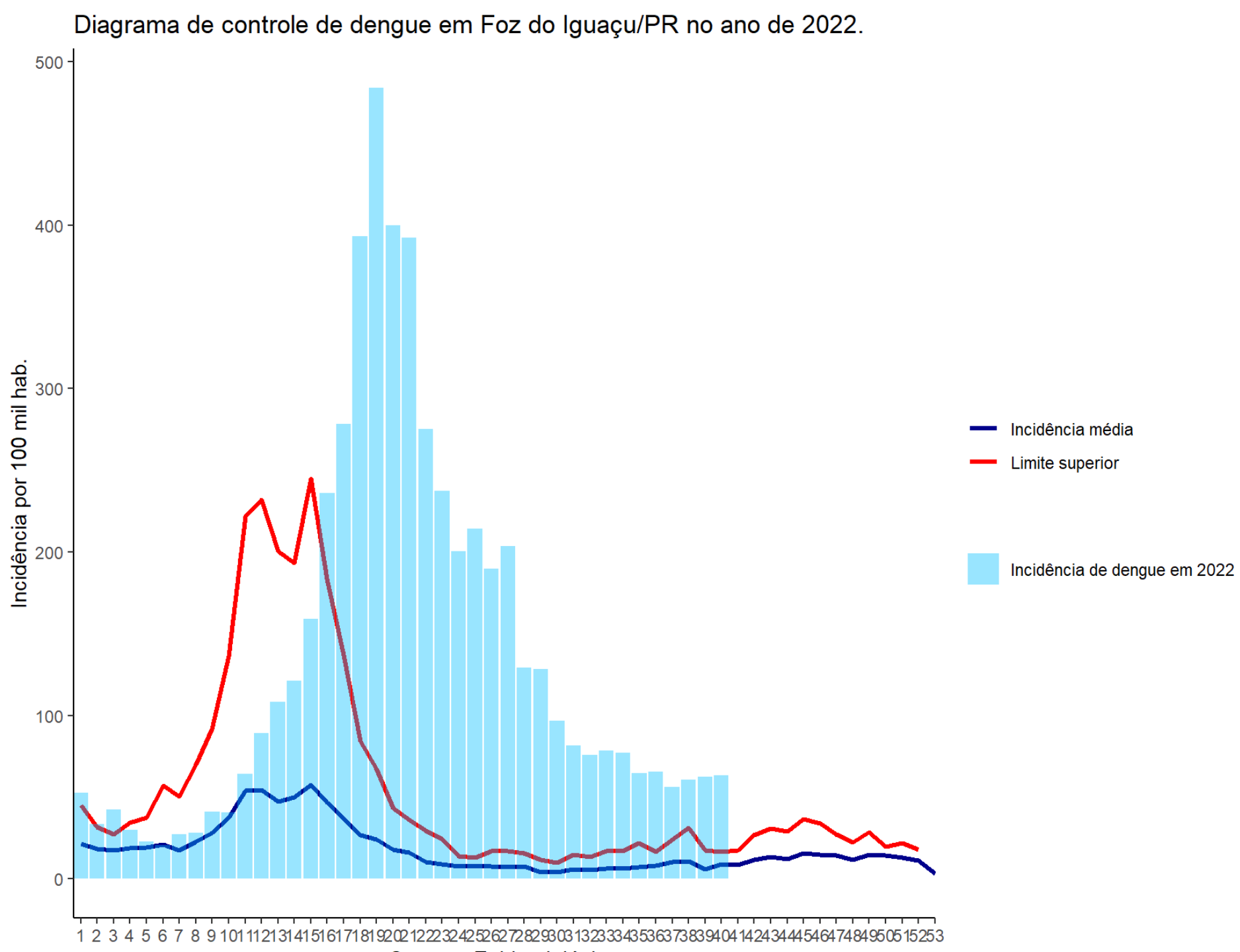
Ao observar a Figura 5 acima verificamos que 2022 foi um ano que ultrapassou o limite superior do diagrama de controle. Portanto, concluímos que Foz do Iguaçu pode estar vivendo um ano epidêmico. Ao observarmos a distribuição dos dados verificamos também que o padrão sazonal foi tardio em relação aos anos não epidêmicos. Isso significa que as semanas de maior incidência estavam entre as semanas epidemiológicas 10 e 16 (entre o mês de março e primeira quinzena de abril). Já em 2022 as semanas com maior incidência ficaram entre 17 e 23 (última semana de abril a primeira semana de junho), com o pico na semana 19 (entre 8 e 14 de maio).
Outro ponto a ser destacado na análise é que o limite superior (linha
de cor vermelha) possui um padrão que oscila um pouco quando temos taxas
de incidência maiores de 200 por 100 mil habitantes. Algumas vezes
precisamos alisar estas oscilações e minimizar esse efeito utilizando a
função stat_smooth() para suavizar as médias e o limite
superior. Para isto, basta substituir a função geom_line()
pela função stat_smooth() no script utilizado.
A função stat_smooth() suavizará a linha do gráfico,
sendo o argumento span responsável em diminuir ou aumentar
a suavização (de 0 a 1, sendo que quanto mais próximo a 1 mais suavidade
se obtém). O argumento se definido como FALSE
desabilita o intervalo de confiança ao redor das linhas. Os demais
argumentos possuem a mesma função.
Acompanhe o script a seguir com atenção e veja a diferença
do diagrama ao suavizarmos os ruídos. Replique os códigos em seu
RStudio:
# Definindo a base a ser utilizada
ggplot(data = dengue_stat) +
# Definindo argumentos para as variáveis do eixo x e y do gráfico
aes(x = sem_epi, y = media) +
# Suavizando a linha referente à incidência média de dengue
# o argumento `size` para definir largura da linha = 1.2 pixel
# o argumento `se` = FALSE desabilita o intervalo de confiança
# e o argumento `span` definindo o valor da suavização
stat_smooth(
aes(color = 'cor_incidencia_media'),
size = 1.2,
se = FALSE,
span = 0.2
) +
# Suavizando a linha referente ao limite superior
stat_smooth(
aes(y = sup, color = 'cor_limite'),
size = 1.2,
se = FALSE,
span = 0.2
) +
# Adicionando uma geometria de colunas utilizando a base de dados
# {`dengue_2022`} e y como a incidência de dengue em 2022.
geom_col(data = dengue_2022,
aes(y = p_inc100k, fill = 'cor_incidencia'),
alpha = 0.4) +
# Arrumando o eixo x, definindo o intervalo de tempo que será utilizado (`breaks`)
# uma sequência de semanas epidemiológicas de 1 a 53
# o argumento `expand` ajuda nesse processo.
scale_x_continuous(breaks = 1:53, expand = c(0, 0)) +
# Definindo os títulos dos eixos x, y e também do gráfico
labs(x = 'Semana Epidemiológica',
y = 'Incidência por 100 mil hab.',
title = 'Diagrama de controle de dengue em Foz do Iguaçu/PR no ano
de 2022.') +
# Definindo o tema do gráfico
theme_classic() +
# Criando a legenda das linhas
scale_color_manual(
name = "",
values = c('cor_incidencia_media' = 'darkblue', 'cor_limite' = 'red'),
labels = c("Incidência média de casos", "Limite superior")
) +
# Criando a legenda das barras
scale_fill_manual(
name = "",
values = c('cor_incidencia' = 'deepskyblue'),
labels = "Incidência de dengue em 2022"
)Figura 6 Gráfico alisado do diagrama de controle de dengue em Foz do Iguaçu/PR.
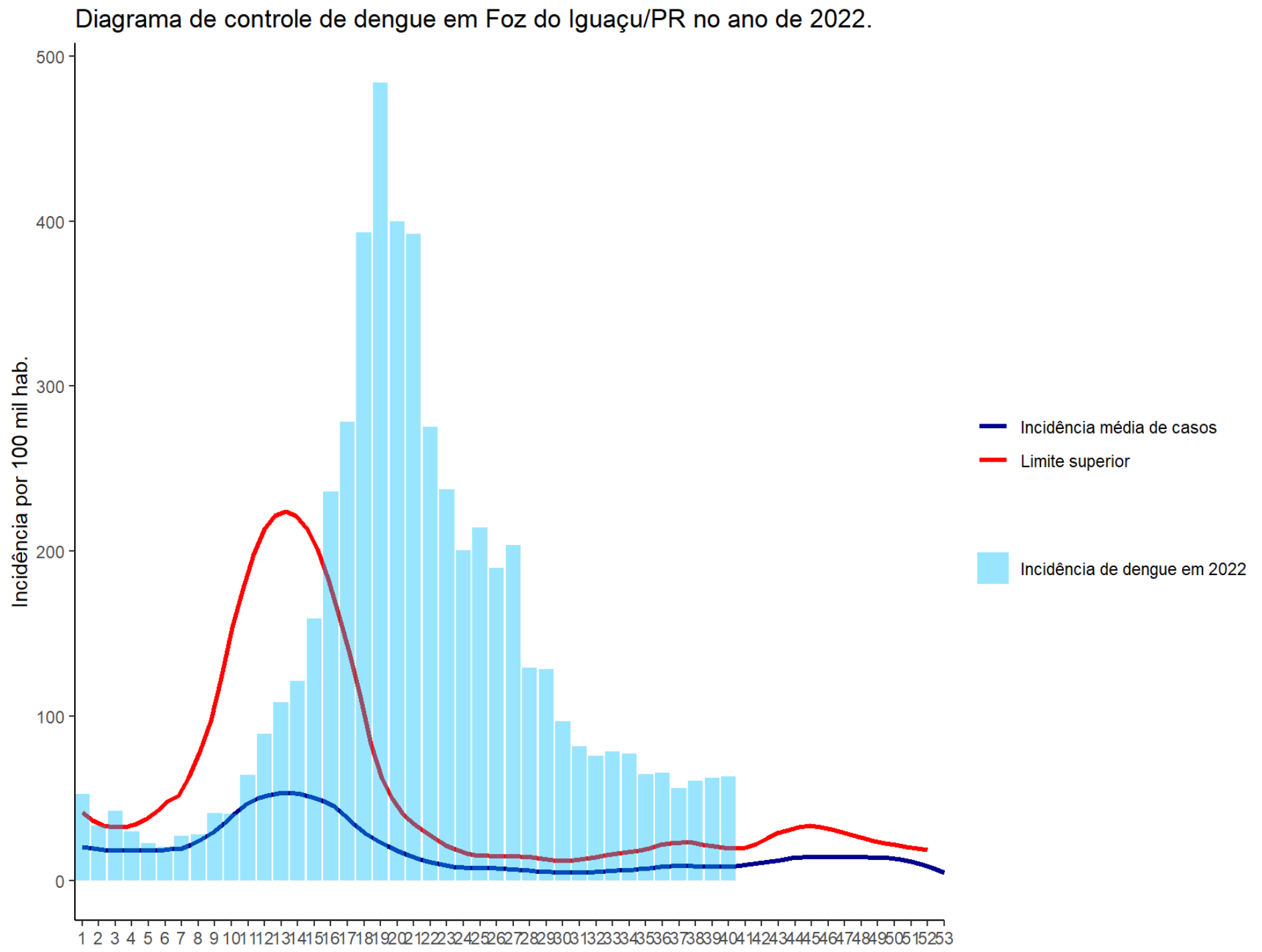
Veja a Figura 6. Ficou bacana, não é mesmo? Com o uso da função
stat_smooth() foi possível visualizar de uma maneira mais
clara que a partir da semana epidemiológica de número 16, em 2022, a
taxa de incidência de dengue ultrapassa o limite superior do diagrama de
controle, exibindo valores acima do esperado até depois da semana
28.
Vale também ressaltar o fato de que não foi usado o limite inferior do diagrama de controle, pois, neste caso, para os anos endêmicos o intervalo inferior seria menor que zero na maior parte das semanas. Isto pode variar conforme o agravo e podemos utilizar essa técnica para monitorar questões operacionais como, por exemplo, atraso de notificação.
Existem diversas tabelas e ferramentas para ajudar a selecionar as cores para a personalização de seu gráfico. Você pode consultar a página da w3 color picker e visualizar algumas cores.
Lembre-se que no R o símbolo (#) indica um
código hexadecimal. Assim, o código #000000 representa a
cor preta e #FFFFFF representa a cor branca. Combinando as
três cores básicas podemos especificar mais de 16 milhões de cores e
tons de cinza!
Para saber mais sobre a visualização gráfica de dados faça o curso “Visualização de dados de interesse para a vigilância em saúde” disponível na nossa coleção de cursos para a vigilância em saúde.
Pronto. Você já é capaz de avaliar a situação de dengue em qualquer município do Brasil. Mas como seria produzir um diagrama de controle para outras doenças? Acompanhe na seção a seguir o desenvolvimento de mais um diagrama de controle utilizando dados de Hepatite A.


