3. Tipo de variáveis
Quais os tipos de dados estão presentes em uma análise? Antes de mais nada, vamos falar sobre o conteúdo presente nas variáveis (colunas). Elas podem conter qualquer característica ou atributo coletado no dia a dia da vigilância, como valores referentes à identificação de uma pessoa como o bairro, sexo ou sua idade, tudo isso pode ser chamado de variável.
Observe na Figura 16 o banco de dados exportado do Sivep Gripe com suas variáveis destacadas.
Figura 16: Tabela do Sivep Gripe com suas variáveis (colunas) para análise.
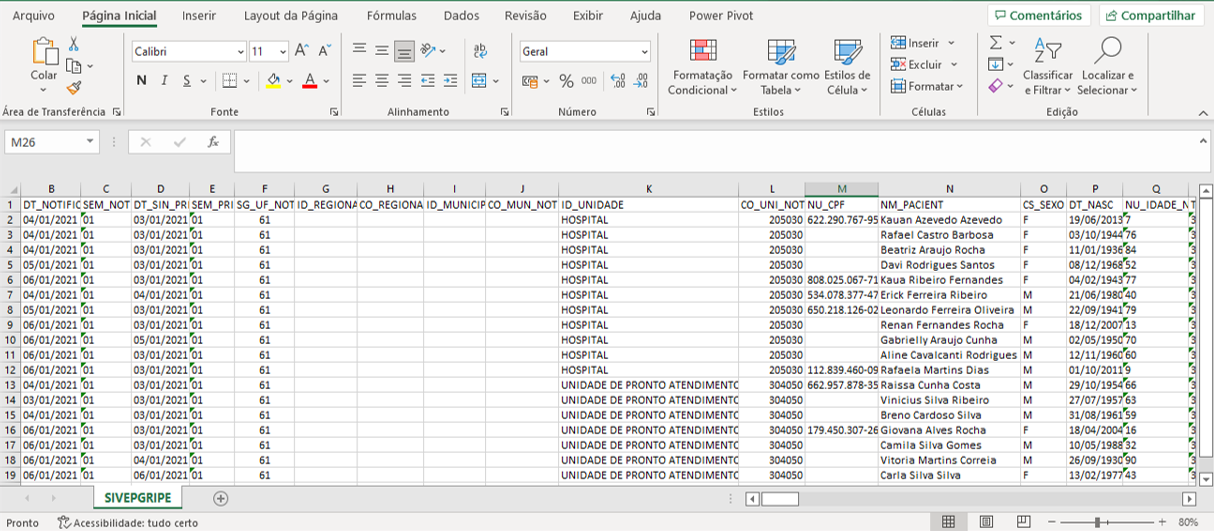
Essas variáveis podem ser classificadas a partir do tipo de dados que armazenam (Figura 17), podendo ser quantitativas quando armazenam dados de medidas como um número, ou qualitativas quando registram as características que não são mensuráveis, como textos. Não se preocupe, ao final deste módulo você estará pronto para analisar qualquer tipo de variável.
Figura 17: Tabela de classificação dos tipos de dados.
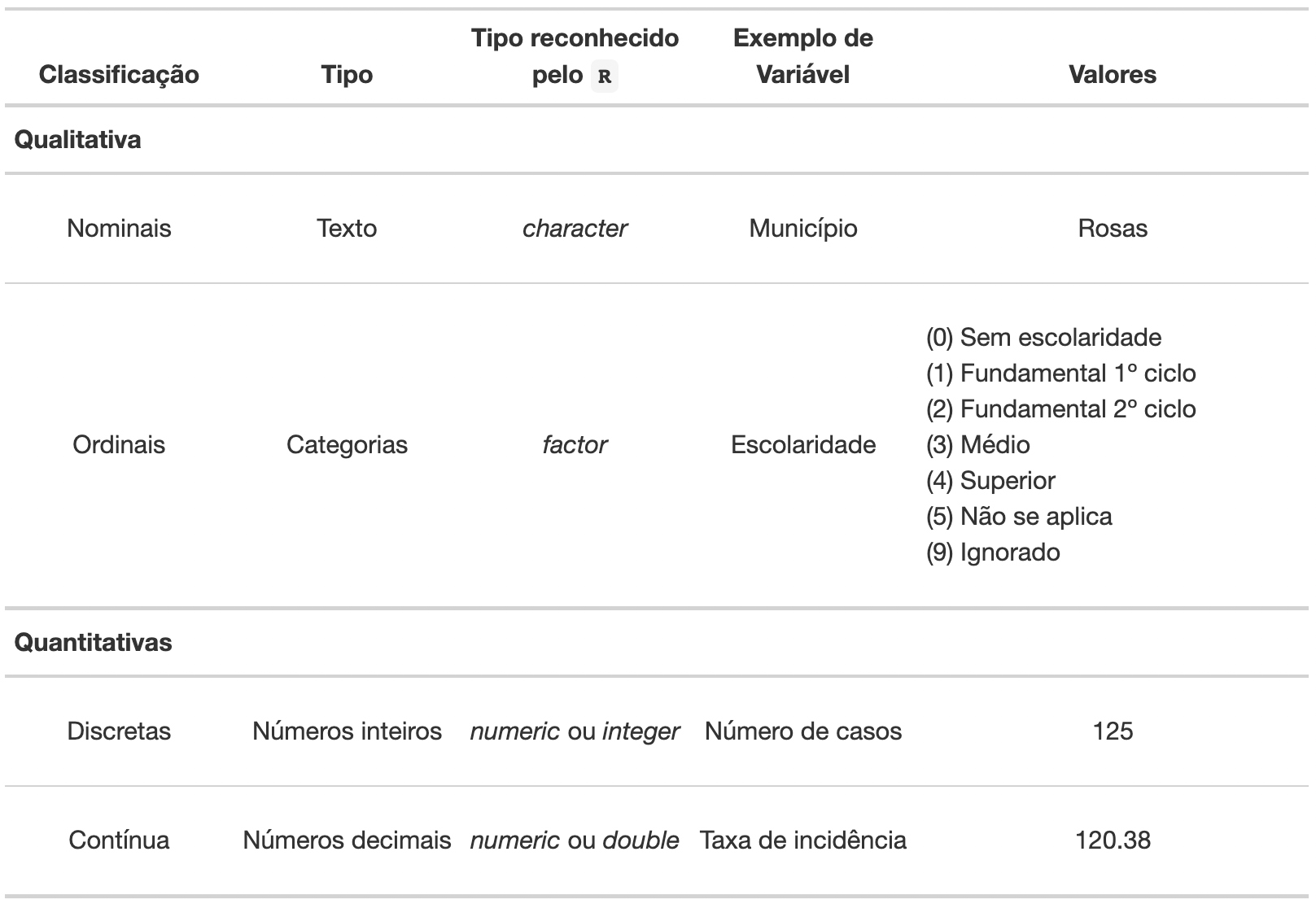
3.1 Variáveis quantitativas
Os dados contidos em variáveis quantitativas são aquelas medidas como um número, com caráter objetivo. Podem ser do tipo discreta ou do tipo contínua:
Discretas: podem assumir somente valores inteiros obtidos por contagem. São exemplos de variáveis numéricas discretas o número de gestações anteriores constante na Declaração de Nascido Vivo (SINASC), o número de contatos examinados coletado na ficha de Tuberculose e o número de lesões cutâneas dermatológicas apresentadas pelo paciente coletado na ficha de Hanseníase.
Contínuas: admitem qualquer valor numérico em determinado intervalo de variação. São exemplos de variáveis numéricas contínuas o peso ao nascer do recém-nascido, coletado na Declaração de Nascido Vivo (SINASC), resultados de exames de glicemia, aferição de pressão.
De modo geral, as variáveis quantitativas no R são
numéricas do tipo inteiro (integer) e do tipo decimal
(double).
Atenção
Ao realizar a etapa de importação de um banco de dados, não necessariamente o tipo da variável estará definido corretamente. O profissional de vigilância que analisa dados deve sempre avaliar, ou seja, inspecionar o banco de dados que está analisando.
Para inspecionar o tipo de variável do banco, vamos utilizar a função
sapply(). Mas para utilizá-la, necessitamos definir os seus
principais argumentos da seguinte maneira:
- argumento
X: incluir qual será o banco de dados, no caso {dados_sivep}; - argumento
FUN: permite que a gente configure o que queremos visualizar ou saber de cada variável ou coluna do banco de dados. Aqui, escolheremos argumentoFUN = typeof, isso porque nos permitirá visualizar o tipo (type, em inglês) de dado armazenado em cada variável.
Observe e reproduza o script abaixo no seu
RStudio e verifique se o output é similar ao que
apresentamos:
sapply(X = dados_sivep, FUN = "typeof")#> NU_NOTIFIC DT_NOTIFIC SEM_NOT DT_SIN_PRI SEM_PRI SG_UF_NOT
#> "double" "character" "character" "character" "character" "double"
#> ID_REGIONA CO_REGIONA ID_MUNICIP CO_MUN_NOT ID_UNIDADE CO_UNI_NOT
#> "logical" "logical" "logical" "logical" "character" "double"
#> NU_CPF NM_PACIENT CS_SEXO DT_NASC NU_IDADE_N TP_IDADE
#> "character" "character" "character" "character" "character" "character"
#> COD_IDADE CS_GESTANT CS_RACA CS_ETINIA CS_ESCOL_N NM_MAE_PAC
#> "character" "character" "character" "logical" "character" "character"
#> NU_CEP ID_PAIS CO_PAIS SG_UF ID_RG_RESI CO_RG_RESI
#> "character" "character" "character" "logical" "logical" "character"
#> ID_MN_RESI CO_MUN_RES NM_BAIRRO NM_LOGRADO NU_NUMERO NM_COMPLEM
#> "character" "logical" "character" "character" "character" "logical"
#> NU_DDD_TEL NU_TELEFON CS_ZONA SURTO_SG NOSOCOMIAL AVE_SUINO
#> "double" "character" "character" "character" "character" "character"
#> FEBRE TOSSE GARGANTA DISPNEIA DESC_RESP SATURACAO
#> "character" "character" "character" "character" "character" "character"
#> DIARREIA VOMITO OUTRO_SIN OUTRO_DES FATOR_RISC PUERPERA
#> "character" "character" "character" "character" "character" "character"
#> CARDIOPATI HEMATOLOGI SIND_DOWN HEPATICA ASMA DIABETES
#> "character" "character" "character" "character" "character" "character"
#> NEUROLOGIC PNEUMOPATI IMUNODEPRE RENAL OBESIDADE OBES_IMC
#> "character" "character" "character" "character" "character" "logical"
#> OUT_MORBI MORB_DESC VACINA DT_UT_DOSE MAE_VAC DT_VAC_MAE
#> "character" "character" "character" "character" "logical" "logical"
#> M_AMAMENTA DT_DOSEUNI DT_1_DOSE DT_2_DOSE ANTIVIRAL TP_ANTIVIR
#> "logical" "logical" "logical" "logical" "character" "character"
#> OUT_ANTIV DT_ANTIVIR HOSPITAL DT_INTERNA SG_UF_INTE ID_RG_INTE
#> "logical" "character" "character" "double" "logical" "logical"
#> CO_RG_INTE ID_MN_INTE CO_MU_INTE NM_UN_INTE CO_UN_INTE UTI
#> "logical" "logical" "logical" "logical" "logical" "character"
#> DT_ENTUTI DT_SAIDUTI SUPORT_VEN RAIOX_RES RAIOX_OUT DT_RAIOX
#> "character" "character" "character" "character" "character" "character"
#> AMOSTRA DT_COLETA TP_AMOSTRA OUT_AMOST REQUI_GAL PCR_RESUL
#> "character" "character" "character" "logical" "logical" "character"
#> DT_PCR POS_PCRFLU TP_FLU_PCR PCR_FLUASU FLUASU_OUT PCR_FLUBLI
#> "character" "character" "logical" "logical" "logical" "logical"
#> FLUBLI_OUT POS_PCROUT PCR_VSR PCR_PARA1 PCR_PARA2 PCR_PARA3
#> "logical" "character" "logical" "logical" "logical" "logical"
#> PCR_PARA4 PCR_ADENO PCR_METAP PCR_BOCA PCR_RINO PCR_OUTRO
#> "logical" "logical" "logical" "logical" "logical" "logical"
#> DS_PCR_OUT LAB_PCR CO_LAB_PCR CLASSI_FIN CLASSI_OUT CRITERIO
#> "logical" "character" "double" "character" "logical" "character"
#> EVOLUCAO DT_EVOLUCA DT_ENCERRA OBSERVA NOME_PROF REG_PROF
#> "character" "character" "character" "character" "logical" "character"
#> DT_DIGITA HISTO_VGM PAIS_VGM CO_PS_VGM LO_PS_VGM DT_VGM
#> "double" "character" "logical" "logical" "logical" "logical"
#> DT_RT_VGM PCR_SARS2 PAC_COCBO PAC_DSCBO OUT_ANIM DOR_ABD
#> "logical" "character" "logical" "logical" "logical" "character"
#> FADIGA PERD_OLFT PERD_PALA TOMO_RES TOMO_OUT DT_TOMO
#> "character" "character" "character" "double" "character" "character"
#> TP_TES_AN DT_RES_AN RES_AN LAB_AN CO_LAB_AN POS_AN_FLU
#> "double" "logical" "character" "logical" "logical" "logical"
#> TP_FLU_AN POS_AN_OUT AN_SARS2 AN_VSR AN_PARA1 AN_PARA2
#> "logical" "logical" "logical" "logical" "logical" "logical"
#> AN_PARA3 AN_ADENO AN_OUTRO DS_AN_OUT TP_AM_SOR SOR_OUT
#> "logical" "logical" "logical" "logical" "double" "logical"
#> DT_CO_SOR TP_SOR OUT_SOR DT_RES RES_IGG RES_IGM
#> "character" "double" "logical" "character" "character" "character"
#> RES_IGA NU_DO POV_CT TP_POV_CT TEM_CPF ESTRANG
#> "logical" "double" "character" "logical" "logical" "logical"
#> NU_CNS VACINA_COV DOSE_1_COV DOSE_2_COV DOSE_REF FAB_COV_1
#> "logical" "logical" "logical" "logical" "logical" "logical"
#> FAB_COV_2 FAB_COVREF LOTE_1_COV LOTE_2_COV LOTE_REF FNT_IN_COV
#> "logical" "logical" "logical" "logical" "logical" "logical"Perceba que ao executar a função sapply() no
output acima é possível visualizar o nome das variáveis e, logo
abaixo de cada uma delas, seu tipo (entre aspas). Observe a variável
NU_IDADE_N, que representa a idade do paciente internado.
Ela foi classificada pelo R como character
(texto), porém sabemos que idade não é um texto, ela apenas está sendo
classificada como. As variáveis devem sempre ser classificadas
corretamente, pois para rodar as suas análises calculando medidas como
frequência, média ou qualquer outro cálculo, será necessário indicar a
função adequada vinculando-a ao tipo de variável para que o
R execute o código sem erros.
3.2 Variáveis qualitativas
Quanto a variáveis qualitativas, são aquelas que registram as características particulares e que não são mensuráveis como um número e, geralmente, são definidas como categorias. Podem ser classificadas em ordinais e nominais:
Ordinais: são aquelas que suas categorias expressam uma ordem ou hierarquia. Exemplos comuns são aquelas variáveis que representam uma resposta de uma avaliação: muito ruim, ruim, regular, bom e excelente. Obviamente a classificação excelente é melhor do que bom e assim sucessivamente. Na Vigilância, temos o exemplo da variável grau de incapacidade física no diagnóstico (notificação de Hanseníase do SINAN), a variável escolaridade categorizada, e outras.
Nominais: são as que possuem categorias que não têm nenhuma ordem entre elas. Podem ser dicotômicas (duas categorias) ou ter várias categorias (politômicas). Como exemplos há coluna com dados de sexo (Masculino, Feminino e Ignorado), o resultado de um exame sorológico (Positivo, Negativo, Inconclusivo, Não realizado) ou até mesmo a ocorrência de uma hospitalização (Sim, Não, Ignorado).
É importante que você saiba que as variáveis qualitativas no
R podem ser do tipo fator (factor) ou somente
do tipo texto (character). Os fatores são um tipo de objeto
no qual as categorias das variáveis são chamadas de levels
(níveis ou hierarquias) e podem possuir um rótulo para cada categoria,
chamado de label (nome ou rótulos). Estas categorias podem
ser estruturadas no formato de:
- números inteiros (
integer), ou - textos (
character).
Atenção
É importante destacar que para a manipulação de uma variável no
R é preciso que ela seja transformada em fator. Para isso
utiliza-se a função factor().
Pronto, vamos praticar! Imagine que você necessita analisar a
classificação final dos casos de SRAG do Estado de Rosas. Para isto
necessitaremos transformar todos os dados contidos na variável
CLASSI_FIN, do banco de dados {dados_sivep}, e
para isso utilizaremos o dicionário do banco de dados disponível no menu
lateral “Arquivos”, do módulo.
Observe na Figura 18 abaixo como está estruturada a variável
CLASSI_FIN do banco de dados {dados_sivep}
:
Figura 18: Variável CLASSI_FIN do Sivep Gripe visualizada pelo Microsoft Excel.
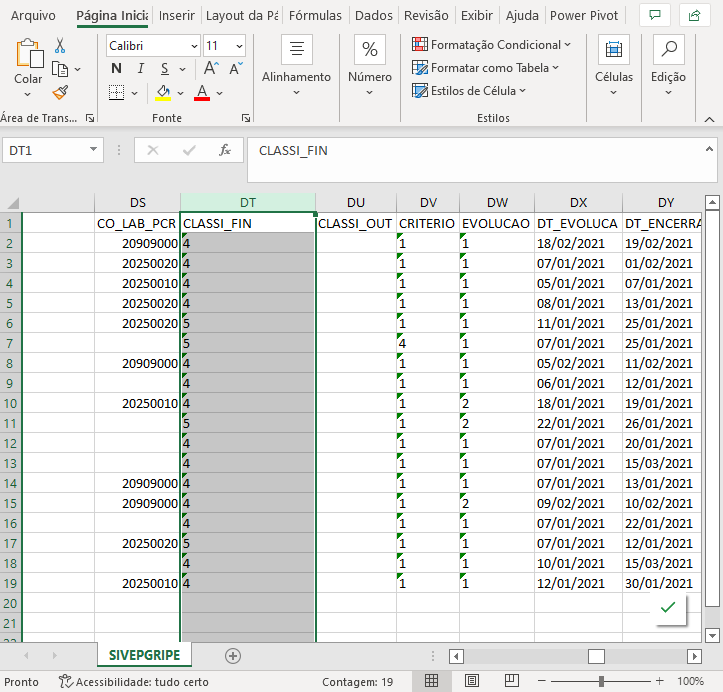
Conforme dicionário de dados, a classificação final do caso (Figura 18) pode ser referida da seguinte forma:
- 1 = SRAG por influenza;
- 2 = SRAG por outro vírus respiratório;
- 3 = SRAG por outro agente etiológico;
- 4 = SRAG não especificado;
- 5 = SRAG por COVID-19.
Observe a Figura 19 com o print de parte do dicionário de dados:
Figura 19: Dicionário de dados do Sivep Gripe da variável CLASSI_FIN.

Agora precisaremos criar um fator, utilizando a factor()
para recodificar, ou categorizar a variável CLASSI_FIN do
Sivep Gripe. Para isso, precisaremos:
- Definir um novo objeto para salvar as modificações escrevendo o
comando:
dados_sivep$CS_CLASSI_FIN_N. Onde criaremos a nova variávelCS_CLASSI_FIN_N, selecionada utilizando cifrão ($), do banco de dados {dados_sivep}.
- Lembre-se de utilizar o sinal de atribuição
<-, após a expressãodados_sivep$CS_CLASSI_FIN_N, pois ele garantirá que todos os comandos solicitados após sua escrita estarão salvos, de forma automática, na nova váriavelCS_CLASSI_FIN_N.
- E aplicar os três argumentos principais da função
factor():
x: indicando os dados que serão categorizados;levels: indicando os valores que serão utilizados como categorias (hierarquia);labels: indicando os nomes (rótulos) que vão identificar as categorias.
Observe o script abaixo e replique-o em seu
RStudio.
Atenção
O sinal de cifrão $ indica que estamos realizando uma
operação de selecionar uma variável no banco de dados escolhido.
Fique atento à escrita do seu script no
RStudio, pois qualquer erro ortográfico ou de pontuação
pode levar a dificuldades de rodar o seu código, e o R te
apresentará um aviso (warning)!
Pronto, vamos praticar! Imagine que você necessita analisar a
classificação final dos casos de SRAG do Estado de Rosas. Para isto
necessitaremos transformar todos os dados contidos na variável
CLASSI_FIN, do banco de dados {dados_sivep}, e
para isso utilizaremos o dicionário do banco de dados disponível no menu
lateral “Arquivos”, do módulo.
Observe na Figura 18 abaixo como está estruturada a variável
CLASSI_FIN do banco de dados {dados_sivep}
:
# Recodificando a coluna CLASSI_FIN utilizando a função factor
# e salvando as modificacoes na nova coluna CS_CLASSI_FIN_N
dados_sivep$CS_CLASSI_FIN_N <- factor(
x = dados_sivep$CLASSI_FIN,
levels = c("1", "2", "3", "4", "5"),
labels = c(
"SRAG por influenza",
"SRAG por outro vírus respiratório",
"SRAG por outro agente etiológico",
"SRAG não especificado",
"SRAG por COVID-19"
)
)Observe que executamos os seguintes argumentos na função
factor ():
x = dados_sivep$CLASSI_FINindicando o banco de dados {dados_sivep} e selecionando com o símbolo$(cifrão) a coluna (CLASSI_FIN) que será recodificada.levels = c("1", "2", "3", "4", "5")indicando quais são os dados que precisam ser transformados.labels = c("SRAG por influenza","SRAG por outro vírus respiratório","SRAG por outro agente etiológico","SRAG não especificado","SRAG por COVID-19")indicando quais são os valores que substituiram os valores citados nolevels(item 2).
Observe também que no output visualizamos as categorias recodificadas corretamente.
Quando transformamos uma variável em factor, estamos
guardando no R cada uma das suas categorias
(“1”,“2”,“3”,“4”,“5”) e associando uma identificação para cada uma
delas, assim:
- Onde está codificado como “1”, receberá o rótulo “SRAG por influenza”;
- Onde está codificado como “2”, receberá o rótulo “SRAG por outro vírus - respiratório”;
- Onde está codificado como “3”, receberá o rótulo “SRAG por outro agente etiológico”;
- Onde está codificado como “4”, receberá o rótulo “SRAG não especificado”;
- Onde está codificado como “5”, receberá o rótulo “SRAG por COVID-19”.
O número de valores informados no argumento
levels deve ser o mesmo do número de valores informados no
argumento labels.
Perceba que utilizamos a função c() no argumento
levels e labels. A função c() tem
o objetivo de concatenar valores em um conjunto. Como estamos criando um
conjunto de textos para os argumentos, os colocamos entre aspas duplas,
separando-os por vírgulas. Essa é uma estrutura comum nas
funções que utilizaremos durante o nosso curso.
Na linguagem de programação R, para ordenar por ordem
hierárquica uma variável ordinal precisamos definir as categorias no
argumento levels da função factor e adicionar
o argumento ordered = TRUE. Veja no código abaixo como
ficaria a recategorização da escolaridade, e replique-o em seu
RStudio:
dados_sivep$CS_ESCOL_N <- factor(
x = dados_sivep$CS_ESCOL_N,
levels = c("0", "1", "2", "3", "4", "5", "9"),
labels = c(
"Sem escolaridade",
"Fundamental 1º ciclo",
"Fundamental 2º ciclo",
"Médio",
"Superior",
"Não se aplica",
"Ignorado"
),
ordered = TRUE
)

